
Wetenschap
Onderzoekers gebruiken modelleringstools voor machinaal leren om de technologie voor het bewerken van zinkvingernucleases te verbeteren
Genome editing maakt steeds meer indruk in het biomedisch onderzoek en de geneeskunde. Door tools voor het modelleren van biomoleculen in te zetten, versnelt een Japans onderzoeksteam het tempo en verlaagt het de kosten van zinkvingernuclease (ZFN), een primair hulpmiddel voor het bewerken van genen.
In een studie gepubliceerd in Advanced Science , laten onderzoekers van de Universiteit van Hiroshima en het Japanse Nationale Instituut voor Geavanceerde Industriële Wetenschappen en Technologie zien hoe machine learning-gestuurde modulaire assemblagesystemen het bewerken van genen kunnen verbeteren.
"Genoombewerking is een veelbelovend hulpmiddel voor de behandeling van genetische aandoeningen op een aantal verschillende gebieden", zegt Shota Katayama, universitair hoofddocent aan het Genome Editing Innovation Center aan de Universiteit van Hiroshima. "Door de efficiëntie van genbewerkingstechnologieën te verbeteren, kunnen we een grotere precisie bereiken bij het wijzigen van de genetische informatie in levende cellen."
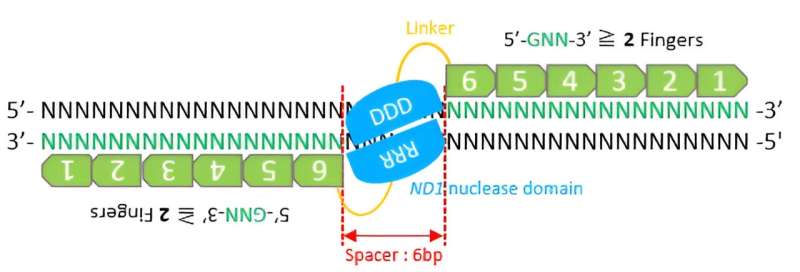
Naast CRISPR/Cas9 en TALEN is zinkvingernuclease een belangrijk hulpmiddel op het gebied van genoombewerking. Deze chimere eiwitten zijn ontworpen om bepaalde bindingen binnen de polynucleotideketen van een DNA-molecuul te verbreken en bestaan uit twee aan elkaar gefuseerde domeinen:DNA-bindende en DNA-splitsingsdomeinen. Het zinkvingereiwitbindende domein (ZF) herkent de beoogde DNA-sequentie binnen het volledige genoom, terwijl bij het splitsingsdomein een speciaal DNA-knippend enzym betrokken is, genaamd ND1-endonucleasen.
ZFN's bieden een aantal voordelen ten opzichte van CRISPR/Cas9 en TALEN:ten eerste zijn, in tegenstelling tot CRISPR-Cas9, de patenten voor ZFN's al verlopen, waardoor hoge patentrechten voor industriële toepassingen uitgesloten zijn. Ten tweede zijn ZFN's kleiner, waardoor voor ZFN coderend DNA gemakkelijk kan worden verpakt in een virale vector met beperkte laadruimte voor in vivo en klinische toepassingen.
Om DNA te knippen, moeten twee ZFN's aan elkaar worden gebonden. Daarom moeten ze in paren worden ontworpen om op elke nieuwe locatie functioneel te zijn. Het construeren van functionele ZFN's en het verbeteren van de efficiëntie van hun genoombewerking is echter een uitdaging gebleken.
"We hebben enorme vooruitgang geboekt in de methoden voor het afleiden van zinkvingersets voor nieuwe genomische doelwitten, maar er is nog steeds ruimte om onze benadering van ontwerp en selectie te verbeteren," zei Katayama.
Op selectie gebaseerde methoden kunnen worden gebruikt om geassembleerde ZF-eiwitten te construeren, maar deze methoden zijn arbeidsintensief en tijdrovend. Een alternatieve benadering voor het construeren van geassembleerde ZF-eiwitten is de assemblage van ZF-modules met behulp van standaard moleculair biologische technieken. Deze methode biedt onderzoekers een veel eenvoudigere methode om geassembleerde ZF-eiwitten te construeren.
Modulair geassembleerde ZFN's hebben echter een klein aantal functionele ZFN-paren met een uitvalpercentage van 94% voor de geteste ZFN-paren.
In hun onderzoek wilden de onderzoekers van de Universiteit van Hiroshima en het Japanse Nationale Instituut voor Geavanceerde Industriële Wetenschappen en Technologie een efficiëntere, gemakkelijker te construeren zinkvingernuclease voor genbewerking creëren met behulp van openbaar beschikbare bronnen in een modulair assemblagesysteem.
Een belangrijke overweging bij het ontwerp van ZFN's is het aantal zinkvingers dat nodig is voor efficiënte en specifieke splitsing. Het team veronderstelde dat de modulaire assemblage van de ZF-modules nuttig zou zijn voor het construeren van ZFN's met vijf of zes vingers.
In hun publicatie presenteerde het onderzoeksteam een methode om de efficiëntie van de constructie van functionele ZFN's en de verbetering van de efficiëntie van hun genoombewerking te verhogen met behulp van drie tools voor het modelleren van biomoleculen:AlphaFold, Coot en Rosetta.
Van de tien geteste ZFN’s verkregen de onderzoekers twee functionele paren. Bovendien verhoogde de engineering van ZFN's met behulp van AlphaFold, Coot en Rosetta de efficiëntie van genoombewerking met 5%, wat de effectiviteit aantoont van de engineering van ZFN's op basis van structurele modellering.