
Wetenschap
Bio-informatici schrappen een onnodige stap in eiwitstabiliteitsanalyse
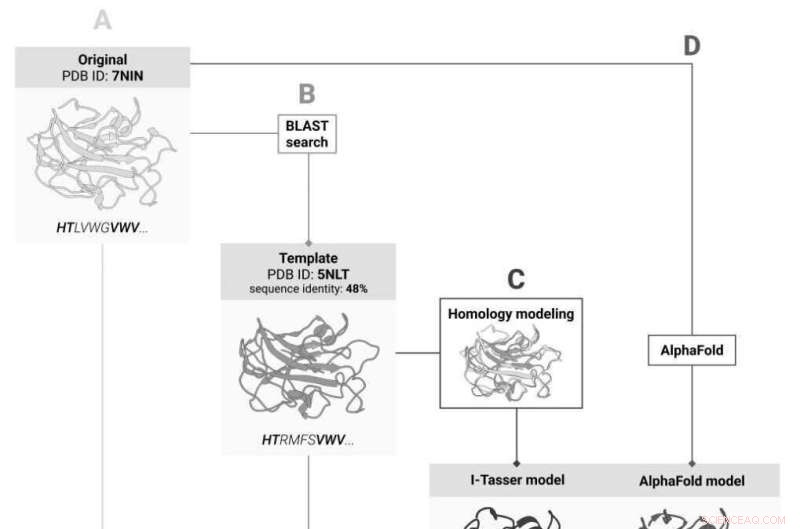
Vier manieren om veranderingen in eiwitstabiliteit na mutatie te voorspellen:(A) door de structuur van het oorspronkelijke eiwit; (B) door de structuur van zijn homoloog; (C) door de structuur van het oorspronkelijke eiwit voorspeld op basis van de structuur van de holomlog, en (D) door de structuur voorspeld door kunstmatige intelligentie op basis van de aminozuursequentie. Krediet:Skolkovo Instituut voor Wetenschap en Technologie
Onderzoekers van het Skoltech Center for Molecular and Cellular Biology vergeleken verschillende methoden voor het voorspellen van de eiwitstructuur in termen van de evaluatie van de stabiliteit van mutante eiwitten en verkregen hetzelfde resultaat voor de AI-voorspelde structuren en experimentele driedimensionale (3D) van eiwitten met vergelijkbare aminozuursequenties. De poging om de structuur van het beoogde eiwit te voorspellen op basis van de bekende structuur van zijn "relatief" maakte de voorspelling echter alleen maar minder nauwkeurig. De bevindingen van het team zullen voorlopige berekeningen vergemakkelijken bij de evaluatie van stabiliteitsveranderingen veroorzaakt door mutatie. Het onderzoek is gepubliceerd in Bioinformatics .
Bij biologische experimenten zijn vaak mutante eiwitten betrokken, die nodig zijn voor de studie van de eiwitstructuur en -functies of celprocessen, evenals eiwitengineering. Van mutaties is bekend dat ze de structuur en stabiliteit van een eiwit beïnvloeden. Omdat experimenten te kostbaar en tijdrovend zijn, creëren wetenschappers een tijdelijke oplossing in de vorm van computationele methoden om de impact van mutaties op de stabiliteit te evalueren. Hun toepassingen vereisen echter kennis van de 3D-structuur van een eiwit.
Een experimentele 3D-structuur is niet voor alle eiwitten beschikbaar en zal waarschijnlijk ontbreken voor degene waarop het team zich richt. Als dit het geval is, kunnen 3D-modellen van de homologen van het eiwit, d.w.z. de 'naaste verwanten', de reddingslijn vormen, omdat de mate van overeenkomst in aminozuursequenties die zorgt voor een goede match tussen de 3D-structuren van de eiwitten bekend is. De oplossing zou zijn om eerst de structuur van het eiwit te voorspellen op basis van de bekende structuur van zijn homoloog en vervolgens de impact van mutaties voor het voorspelde model te berekenen.
Dankzij de doorbraak van vorig jaar in de voorspelling van de eiwitstructuur, hebben de wetenschappers nu een alternatief:in plaats van de 3D-structuur te voorspellen op basis van homologen, kunnen ze de op AI gebaseerde AlphaFold-tool gebruiken die de eiwitstructuur voorspelt op basis van de aminozuursequentie en al heeft behandeld met de overgrote meerderheid van de tot nu toe bekende eiwitten.
In hun recente studie besloten de Skoltech-onderzoekers om uit te zoeken welke van deze benaderingen het beste werkt voor het voorspellen van stabiliteitsveranderingen bij mutatie. Hoe nauwkeurig AlphaFold ook is, het vinden van de eiwitstructuur door middel van experimenten blijft nog steeds de 'gouden standaard'. Bij het vergelijken van de twee benaderingen gebruikte het team zeven stabiliteitsevaluatiemethoden en vergeleek hun resultaten met die van AlphaFold en I-Tasser, het beste op homologen gebaseerde structuurvoorspellingssysteem. Ook hebben de onderzoekers gecontroleerd of ze de op homologen gebaseerde structuurvoorspelling kunnen overslaan en stabiliteit kunnen berekenen voor de bekende structuur van het homologe eiwit.
"We besloten om uit te zoeken hoe ver we zouden afwijken van een nauwkeurige voorspelling als we de 'naburige' eiwitstructuur zouden gebruiken in plaats van de echte. Het bleek dat de op homologie gebaseerde voorspellingsstap de zaken alleen maar erger maakt door een minder nauwkeurig resultaat op te leveren. We hebben laten zien dat het vrijwel geen verschil maakt of je de experimentele structuur van de homoloog of de voorspelling van AlphaFold gebruikt.In zekere zin ging het om validatie:als je met een nieuwe methode wordt geconfronteerd, moet je in de eerste plaats controleren of het werkt voor je taak Dat is precies wat we deden," eerste auteur van de studie, Skoltech Ph.D. student Marina Pak, opmerkingen.
"Met al deze ophef over AlphaFold geloven sommige wetenschappers en niet-professionals dat het alle eiwitonderzoeksproblemen in de computationele biologie heeft opgelost, maar dat is niet het geval. De voorspelling van door mutaties veroorzaakte stabiliteitsveranderingen vertoont bijvoorbeeld nog steeds een vrij lage betrouwbaarheid, zelfs hoewel de verandering in stabiliteit een van de belangrijkste drijfveren is van eiwitfunctionaliteit. Een hulpmiddel dat ondubbelzinnig de impact van mutatie op stabiliteit zou kunnen bepalen, zou zowel helpen bij het plannen van het experiment als bij het interpreteren van de resultaten. Stel dat voor een eiwit dat niet optimaal is in termen van stabiliteit, we willen mutaties vinden die het stabiel maken onder de gewenste omstandigheden, bijvoorbeeld om ervoor te zorgen dat het actief blijft bij hoge temperatuur. Zodra we dit alleen kunnen doen door middel van berekeningen, zal de benadering van herontwerp en optimalisatie van eiwitten drastisch veranderen." hoofdauteur van de studie, concludeert Skoltech-assistent-professor Dmitry Ivankov.
Hoewel het voorspellen van stabiliteitsveranderingen eenvoudiger lijkt dan het voorspellen van de 3D-structuur, blijft het zelfs voor AI een onhandelbare uitdaging. Schaarse trainingsgegevens zijn slechts een van de problemen:AlphaFold had bijna 200.000 eiwitstructuren om te trainen, maar experimentele gegevens over stabiliteitsveranderingen bedragen duizenden sets terwijl slechts enkele tientallen unieke eiwitten worden gebruikt. De auteurs hopen dat als er meer gegevens beschikbaar komen en onderzoekers meer interesse in de taak tonen, er snel een doorbraak zal komen. + Verder verkennen
Natuurkundigen gebruiken AI om de meest complexe eiwitknopen tot nu toe te vinden
 Hoe de Glow in the Dark te maken Chemical
Hoe de Glow in the Dark te maken Chemical Wetenschappers verkrijgen met succes een synthetische groeifactor die compatibel is met het inheemse eiwit
Wetenschappers verkrijgen met succes een synthetische groeifactor die compatibel is met het inheemse eiwit Materiaal met hoge brekingsindex behoudt een hoge doorlaatbaarheid na uitgloeien bij 850 ° C
Materiaal met hoge brekingsindex behoudt een hoge doorlaatbaarheid na uitgloeien bij 850 ° C Nieuw proces voor het produceren van hoogspanningskathodes voor lithium-ionbatterijen
Nieuw proces voor het produceren van hoogspanningskathodes voor lithium-ionbatterijen Onderzoekers onderzoeken ontstekingsremmende activiteit van curcumine
Onderzoekers onderzoeken ontstekingsremmende activiteit van curcumine
 Europa plan voor drijvende gasterminals wekt angst voor klimaat
Europa plan voor drijvende gasterminals wekt angst voor klimaat Akoestische golven geven een boost aan realtime seismische beeldvorming
Akoestische golven geven een boost aan realtime seismische beeldvorming Warmte, vochtigheid houdt het oosten van de VS vast terwijl het weekend ploetert
Warmte, vochtigheid houdt het oosten van de VS vast terwijl het weekend ploetert Studie:klimaatverandering verandert de manier waarop warmte zich over de hele wereld beweegt
Studie:klimaatverandering verandert de manier waarop warmte zich over de hele wereld beweegt Onderzoek naar de impact van klimaatverandering op snowpack
Onderzoek naar de impact van klimaatverandering op snowpack
Hoofdlijnen
- Hoe nauwkeurig is ons mentale beeld van onszelf?
- Makeup Science Projects
- Op AI gebaseerde screeningmethode zou de snelheid van nieuwe geneesmiddelenontdekking kunnen verhogen
- Gestratificeerd epitheelweefsel: definitie, structuur, typen
- Hoe de anatomie van een rundvleeshart en een menselijk hart te vergelijken
- 10 planten verloren in de geschiedenis
- Dieren in overvloed, ruimte voor een premie in de oudste dierentuin van Afrika
- Wetenschappers onderzoeken hoe olie de kleinste organismen in Antarctische wateren beïnvloedt
- Hoe goede bacteriën je genen beheersen
- Het ontdekken van langlevende eiwitten van sirtuin in vroege takken van het dierenleven
- Wetenschappers ontwerpen bacteriën om sonarsignalen te reflecteren voor ultrasone beeldvorming
- Negen verdwaalde olifanten geëlektrocuteerd in Botswana

- Op de maat blijven is gekoppeld aan reproductief succes bij mannelijke rotsklipdassen

- DNA kraakt een eeuwenoud mysterie over de enige uitgestorven zoetwatervis van Nieuw-Zeeland

 ESO's VLT werkt voor het eerst als 16-metertelescoop
ESO's VLT werkt voor het eerst als 16-metertelescoop Energetische kosten van de entatische toestand van cytochroom c gekwantificeerd
Energetische kosten van de entatische toestand van cytochroom c gekwantificeerd Magnetische velden sturen astrofysische straalvormen aan
Magnetische velden sturen astrofysische straalvormen aan DNA-onderzoek toont neolithische Europeanen gekruist met Anatolische migranten
DNA-onderzoek toont neolithische Europeanen gekruist met Anatolische migranten Laura wint aan kracht, kan een onoverleefbare stormvloed veroorzaken
Laura wint aan kracht, kan een onoverleefbare stormvloed veroorzaken Basisonderzoek microbiologie onthult onverwacht praktische bevindingen voor telers
Basisonderzoek microbiologie onthult onverwacht praktische bevindingen voor telers 3 delen Alternative Nobel, Amerikaanse advocaat krijgt ereprijs
3 delen Alternative Nobel, Amerikaanse advocaat krijgt ereprijs Filippijnen plannen gedwongen evacuaties rond uitbarstende vulkaan
Filippijnen plannen gedwongen evacuaties rond uitbarstende vulkaan
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

