
Wetenschap
Genetisch manipulatiemechanisme gevisualiseerd
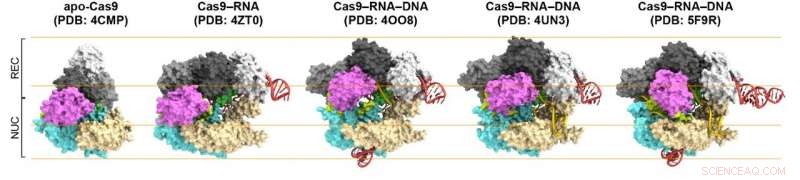
Figuur 1. Structuren van Cas9. Van links naar rechts:Cas9 alleen (apo-Cas9), Cas9 gebonden aan RNA (Cas9-RNA), Cas9-RNA gebonden aan zijn enkelstrengs DNA-doelwit (Cas9-RNA-DNA), Cas9-RNA gebonden aan een gedeeltelijke DNA-duplex (Cas9-RNA-DNA) en Cas9-RNA gebonden aan zijn dubbelstrengs DNA-doelwit (Cas9-RNA-DNA). Krediet:Kanazawa University
Onderzoekers van Kanazawa University en de University of Tokyo rapporteren in Natuurcommunicatie de visualisatie van de dynamiek van 'moleculaire schaar' - het belangrijkste mechanisme van de genetische manipulatietechniek CRISPR-Cas9.
Een van de technieken die worden gebruikt in genetische manipulatie - het proces van het kunstmatig wijzigen van het genoom van een levend organisme - omvat het zogenaamde CRISPR-Cas9-nucleasesysteem. Met behulp van dit systeem, het DNA van een cel kan op een gewenste plaats worden geknipt, waar genen kunnen worden verwijderd of toegevoegd. De selectie van de plaats die moet worden gesneden, wordt gedaan door een 'gids-RNA'-molecuul dat is gebonden aan het Cas9-eiwit. Nutsvoorzieningen, een team van onderzoekers onder leiding van Mikihiro Shibata van Kanazawa University en Osamu Nureki van de University of Tokyo heeft de dynamiek van het CRISPR-Cas9-complex gevisualiseerd, in het bijzonder hoe het DNA knipt, waardevolle inzichten in het CRISPR-Cas9-gemedieerde DNA-splitsingsmechanisme.
Voor hun visualisatiestudies, de wetenschappers gebruikten snelle atoomkrachtmicroscopie (HS-AFM), een methode voor het in beeld brengen van oppervlakken. Een oppervlak wordt onderzocht door er een kleine cantilever overheen te bewegen; de door de sonde ondervonden kracht kan worden omgezet in een hoogtemaat. Een scan van het hele oppervlak resulteert dan in een hoogtekaart van het monster. De supersnelle experimentele opstelling van Shibata en collega's maakte extreem snelle, herhaalde scans - die kunnen worden omgezet in films - van de biomoleculen die deelnemen aan de moleculaire schaaractie.
Eerst, de wetenschappers vergeleken Cas9 zonder en met aangehecht RNA (Cas9-RNA). Ze ontdekten dat de eerste in staat was om flexibel verschillende conformaties aan te nemen, terwijl de laatste een vaste, tweelobbige structuur, het benadrukken van het conformationele stabilisatievermogen van het gids-RNA. Vervolgens, Shibata en collega's keken hoe het gestabiliseerde Cas9-RNA-complex zich op DNA richt. Ze bevestigden dat het bindt aan een vooraf geselecteerde protospacer aangrenzende motief (PAM) plaats in het DNA. Een PAM is een korte nucleotidesequentie die zich naast de doellocatie van het DNA bevindt, die complementair is aan het gids-RNA.
De hogesnelheidsfilms van het onderzoeksteam onthulden verder dat targeting ('DNA-ondervraging') wordt bereikt door 3D-diffusie van het Cas9-RNA-complex. Eindelijk, de onderzoekers slaagden erin om de dynamiek van het splitsingsproces zelf te visualiseren:ze observeerden hoe het gebied van de 'moleculaire schaar' conformationele fluctuaties ondergaat nadat Cas9-RNA het dubbelstrengs DNA lokaal afwikkelt (film 1 [URL]).
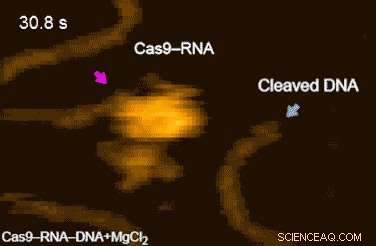
Figuur 2. HS-AFM-films van DNA-splitsing door Cas9-RNA. Fluctuaties van het nucleasedomein worden aangegeven door magenta pijlen. De splitsingsproducten die vrijkomen uit Cas9-RNA worden aangegeven door blauwe pijlen. Krediet:Kanazawa University
Het werk van Shibata bevordert ons begrip van het CRISPR-Cas9-mechanisme voor genoombewerking. In de woorden van de onderzoekers:"... deze studie biedt ongekende details over de functionele dynamiek van CRISPR-Cas9, en benadrukt het potentieel van HS-AFM om de actiemechanismen van RNA-geleide effectornucleasen van verschillende CRISPR-Cas-systemen op te helderen."
CRISPR-Cas9
CRISPR, afkorting voor "geclusterde regelmatig tussenruimte korte palindroom herhalingen", verwijst naar een reeks bacteriële DNA-sequenties die fragmenten bevatten van het DNA van virussen die de bacteriën eerder hebben aangevallen. Deze fragmenten worden door de bacteriën gebruikt om verdere aanvallen van dezelfde virussen te voorkomen. "Cas" verwijst naar CRISPR-geassocieerde genen; "Cas9" is een CRISPR-geassocieerd eiwit met twee nucleasedomeinen (een nuclease is een enzym dat in staat is nucleïnezuren te splitsen, organische moleculen aanwezig in DNA en RNA).
In recente jaren, er is een genetische manipulatietechniek ontwikkeld waarbij een CRISPR-Cas9-complex als 'moleculaire schaar' fungeert; het Cas9-nuclease bindt aan een gids-RNA-molecuul dat informatie bevat over de DNA-plaats waarop het zich moet richten. Met behulp van high-speed atomic force microscopie, Mikihiro Shibata van Kanazawa University en collega's hebben nu de dynamiek van het CRISPR-Cas9-complex tot in detail bestudeerd.
Atoomkrachtmicroscopie
Atoomkrachtmicroscopie (AFM) is een beeldvormende techniek waarbij het beeld wordt gevormd door een oppervlak te scannen met een zeer kleine punt. Horizontal scanning motion of the tip is controlled via piezoelectric elements, while vertical motion is converted into a height profile, resulting in a height distribution of the sample's surface. As the technique does not involve lenses, its resolution is not restricted by the so-called diffraction limit. In a high-speed setup, AFM can be used to produce movies of a sample's evolution in real time. High-speed AFM has been used successfully to study protein dynamics, for example myosin V walking on an actin filament, the photo-induced conformational change of bacteriorhodopsin, and the degradation of cellulose. Shibata and colleagues have now applied the high-speed AFM technique for visualizing the dynamics of DNA cleavage by CRISPR-Cas9.
 Moleculaire trillingen verminderen de maximaal haalbare fotospanning in organische zonnecellen
Moleculaire trillingen verminderen de maximaal haalbare fotospanning in organische zonnecellen Hoe komt zure regen de watercyclus binnen?
Hoe komt zure regen de watercyclus binnen?  Planten modelleren efficiëntere thermische koelingsmethode
Planten modelleren efficiëntere thermische koelingsmethode Wetenschappers onthullen verschillende substraatbindingsmodus in o-succinylbenzoyl-CoA-synthetase
Wetenschappers onthullen verschillende substraatbindingsmodus in o-succinylbenzoyl-CoA-synthetase Chemici ontwikkelen milieuvriendelijke nanocomposieten uit fruit- en bessenschillen
Chemici ontwikkelen milieuvriendelijke nanocomposieten uit fruit- en bessenschillen
 Zeer weinig publieke steun voor versoepeling van regels en voorschriften rond fracking
Zeer weinig publieke steun voor versoepeling van regels en voorschriften rond fracking Milieuvervuiling in China neemt af
Milieuvervuiling in China neemt af Kleine stroompjes en wetlands zijn belangrijke onderdelen van riviernetwerken - dit is waarom ze bescherming nodig hebben
Kleine stroompjes en wetlands zijn belangrijke onderdelen van riviernetwerken - dit is waarom ze bescherming nodig hebben Welke economische systemen reageren het best op rampen?
Welke economische systemen reageren het best op rampen?  Planten nemen minder koolstof op in een opwarmende wereld
Planten nemen minder koolstof op in een opwarmende wereld
Hoofdlijnen
- Radiometrische datering: definitie, hoe werkt het, gebruik & voorbeelden
- Volledig diagram van het menselijk lichaam
- Zwaarste beenvissen ter wereld geïdentificeerd en correct benoemd
- Vier klassen van macromoleculen belangrijk voor levende dingen
- Voorbeelden van stoffen die gefaciliteerde diffusie gebruiken
- Wat zijn de chemische zintuigen?
- Wat is het verschil tussen Ribosoom en Ribosomaal DNA?
- Hoe bacteriën met zout te doden
- Waarom zijn er 61 Anticodonen?
- Leven in drijfhout in zee:het geval van drijfhoutspecialist talitrids

- Chromatid: wat is het?

- Genetische studie onthult seksgeheimen van schimmels werpt licht op candidiasis
- Laatste gemeenschappelijke aap-menselijke voorouder was waarschijnlijk zo groot als een gibbon

- Hoe wordt water gevormd tijdens cellulaire ademhaling?
 Cassini bereidt zich voor op ringgrazende banen
Cassini bereidt zich voor op ringgrazende banen Hoe snel loopt een tijger?
Hoe snel loopt een tijger?  Duitsland stemt ermee in om drones van Israëlische makelij te leasen:fabrikant
Duitsland stemt ermee in om drones van Israëlische makelij te leasen:fabrikant Zullen aardbevingen ooit voorspelbaar zijn?
Zullen aardbevingen ooit voorspelbaar zijn?  De nieuwe Apple Watch is een Legit Medical Device - maar er is een vangst
De nieuwe Apple Watch is een Legit Medical Device - maar er is een vangst  Nieuwe theorie illustreert dat de ontwikkeling van het universum anders kan zijn dan we dachten
Nieuwe theorie illustreert dat de ontwikkeling van het universum anders kan zijn dan we dachten Gesprayde tactiele sensor op een 3D-oppervlak voor robothuid
Gesprayde tactiele sensor op een 3D-oppervlak voor robothuid Sterkere wind zorgt voor smeltend ijs op West-Antarctisch gebied
Sterkere wind zorgt voor smeltend ijs op West-Antarctisch gebied
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

