
Wetenschap
De toekomst van gegevensopslag is dubbel spiraalvormig, blijkt uit onderzoek
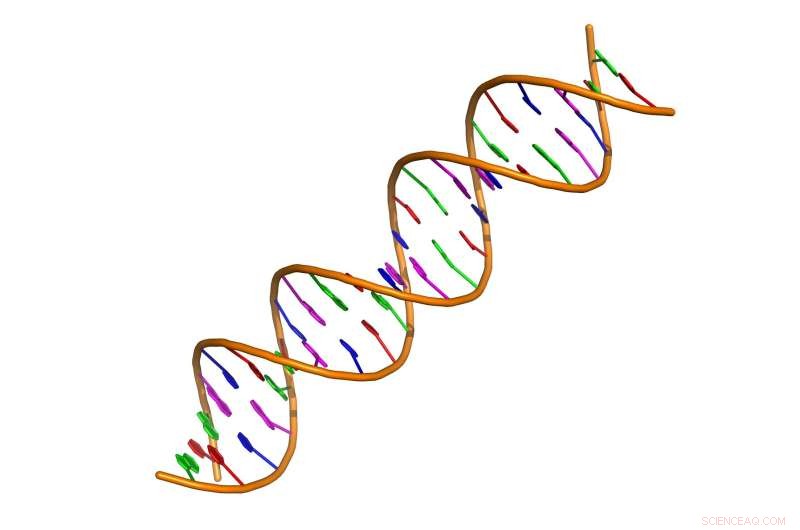
Een dubbelstrengs DNA-fragment. Krediet:Vcpmartin/Wikimedia/CC BY-SA 4.0
Stel je voor dat Bach's "Cello Suite nr. 1" op een DNA-streng wordt gespeeld.
Dit scenario is niet zo onmogelijk als het lijkt. DNA is te klein om een ritmische tokkel of glijdende boogpees te weerstaan en is een krachtpatser voor het opslaan van audiobestanden en allerlei andere media.
"DNA is het oorspronkelijke gegevensopslagsysteem van de natuur. We kunnen het gebruiken om alle soorten gegevens op te slaan:afbeeldingen, video, muziek - alles", zegt Kasra Tabatabaei, een onderzoeker aan het Beckman Institute for Advanced Science and Technology en een co-auteur van deze studie.
Door de moleculaire samenstelling van DNA uit te breiden en een nauwkeurige nieuwe sequentiemethode te ontwikkelen, kon een multi-institutioneel team de dubbele helix transformeren in een robuust, duurzaam gegevensopslagplatform.
De paper van het team verscheen in Nano Letters in februari 2022.
In het tijdperk van digitale informatie voelt iedereen die dapper genoeg is om door het dagelijkse nieuws te navigeren het wereldwijde archief met de dag zwaarder worden. Papieren dossiers worden steeds vaker gedigitaliseerd om ruimte te besparen en informatie te beschermen tegen natuurrampen.
Van wetenschappers tot influencers op sociale media, iedereen die informatie heeft om op te slaan, kan profiteren van een veilige, duurzame datalock-box - en de dubbele helix past precies.
"DNA is een van de beste opties, zo niet de beste optie, om vooral archiefgegevens op te slaan", zegt Chao Pan, een afgestudeerde student aan de University of Illinois Urbana-Champaign en een co-auteur van deze studie.
Zijn lange levensduur kan alleen worden geëvenaard door duurzaamheid, DNA is ontworpen om de zwaarste omstandigheden op aarde te doorstaan - soms tienduizenden jaren - en een levensvatbare gegevensbron te blijven. Wetenschappers kunnen gefossiliseerde strengen sequensen om genetische geschiedenissen te ontdekken en leven in te blazen in lang verloren gewaande landschappen.
Ondanks zijn kleine gestalte lijkt DNA een beetje op de beruchte politiebox van Dr. Who:groter van binnen dan het lijkt.
"Elke dag worden er meerdere petabytes aan gegevens op internet gegenereerd. Slechts één gram DNA zou voldoende zijn om die gegevens op te slaan. Zo dicht is DNA als opslagmedium", zegt Tabatabaei, die ook een vijfdejaars Ph. D. leerling.
Een ander belangrijk aspect van DNA is zijn natuurlijke overvloed en bijna oneindige hernieuwbaarheid, een eigenschap die niet wordt gedeeld door het meest geavanceerde gegevensopslagsysteem dat momenteel op de markt is:siliciummicrochips, die vaak slechts tientallen jaren circuleren voordat ze zonder pardon begraven worden op een hoop gestorte e -afval.
"In een tijd waarin we worden geconfronteerd met ongekende klimaatuitdagingen, kan het belang van duurzame opslagtechnologieën niet worden overschat. Er komen nieuwe, groene technologieën voor DNA-registratie op die moleculaire opslag in de toekomst nog belangrijker zullen maken", zegt Olgica Milenkovic, de Franklin W. Woeltge Hoogleraar Electrical and Computer Engineering en een co-PI op de studie.
Met het oog op de toekomst van gegevensopslag, onderzocht het interdisciplinaire team de millennia-oude MO van DNA. Vervolgens voegden de onderzoekers hun eigen 21e-eeuwse draai toe.
In de natuur bevat elke DNA-streng vier chemicaliën - adenine, guanine, cytosine en thymine - vaak aangeduid met de initialen A, G, C en T. Ze rangschikken en herschikken zichzelf langs de dubbele helix in combinaties die wetenschappers kunnen decoderen , of reeks, om betekenis te geven.
The researchers expanded DNA's already broad capacity for information storage by adding seven synthetic nucleobases to the existing four-letter lineup.
"Imagine the English alphabet. If you only had four letters to use, you could only create so many words. If you had the full alphabet, you could produce limitless word combinations. That's the same with DNA. Instead of converting zeroes and ones to A, G, C, and T, we can convert zeroes and ones to A, G, C, T, and the seven new letters in the storage alphabet," Tabatabaei said.
Because this team is the first to use chemically modified nucleotides for information storage in DNA, members innovated around a unique challenge:Not all current technology is capable of interpreting chemically modified DNA strands. To solve this problem, they combined machine learning and artificial intelligence to develop a first-of-its-kind DNA sequence readout processing method.
Their solution can discern modified chemicals from natural ones, and differentiate each of the seven new molecules from one another.
"We tried 77 different combinations of the 11 nucleotides, and our method was able to differentiate each of them perfectly," Pan said. "The deep learning framework as part of our method to identify different nucleotides is universal, which enables the generalizability of our approach to many other applications."
This letter-perfect translation comes courtesy of nanopores:proteins with an opening in the middle through which a DNA strand can easily pass. Remarkably, the team found that nanopores can detect and distinguish each individual monomer unit along the DNA strand—whether the units have natural or chemical origins.
"This work provides an exciting proof-of-principle demonstration of extending macromolecular data storage to non-natural chemistries, which hold the potential to drastically increase storage density in non-traditional storage media," said Charles Schroeder, the James Economy Professor of Materials Science and Engineering and a co-PI on this study.
DNA literally made history by storing genetic information. By the looks of this study, the future of data storage is just as double-helical. + Verder verkennen
Using DNA-like punch cards to store data
 Nieuwe wet zou de aanvoer van helium in de VS kunnen versterken
Nieuwe wet zou de aanvoer van helium in de VS kunnen versterken Neutronen maken structurele veranderingen in moleculaire borstels zichtbaar
Neutronen maken structurele veranderingen in moleculaire borstels zichtbaar Zware metalen uit bier en wijn houden
Zware metalen uit bier en wijn houden Team ontwikkelt machine met als doel een einde te maken aan textielverspilling
Team ontwikkelt machine met als doel een einde te maken aan textielverspilling Chemici ontwikkelen nieuwe technologie om te voorkomen dat lithium-ionbatterijen vlam vatten
Chemici ontwikkelen nieuwe technologie om te voorkomen dat lithium-ionbatterijen vlam vatten
 Deze klimaatactivisten willen dat je de hoop opgeeft
Deze klimaatactivisten willen dat je de hoop opgeeft Planetaire grenzen:interacties in het aardsysteem versterken de menselijke impact
Planetaire grenzen:interacties in het aardsysteem versterken de menselijke impact Getijdenmoeras of nephabitat? Milieuproject in Californië krijgt kritiek
Getijdenmoeras of nephabitat? Milieuproject in Californië krijgt kritiek Nieuwe metingen van breukzones kunnen ons helpen om subductie-aardbevingen te begrijpen
Nieuwe metingen van breukzones kunnen ons helpen om subductie-aardbevingen te begrijpen Laat roet van bosbranden een stempel achter op de oceanen van de wereld?
Laat roet van bosbranden een stempel achter op de oceanen van de wereld?
Hoofdlijnen
- Eerste celkaart van 20, 000 cellen in zoogdierembryo
- Nieuwe studie verifieert meer manieren om te overleven voor bedreigde Chinook-zalm in de winter
- Een nieuw type robotmicroscoop
- Hoe celbrochures te maken
- Een betere manier om pesticiden van appels te wassen
- De evolutionaire voordelen van botgewicht onderzoeken
- Vergroening van citrusvruchten behandelen met koper:effecten op bomen, bodems
- Eencellige tools geven inzicht in actieve antibioticaresistentie in bodems
- Hoe evolueren bacteriën in de darm in de loop van een jaar?
- Nieuwe studie is een stap in de richting van het creëren van vliegtuigen die met hypersonische snelheid reizen

- Wetenschapper maakt zonnebrandcrème van Ivy

- Helderheid-geëgaliseerde kwantumstippen verbeteren biologische beeldvorming
- Lichtvoortplanting in zonnecellen zichtbaar gemaakt
- Efficiëntere omzetting van warmte in elektriciteit door te sleutelen aan nanostructuur
 Russische wetenschappers ontwikkelen een slim sorptiemiddel voor waterzuivering
Russische wetenschappers ontwikkelen een slim sorptiemiddel voor waterzuivering Pine Island Glaciers-ijsplaat scheurt uit elkaar, de belangrijkste Antarctische gletsjer versnellen
Pine Island Glaciers-ijsplaat scheurt uit elkaar, de belangrijkste Antarctische gletsjer versnellen Hersenen van de operatie - NASA-team ontwikkelt modulaire luchtvaartelektronicasystemen voor kleine missies
Hersenen van de operatie - NASA-team ontwikkelt modulaire luchtvaartelektronicasystemen voor kleine missies Onderzoek onthult de impact van de consumptie van wild vlees op de uitstoot van broeikasgassen
Onderzoek onthult de impact van de consumptie van wild vlees op de uitstoot van broeikasgassen BigH1 - de belangrijkste histon voor mannelijke vruchtbaarheid
BigH1 - de belangrijkste histon voor mannelijke vruchtbaarheid Gevaren & Gebruik van Radioactiviteit
Gevaren & Gebruik van Radioactiviteit Tardigrades:de laatste overlevenden op aarde
Tardigrades:de laatste overlevenden op aarde Nieuw portaal om de donkere sector van het heelal te onthullen
Nieuw portaal om de donkere sector van het heelal te onthullen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

