
Wetenschap
Team suggereert elektronische bewegingssensor op nanoschaal als DNA-sequencer
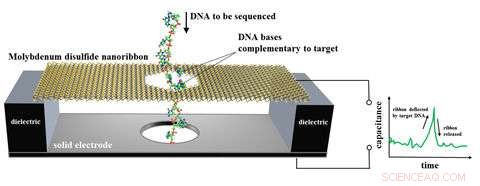
Het door NIST voorgestelde ontwerp voor een DNA-sequencer op basis van een elektronische bewegingssensor. Een lint van molybdeendisulfide op nanoschaal wordt over een metalen elektrode opgehangen en in water ondergedompeld. Enkelstrengs DNA, met een reeks basen (stukjes genetische code), wordt door een gat in het lint geregen, die alleen buigt wanneer een DNA-base paren met en vervolgens scheidt van een complementaire base die aan het gat is bevestigd. De membraanbeweging wordt gedetecteerd als een elektrisch signaal. Numerieke simulaties en theoretische schattingen tonen aan dat het membraan 79 tot 86 procent nauwkeurig zou zijn in het identificeren van DNA-basen in een enkele meting met snelheden tot ongeveer 70 miljoen basen per seconde. Krediet:NIST
Onderzoekers van het National Institute of Standards and Technology (NIST) en medewerkers hebben een ontwerp voorgesteld voor de eerste DNA-sequencer op basis van een elektronische nanosensor die kleine bewegingen zo klein als een enkel atoom kan detecteren.
Het voorgestelde apparaat - een type condensator, die elektrische lading opslaat - is een klein lint van molybdeendisulfide dat over een metalen elektrode is opgehangen en in water is ondergedompeld. Het lint is 15,5 nanometer (nm, miljardsten van een meter) lang en 4,5 nm breed. Enkelstrengs DNA, met een reeks basen (stukjes genetische code), wordt door een gat van 2,5 nm breed in het dunne lint geregen. Het lint buigt alleen wanneer een DNA-base paren met en vervolgens scheidt van een complementaire base die aan het gat is bevestigd. De membraanbeweging wordt gedetecteerd als een elektrisch signaal.
Zoals beschreven in een nieuw artikel, het NIST-team maakte numerieke simulaties en theoretische schattingen om aan te tonen dat het membraan 79 tot 86 procent nauwkeurig zou zijn in het identificeren van DNA-basen in een enkele meting met snelheden tot ongeveer 70 miljoen basen per seconde. Geïntegreerde schakelingen zouden elektrische signalen detecteren en meten en basen identificeren. De resultaten suggereren dat een dergelijk apparaat een snelle, nauwkeurige en kosteneffectieve DNA-sequencer, volgens het papier.
Conventionele sequentiëring, ontwikkeld in de jaren 70, gaat scheiden, kopiëren, het labelen en samenvoegen van stukjes DNA om de genetische informatie te lezen. Nieuwere methoden omvatten geautomatiseerde sequencing van veel DNA-fragmenten tegelijk - nog steeds kostbaar - en nieuwe concepten voor 'nanoporesequencing'. Bijvoorbeeld, dezelfde NIST-groep demonstreerde onlangs het idee om DNA te sequencen door het door een grafeen-nanoporie te leiden, en meten hoe de elektronische eigenschappen van grafeen reageren op spanning.
Het nieuwste NIST-voorstel is gebaseerd op een dunne film van molybdeendisulfide - een stabiel, gelaagd materiaal dat elektriciteit geleidt en vaak als smeermiddel wordt gebruikt. Onder andere voordelen, dit materiaal hecht niet aan DNA, wat een probleem kan zijn met grafeen. Het NIST-team suggereert dat de methode zelfs zou kunnen werken zonder een nanoporie - een eenvoudiger ontwerp - door DNA over de rand van het membraan te leiden.
"Deze benadering lost mogelijk het probleem op waarbij DNA aan grafeen blijft kleven als het onjuist wordt ingebracht, omdat deze benadering geen gebruik maakt van grafeen, punt uit, "NIST-theoreticus en hoofdauteur Alex Smolyanitsky zei. "Een ander groot verschil is dat in plaats van te vertrouwen op de eigenschappen van grafeen of een bepaald materiaal dat wordt gebruikt, we lezen bewegingen elektrisch op een gemakkelijkere manier door een condensator te vormen. Dit maakt elk elektrisch geleidend membraan geschikt voor de toepassing."
Nanomaterialen-expert Boris Yakobson van Rice University, een co-auteur op het papier, stelde het condensatoridee voor. Computational support werd geleverd door de Rijksuniversiteit Groningen in Nederland.
DNA heeft vier basen. Voor de simulaties cytosine (C), die van nature gepaard gaat met guanine (G), zit vast aan de binnenkant van de porie. Als een stukje DNA door de porie gaat, elke G in de streng hecht tijdelijk aan de ingebedde C, aan het nanolint trekken en de elektrode signaleren. De DNA-sequentie wordt bepaald door te meten hoe en wanneer elektrische blips in de loop van de tijd variëren. Om alle vier de basen te detecteren, vier nanolinten, elk met een andere basis bevestigd aan de porie, kan verticaal worden gestapeld om een geïntegreerde DNA-sensor te creëren.
Het molybdeendisulfidelint is flexibel genoeg om meetbaar te vervormen als reactie op de krachten die nodig zijn om een DNA-paar te verbreken, maar rigide genoeg om minder aan de gang te hebben, zinloze beweging dan grafeen, mogelijk ongewenste ruis in de sequentiëringssignalen verminderen. De doorbuiging van het lint is buitengewoon klein, in de orde van één angstrom, de grootte van een waterstofatoom. De trekkracht is in de orde van 50 piconewton, of biljoensten van een newton, genoeg om de delicate chemische bindingen tussen DNA-basen te verbreken.
Onderzoekers schatten hoe het apparaat zou presteren in een geïntegreerd circuit en ontdekten dat de piekstromen door de condensator meetbaar waren (50 tot 70 picoampère), zelfs voor de kleine nanoribbons die werden bestudeerd. In fysieke systemen zullen de stroompieken naar verwachting nog groter zijn. De grootte van het apparaat kan worden aangepast om het nog eenvoudiger te maken om sequencing-signalen te meten.
De NIST-auteurs hopen in de toekomst een fysieke versie van het apparaat te bouwen. Voor praktische toepassingen, the chip-sized DNA sequencing microfluidic technology might be combined with electronics into a single device small enough to be handheld.
 Het terugdringen van methaanemissies die waarschijnlijk minder kosten dan geraamd, doelen voor 2025 missen
Het terugdringen van methaanemissies die waarschijnlijk minder kosten dan geraamd, doelen voor 2025 missen Nieuw rapport:stand van de wetenschap over westerse bosbranden, bossen en klimaatverandering
Nieuw rapport:stand van de wetenschap over westerse bosbranden, bossen en klimaatverandering Snelle landveranderingen voorspeld voor Oost-Afrikaanse savannes
Snelle landveranderingen voorspeld voor Oost-Afrikaanse savannes De effecten van cyclonen op de omgeving
De effecten van cyclonen op de omgeving Huis keurt wetgeving goed om chemische verontreiniging met PFAS aan te pakken
Huis keurt wetgeving goed om chemische verontreiniging met PFAS aan te pakken
Hoofdlijnen
- Biologen kijken naar het verleden voor vroege genetische ontwikkeling van kleine spinnen- en insectenogen
- Sorry,
- Bronnen van fouten in gelelektroforese
- De meest voorkomende organische moleculen in cellen
- Studie biedt gedetailleerd inzicht in het vroege leven van grijze zeehondenpups op zee
- Trilaminaire structuur van het celmembraan
- Gemengde organisatie van darmbacteriën wordt onthuld door microbioombeeldvormingstechnologie
- Wat zijn de primaire functies van fosfolipiden?
- Lichaamssystemen en hun functies
- Onderzoekers ontwikkelen baanbrekend nieuw op grafeen gebaseerd MRI-contrastmiddel

- Grafeenbatterij waarvan is aangetoond dat deze een LED van stroom voorziet

- Nanokristallen niet klein genoeg om defecten te voorkomen

- Grote stap naar kleine draden

- Nieuwe nanotherapie biedt hoop bij de behandeling van geneesmiddelresistent niercelcarcinoom

 Frontlader specificaties
Frontlader specificaties  Studie identificeert meest veelbelovende grondstoffen voor op pyrolyse gebaseerde bioraffinage
Studie identificeert meest veelbelovende grondstoffen voor op pyrolyse gebaseerde bioraffinage Termietendarmmicroben halen schone energie uit steenkool
Termietendarmmicroben halen schone energie uit steenkool Risico's van nanomaterialen onder de loep
Risico's van nanomaterialen onder de loep Belangrijkste bosbranden in Portugal onder controle (update)
Belangrijkste bosbranden in Portugal onder controle (update) Wat is complementair in Math?
Wat is complementair in Math?  De fabrieken in Wuhan zijn gesloten, Honda meldt kwartaalwinstdaling
De fabrieken in Wuhan zijn gesloten, Honda meldt kwartaalwinstdaling Kan het verkleinen van een belangrijk onderdeel helpen om autonome auto's betaalbaar te maken?
Kan het verkleinen van een belangrijk onderdeel helpen om autonome auto's betaalbaar te maken?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

