
Wetenschap
draagbaar, snelle DNA-test kan ebola en andere pathogenen detecteren
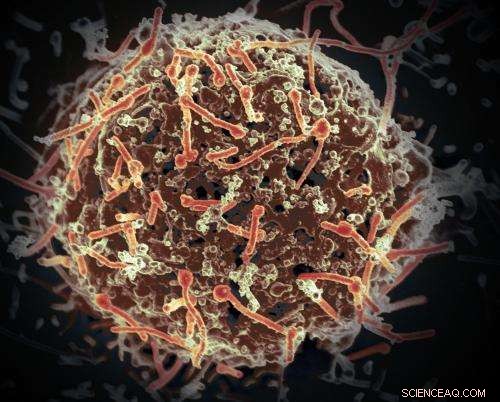
Het ebolavirus, geïsoleerd in november 2014 uit bloedmonsters van patiënten verkregen in Mali. Het virus werd geïsoleerd op Vero-cellen in een BSL-4-suite bij Rocky Mountain Laboratories. Krediet:NIAID
Met behulp van technische vooruitgang die nog niet was ontwikkeld toen de ebola-uitbraak in 2014 begon, Wetenschappers onder leiding van UC San Francisco voltooiden een proof-of-principle-onderzoek naar een realtime bloedtest op basis van DNA-sequencing die kan worden gebruikt om snel ebola en andere acute infecties te diagnosticeren. De onderzoekers zeiden dat de test zelfs kan worden gebruikt waar laboratoriumruimte en medische infrastructuur schaars zijn.
Charles Chiu, MD, doctoraat, universitair hoofddocent laboratoriumgeneeskunde aan UCSF, leidde een team dat de genetische vingerafdrukken van ebola detecteerde in opgeslagen bloedmonsters van twee Afrikaanse patiënten met acute hemorragische koorts, het voltooien van de diagnose binnen vijf uur na opening van de monsters - de DNA-sequencing zelf duurde slechts 10 minuten.
De meeste commercieel beschikbare of op onderzoek gebaseerde genetische diagnostische tests zijn gericht op specifieke pathogenen. Maar Chiu en UCSF-collega's hebben baanbrekend werk verricht in technieken waarbij verdachte pathogenen niet vooraf moeten worden geïdentificeerd om hun unieke genetische vingerafdrukken te detecteren. Deze onbevooroordeelde benadering van het analyseren van al het DNA in een klinisch monster zonder te weten welke soorten aanwezig zijn, die werd gebruikt bij de ebola-detectie, wordt "metagenomische" analyse genoemd.
Om zulke snelle resultaten te verkrijgen, ontwikkelden de onderzoekers nieuwe analyse- en visualisatiesoftware en gebruikten deze op een laptop om gebruik te maken van een opkomende DNA-sequencing-technologie die bekend staat als nanopore-sequencing.
In dezelfde reeks experimenten, online gepubliceerd in Genoomgeneeskunde op 28 sept. konden de onderzoekers het Chikungunya-virus detecteren, van een Puerto Ricaanse uitbraak, net zo snel in een bloedmonster van een donor zonder symptomen, maar die uiteindelijk meldde koorts en gewrichtspijn te hebben. In een ander voorbeeld van de kracht van de techniek, detectie van hepatitis C-virus in bloed van een geïnfecteerde UCSF-patiënt, aanwezig in een veel lagere concentratie dan de andere virussen, duurde slechts 40 minuten vanaf het begin van de sequencing.
"Deze point-of-care genomische technologie zal bijzonder aantrekkelijk zijn in de derde wereld, waar cruciale middelen, inclusief betrouwbare stroom, laboratoriumruimte, en computationele servercapaciteit, zijn vaak ernstig beperkt, ' zei Chiu.
Veel bedrijven ontwikkelen nanoporiëntechnologie, die individuele nucleïnezuren onderscheidt door de kenmerkende verstoringen die ze creëren in elektrische stromen als ze individueel door microscopisch kleine poriën gaan. Chiu's lab-groep was een van de eersten die $ 1 betaalde, 000 voor toegang tot een experimentele DNA-nanopore-sequencer gemaakt door Oxford Nanopore Technologies, genaamd de MinION. Het apparaat is klein genoeg om in de palm van de hand te passen en wordt gevoed door een USB-verbinding met een laptop.
Vorig jaar, met behulp van een vergelijkbare metagenomische benadering voor de detectie van pathogenen, Chiu werkte samen met UCSF-collega's om een medisch mysterie op te lossen dat werd benadrukt in een New England Journal of Medicine casus. De onderzoekers gebruikten hun software en een andere DNA-sequencing-technologie om al het DNA in een ruggenmergvloeistofmonster te analyseren, wat leidde tot de diagnose van een ongewone maar behandelbare bacteriële oorzaak van encefalitis bij een ernstig zieke jongen uit Wisconsin wiens gezondheid al maanden verslechterde.
Die eerdere analyse duurde twee dagen. De detectie van Ebola in de nieuwe studie was sneller omdat nanopore-sequencing gegevens onmiddellijk en in realtime oplevert, in tegenstelling tot de technologie die werd gebruikt in de zaak Wisconsin, wat veel langer duurt om gegevens voor analyse te verstrekken.
Nanopore-technologie is nieuw en nog steeds foutgevoelig, Chiu zei, maar snelheid en nauwkeurigheid verbeteren in een snel tempo. Met de tijd die nodig is voor DNA-sequencing, analyse en rapportage nu teruggebracht tot minuten, Chiu heeft zijn zinnen gezet op het stroomlijnen en automatiseren van de monstervoorbereidingsstap, die nog enkele uren nodig heeft, voor gebruik in zowel klinische laboratorium- als veldomgevingen.
"Voor zover we weten, dit is de eerste keer dat nanopore-sequencing is gebruikt voor realtime metagenomische detectie van pathogenen in complexe klinische monsters in de setting van menselijke infecties, " Zei Chiu. "Onbevooroordeelde point-of-care-tests voor pathogenen door snelle metagenomische sequencing hebben het potentieel om de diagnose van infectieziekten radicaal te transformeren in zowel klinische als openbare gezondheidsinstellingen."
 Chemici maken playdough / Lego-achtige hybride om kleine bouwstenen te maken
Chemici maken playdough / Lego-achtige hybride om kleine bouwstenen te maken Reparatie van kuilen milieuvriendelijk gemaakt met gruis van afvalwaterzuivering
Reparatie van kuilen milieuvriendelijk gemaakt met gruis van afvalwaterzuivering Chemische reacties voltooien
Chemische reacties voltooien Nieuw katalysatorcomposiet vermindert het gebruik van zeldzame aardelementen
Nieuw katalysatorcomposiet vermindert het gebruik van zeldzame aardelementen Hoe verleng je de levensduur van smeermiddelen?
Hoe verleng je de levensduur van smeermiddelen?
 Geowetenschappers ontdekken dat ondiepe afvalwaterinjectie diepe aardbevingen in Texas veroorzaakt
Geowetenschappers ontdekken dat ondiepe afvalwaterinjectie diepe aardbevingen in Texas veroorzaakt Waarom er meer broeikasgas in de atmosfeer zit dan je je misschien realiseerde
Waarom er meer broeikasgas in de atmosfeer zit dan je je misschien realiseerde Wat maakt dat boeren nieuwe praktijken uitproberen?
Wat maakt dat boeren nieuwe praktijken uitproberen? Grote Brusselse klimaatmars markeert start COP24
Grote Brusselse klimaatmars markeert start COP24 Plastic waait in de wind
Plastic waait in de wind
Hoofdlijnen
- Huismuizen kunnen hun vocalisaties moduleren, afhankelijk van het geslacht van de ontvanger
- Hoe chromosomen vals spelen om kans te maken in een ei te komen
- Zoogdierhersenen identificeren type geur sneller dan ooit werd gedacht
- Jonge vleermuizen leren vleermuisdialecten van hun nestgenoten
- Commerciële voorouderstests kunnen onthullen hoeveel Neanderthaler-DNA u heeft
- Wat hebben we geleerd van het Human Genome Project?
- Deoxyribonucleïnezuur (DNA): structuur, functie en belang
- Is er verteld dat je te gevoelig bent? Je zou een empaat kunnen zijn
- Huidige vee-injecties verhogen het risico op letsel, onderzoek vindt
- Modellering van microzwemmers voor medicijnafgifte
- Nieuwe nanostrategie bestrijdt superbacteriën
- [APRIL FOOLS DAY!] Onderzoekers creëren Rubiks Cube op moleculaire schaal

- Nieuwe optofluïdische technologie maakt gebruik van de kracht van diatomeeën om de sensorprestaties te verbeteren
- Een pagina uit de natuur om betere nanomaterialen te bouwen

 Milieuproblemen in El Paso
Milieuproblemen in El Paso  Eerste wet van de thermodynamica: definitie en voorbeeld
Eerste wet van de thermodynamica: definitie en voorbeeld Luchtvervuiling binnenshuis in India terugdringen
Luchtvervuiling binnenshuis in India terugdringen Wetenschappers gebruiken licht om virale activiteit om te schakelen en ladingen naar cellen te brengen
Wetenschappers gebruiken licht om virale activiteit om te schakelen en ladingen naar cellen te brengen Een neutronenster met een ongebruikelijke magnetische veldstructuur
Een neutronenster met een ongebruikelijke magnetische veldstructuur Krijg ze terwijl ze jong zijn:astronomen vangen een exploderende supernova vroeg op (update)
Krijg ze terwijl ze jong zijn:astronomen vangen een exploderende supernova vroeg op (update) Burgerwetenschappers helpen onderzoekers de waterkwaliteit van Susquehanna te meten
Burgerwetenschappers helpen onderzoekers de waterkwaliteit van Susquehanna te meten Kraakbeen kweken - geen gemakkelijke taak
Kraakbeen kweken - geen gemakkelijke taak
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

