
Wetenschap
Natuurkundige optimaliseert DNA-microscopietechniek om de beeldsnelheid te verbeteren, kleur toevoegen
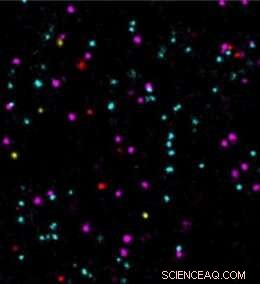
Krediet:S. Strauss/MPI of Biochemistry
Superresolutie fluorescentiemicroscopie kan worden gebruikt om structuren kleiner dan 200 nanometer te visualiseren, d.w.z., onder de diffractiegrens van het licht. Een van de microscopietechnieken, genaamd DNA-VERF, is ontwikkeld door Ralf Jungmann, onderzoeksgroepleider bij de MPI of Biochemistry en Professor voor Experimentele Fysica bij LMU, samen met collega's. De techniek maakt gebruik van korte 'imagers', kleurstof-gelabelde DNA-strengen die tijdelijk op een complementaire manier aan hun doelmoleculen binden om het noodzakelijke "knipperen" te produceren voor superresolutie-reconstructie van de afbeeldingen.
"We hebben onlangs de traditioneel vrij trage acquisitiesnelheid van DNA-PAINT met een orde van grootte verbeterd door het ontwerp van de DNA-sequentie te optimaliseren, ", zegt Jungmann. "Echter, dit ging ten koste van het verlies van multiplexen, wat betekent dat meerdere structuren in de cel niet tegelijkertijd kunnen worden waargenomen, " voegde Jungmann eraan toe. De gelijktijdige waarneming van verschillende eiwitten, echter, is belangrijk voor een beter begrip van complexe signaalcascades tussen tumor en normale cellen.
Dit multiplexvermogen was niet haalbaar in voor snelheid geoptimaliseerde DNA-PAINT, omdat er slechts een enkele geoptimaliseerde sequentie met verbeterde hybridisatiekenmerken beschikbaar was. "We vroegen ons af hoe we multiplex-beeldvorming kunnen toestaan en, tegelijkertijd, de beeldacquisitiesnelheid nog verder verhogen, " zegt Sebastian Strauss, eerste auteur van het artikel en medewerker in de groep van Jungmann.
In de huidige studie, de onderzoekers presenteren een nieuw concept dat de beeldsnelheid met succes heeft verbeterd. Ze profiteerden van het feit dat de frequentie van binding van de imagers aan hun doelstrengen lineair schalen met het aantal beschikbare bindingsplaatsen. "Hoe meer bindingsplaatsen er zijn, hoe sneller de beeldacquisitie verloopt. Echter, het eenvoudig aaneenschakelen van bindingsplaatsen zou leiden tot ongewenst lange koppelingssequenties, mogelijk het verminderen van de haalbare beeldresolutie en het verhogen van niet-specifieke binding, ", zegt Strauss. Om deze problemen te omzeilen, de onderzoekers ontwierpen repetitieve sequentiemotieven, bijv. (TCC)n, die zouden kunnen worden aaneengeschakeld om overlappende bindingsplaatsen te verschaffen, maar die de strenglengte slechts in geringe mate vergroten. "We hebben zes individuele, periodieke sequentiemotieven, waarmee we multiplexen kunnen introduceren in voor snelheid geoptimaliseerde DNA-PAINT, " zei Strauss. "In combinatie met eerdere verbeteringen, we kunnen DNA-PAINT nu met een factor 100 versnellen, ", voegt Jungmann eraan toe.
Om de nieuwe sequentiemotieven te optimaliseren en hun verbeteringen te benchmarken, de groep gebruikte DNA-origamistructuren, die zelf samengesteld zijn, DNA-objecten van nanometerformaat die autonoom in vooraf gedefinieerde vormen vouwen. Deze structuren kunnen worden gebruikt om DNA-PAINT-bindingsplaatsen te rangschikken die precies op afstand van b.v. afstanden van 5 nm. Dit stelde de onderzoekers in staat om de verbeteringen aan DNA-PAINT onder gedefinieerde omstandigheden te evalueren. "De nieuwe geoptimaliseerde DNA-sequenties stelden ons in staat om zes verschillende DNA-origamistructuren op te lossen in plaats van slechts één, in slechts een paar minuten, ", legt Strauss uit.
"We zijn verheugd om de nu verder verbeterde beeldsnelheid in DNA-PAINT toe te passen om biologische vragen aan te pakken. tumormarkers konden voorheen alleen langzaam en niet duidelijk op een enkel molecuulniveau worden onderzocht. In onze studie, de meting van vier verschillende tumormarkers bevestigt een snelle en nauwkeurige analyse van hun moleculaire posities en interacties. Dit zou belangrijke inzichten kunnen opleveren voor de ontwikkeling van geneesmiddelen en hun werkingsmechanismen, ’ besluit Jungmann.
 Onderzoekers vinden een nieuwe toepassing voor afval
Onderzoekers vinden een nieuwe toepassing voor afval Directe methaanomzetting onder milde omstandigheden door thermo-, elektro- of fotokatalyse beoordeeld
Directe methaanomzetting onder milde omstandigheden door thermo-, elektro- of fotokatalyse beoordeeld Wat zijn de zes meest overvloedige elementen die voorkomen in levende organismen?
Wat zijn de zes meest overvloedige elementen die voorkomen in levende organismen?  Wat is de chemische MDI?
Wat is de chemische MDI?  JCESR legt basis voor veiliger, batterijen die langer meegaan
JCESR legt basis voor veiliger, batterijen die langer meegaan
 Verbetering van de nauwkeurigheid van projecties van klimaatmodellen met opkomende beperkingen
Verbetering van de nauwkeurigheid van projecties van klimaatmodellen met opkomende beperkingen Suomi NPP-satelliet ziet Rosa intensiveren tot tiende orkaan in de oostelijke Stille Oceaan
Suomi NPP-satelliet ziet Rosa intensiveren tot tiende orkaan in de oostelijke Stille Oceaan Studie zegt dat aardbevingen in Zuid-Californië de spanning op de grote breuklijn verhoogden
Studie zegt dat aardbevingen in Zuid-Californië de spanning op de grote breuklijn verhoogden Wat we weten (en niet weten) over zeespiegelstijging
Wat we weten (en niet weten) over zeespiegelstijging Bij gebruik van pyriet om de oceaan en atmosfeer van de aarde te begrijpen:denk lokaal, niet wereldwijd
Bij gebruik van pyriet om de oceaan en atmosfeer van de aarde te begrijpen:denk lokaal, niet wereldwijd
Hoofdlijnen
- Nieuw apparaat zoomt in op microbengedrag op de juiste schaal
- Sporen van historische rendierweide zijn na 100 jaar nog steeds te zien
- In de mangoestenmaatschappij immigranten zijn een bonus - als ze tijd krijgen om zich te vestigen
- Onderzoek verduidelijkt de functie van de nucleaire hormoonreceptor in planten
- DNA is gecondenseerd in welke fasen?
- Waarom zijn mensen hun staart kwijtgeraakt?
- Waarom helpen rijmpjes mensen dingen te onthouden?
- Hoe virussen de manier veranderen waarop we naar evolutie kijken
- Welke gebeurtenis volgt DNA-replicatie in een celcyclus?
- Wetenschappers verfijnen de zoektocht naar donkere materie

- Fusiewetenschappers vinden inspiratie in atmosferische fluitjes
- Unieke eigenschappen van kwantummateriaal voor het eerst uitgelegd

- Een miniatuur laserachtig apparaat voor oppervlakteplasmonen

- Einstein-puzzel opgelost, ontbrekende pagina duikt op in nieuwe schat

 Een universele voedsel- en alarmsignaal gevonden in zoogdierbloed
Een universele voedsel- en alarmsignaal gevonden in zoogdierbloed Amazon vuurt:acht manieren waarop je kunt helpen
Amazon vuurt:acht manieren waarop je kunt helpen Belang van Phytoplankton
Belang van Phytoplankton Europees parlement roept klimaatnoodtoestand uit
Europees parlement roept klimaatnoodtoestand uit 70% van Californië zit officieel in een droogte. Hier zijn enkele huishoudelijke tips om water te besparen
70% van Californië zit officieel in een droogte. Hier zijn enkele huishoudelijke tips om water te besparen Minuscule moleculaire bewegingen kunnen leiden tot efficiëntere biobrandstofcellen
Minuscule moleculaire bewegingen kunnen leiden tot efficiëntere biobrandstofcellen Voor deze baleinwalvissen, jagen vereist weinig meer dan watertrappelen
Voor deze baleinwalvissen, jagen vereist weinig meer dan watertrappelen Wat als we de samenleving niet besturen op basis van de markt, maar op bewijs?
Wat als we de samenleving niet besturen op basis van de markt, maar op bewijs?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | German | Dutch | Danish | Norway | Swedish |

-
Wetenschap © https://nl.scienceaq.com

