
Wetenschap
Slow motion golven van springende genen in het menselijk genoom

Natuurkundigen Nigel Goldenfeld (links) en Chi Xue, aan het Carl R. Woese Instituut voor Genomische Biologie aan de Universiteit van Illinois in Urbana-Champaign. Krediet:Kathryn Faith.
De natuur zit vol met parasieten - organismen die gedijen en zich vermenigvuldigen ten koste van een andere soort. Verrassend genoeg, dezelfde concurrerende rollen van parasiet en gastheer kunnen worden gevonden in de microscopische moleculaire wereld van de cel. Een nieuwe studie door twee onderzoekers uit Illinois heeft aangetoond dat dynamische elementen in het menselijk genoom met elkaar interageren op een manier die sterk lijkt op de patronen die te zien zijn in populaties van roofdieren en prooien.
De bevindingen, gepubliceerd in Fysieke beoordelingsbrieven door natuurkundigen Chi Xue en Nigel Goldenfeld, zijn een belangrijke stap in de richting van het begrijpen van de complexe manieren waarop genomen veranderen gedurende de levensduur van individuele organismen, en hoe ze zich over generaties ontwikkelen.
"Dit zijn genen die actief zijn en in realtime genoombewerkingen uitvoeren in levende cellen, en dit is een begin om ze echt in veel meer detail te begrijpen dan eerder is gedaan, " zei Goldenfeld, die het onderzoeksthema Biocomplexity leidt bij het Carl R. Woese Institute for Universal Biology (IGB). "Dit helpt ons de evolutie van complexiteit en de evolutie van genomen te begrijpen."
De studie werd ondersteund door Centre for the Physics of Living Cells, een Physics Frontiers Center in Illinois, ondersteund door de National Science Foundation, en het NASA Astrobiology Institute for Universal Biology in Illinois, die Goldenfeld regisseert.
Goldenfeld en Xue begonnen aan dit werk vanwege hun interesse in transposons, kleine stukjes DNA die zichzelf tijdens de levensduur van een cel van het ene deel van het genoom naar het andere kunnen verplaatsen - een vermogen dat hen de naam 'springende genen' heeft opgeleverd. collectief, verschillende soorten transposons vormen bijna de helft van het menselijk genoom. Als ze zich verplaatsen, ze kunnen mutaties veroorzaken in of de activiteit van een functioneel gen veranderen; transposons kunnen daarom nieuwe genetische profielen creëren in een populatie waarop natuurlijke selectie kan inwerken, op een positieve of negatieve manier.
De Illinois-onderzoekers wilden meer leren over hoe evolutie op dit niveau werkt, het niveau van hele organismen, door te kijken naar het metaforische ecosysteem van het menselijk genoom. In deze weergave, de fysieke structuur van het DNA waaruit het genoom bestaat, werkt als een omgeving, waarin twee soorten transposons, lange afgewisselde nucleaire elementen (LINE's) en korte afgewisselde nucleaire elementen (SINE's), een concurrentieverhouding met elkaar hebben. Om te repliceren, SINE's stelen de moleculaire machinerie die LINE's gebruiken om zichzelf te kopiëren, een beetje zoals een koekoeksvogel andere vogels bedriegt om haar kuikens voor haar groot te brengen terwijl ze hun eigen kuikens in de steek laten.
Met hulp van Oleg Simakov, een onderzoeker aan het Okinawa Institute of Science and Technology, Xue en Goldenfeld concentreerden zich op de biologie van L1-elementen en Alu-elementen, respectievelijk voorkomende typen LINE's en SINE's in het menselijk genoom.
De onderzoekers hebben methoden uit de moderne statistische fysica overgenomen en de interactie tussen Alu- en L1-elementen wiskundig gemodelleerd als een stochastisch proces - een proces dat is ontstaan uit toevallige interacties. Deze methode is met succes toegepast in de ecologie om interacties tussen roofdieren en prooien te beschrijven; Xue en Goldenfeld simuleerden de bewegingen van transposons in het menselijk genoom met dezelfde wiskundige methode. Hun modellen bevatten een gedetailleerde beschrijving van hoe Alu-elementen de moleculaire machinerie stelen die L1-elementen gebruiken om zichzelf te kopiëren.
De resultaten van Xue en Goldenfeld voorspelden dat populaties van LINE- en SINE-elementen in het genoom naar verwachting zullen oscilleren zoals die van, bijvoorbeeld, wolven en konijnen misschien.
"We realiseerden ons dat de interactie van de transposons eigenlijk veel leek op de interactie tussen roofdier en prooi in de ecologie, "zei Xue. "We kwamen op het idee, waarom passen we niet hetzelfde idee van roofdier-prooi-dynamiek toe? . .we verwachtten de oscillaties te zien die we zien in het roofdier-prooimodel. Dus we deden eerst de simulatie en we zagen de oscillaties die we verwachtten, en we werden echt enthousiast."
Met andere woorden, te veel SINE's en de LINE's beginnen te lijden, en al snel zijn er niet genoeg voor alle SINE's om te exploiteren. SINE's beginnen te lijden, en de LINE's maken een comeback. Het model van Xue en Goldenfeld deed de verrassende voorspelling dat deze oscillaties plaatsvinden over een tijdschaal die langer is dan de menselijke levensduur - golven van Alu-elementen en L1-elementen die elkaar in slow motion duwen en trekken over generaties van de menselijke genomen die ze dragen.
"Het meest verhelderende aspect van de studie voor mij was het feit dat we echt de tijdschalen konden berekenen, en zien dat het mogelijk is dat we deze dingen kunnen waarnemen, " zei Goldenfeld. "We hebben een voorspelling voor wat er gebeurt in afzonderlijke cellen, en we kunnen misschien een experiment doen om deze dingen te observeren, hoewel de periode langer is dan de levensduur van een enkele cel."
In een verwante studie, Het laboratorium van Goldenfeld heeft samengewerkt met het laboratorium van collega-fysicus en IGB Biocomplexity-onderzoeksthemalid Thomas Kuhlman om de bewegingen van transposons in de genomen van levende cellen te visualiseren. Door gebruik te maken van dit soort innovatieve technologie, en door de geschiedenis van moleculaire evolutie bij andere soorten te bestuderen, Goldenfeld en Xue hopen enkele van de voorspellingen van hun model te testen en inzicht te blijven krijgen in de dynamische wereld van het genoom.
 Verdunningen maken
Verdunningen maken We huwen wanorde met orde:de effecten van geometrische wanorde op vloeistoffen en vaste stoffen in mesoporeuze materialen
We huwen wanorde met orde:de effecten van geometrische wanorde op vloeistoffen en vaste stoffen in mesoporeuze materialen Single fotonen emissie van geïsoleerde monolaag eilanden van InGaN
Single fotonen emissie van geïsoleerde monolaag eilanden van InGaN efficiënt, stabiele thermo-elektrische module op basis van hoogwaardige vloeistofachtige materialen
efficiënt, stabiele thermo-elektrische module op basis van hoogwaardige vloeistofachtige materialen Cellulaire stress verdediging
Cellulaire stress verdediging
 Interactieve kaart laat zien hoe de Britse eilanden werden beïnvloed door de laatste glaciale cyclus 22, 000 jaar geleden
Interactieve kaart laat zien hoe de Britse eilanden werden beïnvloed door de laatste glaciale cyclus 22, 000 jaar geleden Zorgen voor stabiliteit op lange termijn van pesticiden in honing
Zorgen voor stabiliteit op lange termijn van pesticiden in honing Mushroom Hunting In Wisconsin
Mushroom Hunting In Wisconsin  Hydrothermale ventilatieopeningen kunnen oude koolstof toevoegen aan oceaanwater
Hydrothermale ventilatieopeningen kunnen oude koolstof toevoegen aan oceaanwater Wisselende stromingen sleutel tot het verwijderen van nitraat uit stromen, studie vondsten
Wisselende stromingen sleutel tot het verwijderen van nitraat uit stromen, studie vondsten
Hoofdlijnen
- Sumatraanse neushoorns zijn nooit hersteld van verliezen tijdens het Pleistoceen, genoom bewijs toont
- Maatregelen voor welzijn en duurzaamheid heroverwegen van lokale naar mondiale schaal
- Hoe stuifmeel werkt
- Onderzoeker rapporteert belangrijke componenten van antivirale verdediging van honingbijen
- Drie voorbeelden van protisten met wetenschappelijke namen
- Achter de puppy-hondenogen
- Waarom zijn er geen zeeslangen in de Atlantische Oceaan?
- Welke soorten moleculen kunnen door het plasmamembraan passeren via eenvoudige diffusie?
- Moet je intelligent zijn om slecht te zijn?
- Aan de rand van chaos:nieuwe methode voor analyse van stabiliteit van exoplaneten
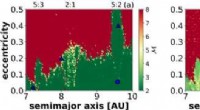
- Nieuwe soliton-laserpulsen leveren hoge energie in een biljoenste van een seconde

- Nieuwe lasertechniek maakt krachtigere - en kleinere - deeltjesversnellers mogelijk
- Een nieuw stukje van de kwantumcomputerpuzzel

- Een dieper begrip van kwantumchaos kan de sleutel zijn tot kwantumcomputers

 Grafeen onthult zijn magnetische persoonlijkheid
Grafeen onthult zijn magnetische persoonlijkheid Gespecialiseerde weeftechnieken maken een nieuw hitteschild mogelijk voor planetaire verkenning
Gespecialiseerde weeftechnieken maken een nieuw hitteschild mogelijk voor planetaire verkenning Studie introduceert nieuwe vacuümkanaaltransistors op nanoschaal
Studie introduceert nieuwe vacuümkanaaltransistors op nanoschaal Alles zal ooit verbinding maken met internet, en deze biobatterij zou kunnen helpen
Alles zal ooit verbinding maken met internet, en deze biobatterij zou kunnen helpen Menselijke migratie uit Afrika is mogelijk gevolgd door moessons in het Midden-Oosten
Menselijke migratie uit Afrika is mogelijk gevolgd door moessons in het Midden-Oosten NASA's TROPICS Pathfinder-satelliet produceert wereldwijde beelden van het eerste licht en legt orkaan Ida . vast
NASA's TROPICS Pathfinder-satelliet produceert wereldwijde beelden van het eerste licht en legt orkaan Ida . vast Nieuwe afstandsmetingen versterken de uitdaging voor het basismodel van het universum
Nieuwe afstandsmetingen versterken de uitdaging voor het basismodel van het universum Elementaire wiskundige breuken
Elementaire wiskundige breuken
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

