
Wetenschap
Nieuwe software om waardevolle verbindingen te ontdekken
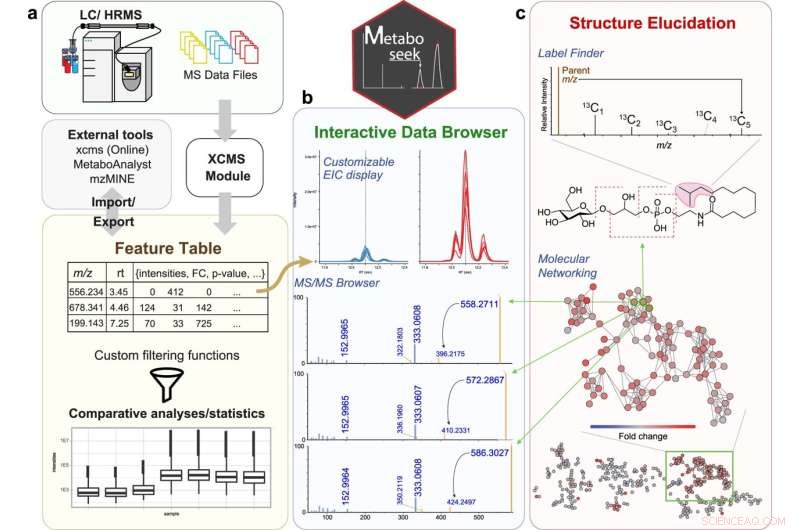
Vergelijkende metabolomics met Metaboseek. a Metaboseek bevat een geïntegreerde XCMS-module voor functiedetectie en functiegroepering (met CAMERA-annotatie) en accepteert functietabellen die door andere software zijn gegenereerd. b Functies kunnen worden geannoteerd en geprioriteerd met behulp van uitgebreide filteropties en geïntegreerde statistiekentools. Onbewerkte gegevens voor elke moleculaire functie kunnen snel worden doorzocht, inclusief bijbehorende EIC's, MS1 en MS/MS-spectra. c De gegevensbrowser werkt samen met een reeks hulpmiddelen voor structuuropheldering, bijvoorbeeld op SIRIUS gebaseerde moleculaire formule- en structuurvoorspelling, de Label Finder om isotoop-gelabelde verbindingen te identificeren, en de MS/MS-patroonzoeker om MS-kenmerken met karakteristieke fragmentatiepatronen te identificeren. Krediet:Natuurcommunicatie (2022). DOI:10.1038/s41467-022-28391-9
Als postdoctoraal onderzoeksmedewerker in het laboratorium van BTI-faculteitslid Frank Schroeder, zag Max Helf zijn laboratoriumgenoten voortdurend worstelen bij het analyseren van gegevens. Dus besloot hij er iets aan te doen en ontwikkelde een gratis, open-source app genaamd Metaboseek, die nu essentieel is voor het werk van het lab.
Het Schroeder-lab bestudeert de rondworm Caenorhabditis elegans, een van de meest succesvolle modelsystemen voor de menselijke biologie, om nieuwe metabolieten te ontdekken die evolutionair geconserveerde signaalroutes bepalen en die nuttig kunnen zijn als aanknopingspunten voor de ontwikkeling van nieuwe geneesmiddelen of landbouwchemicaliën. De onderzoekers volbrengen deze taak door de metabolieten tussen twee verschillende wormpopulaties te vergelijken - een proces dat vergelijkende metabolomics wordt genoemd.
Aangezien monsters routinematig meer dan 100.000 verbindingen bevatten, zijn computationele benaderingen essentieel om de analyse uit te voeren.
Het team vertrouwde op softwarepakketten die niet het vereiste niveau van flexibiliteit boden om analyseparameters gemakkelijk aan te passen. Die beperking, en het ontbreken van een geschikte grafische gebruikersinterface, betekende dat de collega's van Helf voor de omslachtige taak stonden om bergen gegevens visueel te inspecteren - bijvoorbeeld om mogelijke valse positieven op te sporen - en tussen verschillende andere softwaretools te springen om die zinloze resultaten.
"Het leek me gewoon erg inefficiënt en ik kon de tekortkomingen van andere softwareoplossingen voor dit probleem niet overwinnen", zei Helf. "Ik dacht dat er een eenvoudigere manier moest zijn, dus begon ik code te schrijven voor mijn eigen software."
Helf ontwikkelde de eerste versie van zijn software in 2017 en bleef deze de komende twee jaar verbeteren. "Naast het aanpakken van de problemen waar mijn labgenoten al mee te maken hadden, sprak ik met hen over wat hen nog meer tegenhield - wat ze wilden doen maar niet eens probeerden - en die functies in de app ingebouwd", zei Helf, die nu een productmanager bioinformatica bij het proteomics-bedrijf Biognosys AG. "Ik wilde dat deze nieuwe tool gebruiksvriendelijk en toegankelijk zou zijn voor iedereen die aan chemische biologie doet."
Het resultaat was Metaboseek, een app met een grafische interface die meerdere data-analysetools bevat die niet-coderende onderzoekers anders niet zouden hebben. De app stroomlijnt de analyse van vergelijkende metabolomics-gegevens door de onderzoeker te helpen bepalen welke gegevensfuncties echt zijn en hen dieper in die functies te laten graven, allemaal binnen dezelfde tool.
"Max deed dit zonder dat ik erom vroeg", zei Schroeder. "Voordat ik wist dat dit gebeurde, was er Metaboseek. We begonnen het te gebruiken, en nu zouden ons lab en vele medewerkers niet zonder kunnen bestaan."
In een studie gepubliceerd in Nature Communications op 10 februari leverde het team van Schroeder een proof-of-concept voor Metaboseek door het toe te passen op een belangrijke vetmetabolismeroute die nog niet was onderzocht:de α-oxidatieroute in C. elegans die helpt bij het afbreken van een klasse vetzuren.
Met behulp van Metaboseek ontdekte het team dat rondwormen zonder een sleutelgen in de α-oxidatieroute honderden eerder niet-gerapporteerde metabolieten verzamelden. De bevindingen zijn belangrijk omdat α-oxidatie een fundamentele biochemische route is in wormen die bij mensen behouden blijft, zei Schroeder.
"Bennett Fox deed het scheikundewerk, dus deze studie was een mooie samenwerking tussen de twee postdocs", voegde Schroeder toe, die ook professor is aan de afdeling Scheikunde en Chemische Biologie van Cornell University.
Volgens Schroeder en Helf zijn er een paar redenen waarom er niet veel goede analytische tools zijn om metabolomics-gegevens te vergelijken. Ten eerste is vergelijkende metabolomics een relatief jong veld in vergelijking met andere data-intensieve gebieden van de biologie zoals genomics (die zich richten op DNA) en proteomics (die zich richten op eiwitten), dus er is niet genoeg tijd geweest om softwaretools en database-infrastructuur te ontwikkelen .
Bovendien is de komst van betaalbare massaspectrometers met ultrahoge resolutie voor het verzamelen van metabolomics-gegevens de afgelopen tien jaar misschien wel meer dan tien keer zo groot geworden als de hoeveelheid gegevens die één monster kan genereren, waardoor er een nog grotere behoefte is ontstaan aan geavanceerde tools die de mee met de stortvloed aan gegevens.
Metaboseek komt aan deze behoeften tegemoet met een reeks functies voor het analyseren van verschillende soorten gegevens ter ondersteuning van de identificatie van verbindingen, het bepalen van de structuur, het toewijzen van metabolieten aan families op basis van structurele overeenkomsten, het volgen van radioactief gelabelde verbindingen en meer. + Verder verkennen
Een R-pakket voor uitgebreide data-analyse van op peptiden en eiwitten gerichte bottom-up proteomics-data
 Hoe de molaire hitte van neutralisatie te berekenen
Hoe de molaire hitte van neutralisatie te berekenen De witste verf is hier - en het is de coolste. Letterlijk.
De witste verf is hier - en het is de coolste. Letterlijk. Schudden en roeren:wetenschappers leggen het vervormingseffect van schokgolven op een materiaal vast
Schudden en roeren:wetenschappers leggen het vervormingseffect van schokgolven op een materiaal vast Wetenschappers onthullen verschillende substraatbindingsmodus in o-succinylbenzoyl-CoA-synthetase
Wetenschappers onthullen verschillende substraatbindingsmodus in o-succinylbenzoyl-CoA-synthetase Op eieren gebaseerde griepvaccins:niet alles wat ze zijn?
Op eieren gebaseerde griepvaccins:niet alles wat ze zijn?
 4 beroemde gigantische bomen ongedeerd door vuur in Sequoia National Park
4 beroemde gigantische bomen ongedeerd door vuur in Sequoia National Park Nieuw model onthult voorheen niet-herkende complexiteit van oceanische aardbevingszones
Nieuw model onthult voorheen niet-herkende complexiteit van oceanische aardbevingszones Wat is een semi-droog klimaat?
Wat is een semi-droog klimaat?  Nieuwe PFAS omvatten 24% van die gemeten in het bloed van Wilmington, Inwoners van Noord-Carolina
Nieuwe PFAS omvatten 24% van die gemeten in het bloed van Wilmington, Inwoners van Noord-Carolina Nog een milde algenbloeivoorspelling voor Lake Erie deze zomer
Nog een milde algenbloeivoorspelling voor Lake Erie deze zomer
Hoofdlijnen
- Vissen gebruiken doofheidsgen om waterbeweging waar te nemen
- Een titerrapport lezen
- De lange en korte van een supergen voor efficiënte bestuiving
- Een enzym dat de vorming van het DNA katalyseert Molecuul
- Dieren die menselijke DNA-sequenties delen
- Bosbessen en hun bestuivers komen niet van nature voor in Zuid-Afrika, maar lokale honingbijen kunnen helpen
- Hoe hebben Watson en Crick baseparing bepaald?
- Drummen bij spechten lijkt neurologisch op zingen bij zangvogels
- Onderzoek naar de trends waarmee de Europese landbouw te maken zal krijgen
- Voedingswetenschappers creëren nieuwe magnetische nanodeeltjes voor snelle screening van bestrijdingsmiddelenresiduen in groenten

- Defecte koolstof vereenvoudigt de productie van waterstofperoxide

- Wetenschappers ontrafelen eiwit-nanodeeltjes interacties, pad naar drug-sensing tool voorstellen

- Fluor versnelt de groei van tweedimensionale materialen
- Door stro opnieuw te gebruiken, kunnen boeren meer voedsel verbouwen met minder water en kunstmest

 Verdere aanjagers van oceaandeoxygenatie geïdentificeerd
Verdere aanjagers van oceaandeoxygenatie geïdentificeerd Lockdowns maken mensen eenzaam:drie stappen die we nu kunnen nemen om elkaar te helpen
Lockdowns maken mensen eenzaam:drie stappen die we nu kunnen nemen om elkaar te helpen Tienvoudige verbetering in vloeibare batterijen betekent dat het tanken van elektrische auto's minuten kan duren
Tienvoudige verbetering in vloeibare batterijen betekent dat het tanken van elektrische auto's minuten kan duren Hoe u betere advertenties voor adoptie van huisdieren schrijft
Hoe u betere advertenties voor adoptie van huisdieren schrijft Ingenieurs creëren sociale media-infrastructuur voor noodbeheer
Ingenieurs creëren sociale media-infrastructuur voor noodbeheer Hoe de boog van St. Louis tegen alle verwachtingen in staat
Hoe de boog van St. Louis tegen alle verwachtingen in staat  Exosome nanoporator:een nanofluïdisch apparaat om op exosoom gebaseerde voertuigen voor medicijnafgifte te ontwikkelen
Exosome nanoporator:een nanofluïdisch apparaat om op exosoom gebaseerde voertuigen voor medicijnafgifte te ontwikkelen Kubieke voet van een cilinder berekenen
Kubieke voet van een cilinder berekenen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

