
Wetenschap
Een veelzijdige, geïntegreerde workflow voor interactie proteomics
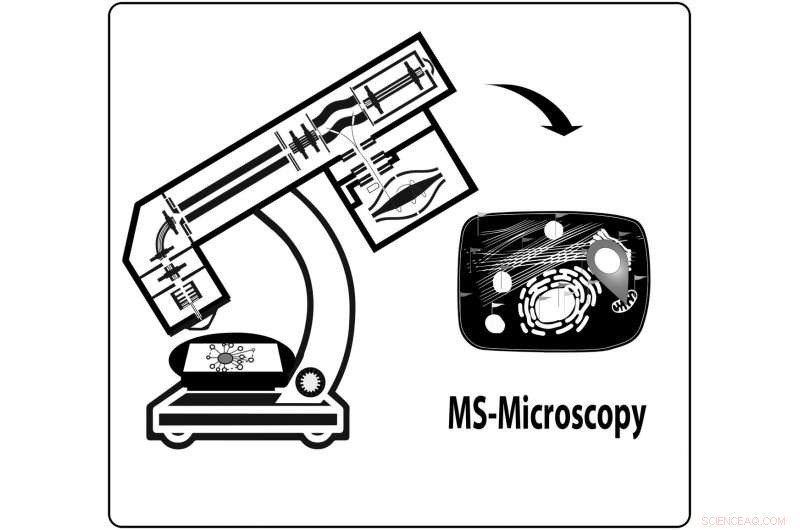
Onderzoekers ontwikkelden een geïntegreerde workflow voor interactie proteomics, die bijna net zo veelzijdig is als het Zwitserse zakmes. Krediet:Varjosalo Lab
Eiwitten functioneren niet op zichzelf, en hun interacties met andere eiwitten bepalen hun cellulaire functies. Daarom, gedetailleerd begrip van eiwit-eiwitinteracties (PPI's) is de sleutel voor het ontcijferen van de regulatie van cellulaire netwerken en routes. Deze complexe netwerken van stabiele en voorbijgaande associaties kunnen worden bestudeerd door middel van affiniteitszuiveringsmassaspectrometrie (AP-MS) en complementaire op nabijheid gebaseerde labelingsmethoden zoals BioID.
In een studie gepubliceerd in Natuurcommunicatie , een onderzoeksteam onder leiding van Dr. Markku Varjosalo aan de Universiteit van Helsinki ontwikkelde een geoptimaliseerde en geïntegreerde aanpak die AP-MS en BioID combineert in een enkele workflow. Naast het benutten van de voordelen van beide strategieën, de auteurs laten zien dat hun aanpak identificatie en kwantificering van eiwit-eiwit-interacties en eiwitcomplex-stoichiometrieën mogelijk maakt, identificatie van tijdelijke of nabije interacties met BioID, visualisatie van het aas-eiwit en de proximale interactoren met immunofluorescentiemicroscopie, en het definiëren van de moleculaire context met MS-microscopie met behulp van de referentiegegevensset die is verkregen door het identificeren van proximale interactoren voor bonafide subcellulaire lokalisatiemarkers.
De auteurs laten zien dat MS-microscopie het mogelijk maakt om het bestudeerde eiwit toe te wijzen aan de juiste cellulaire of zelfs subcellulaire locatie in een nog hogere resolutie dan met confocale microscopie. "Deze studie is een continuüm van onze rigoureuze inspanningen bij het ontwikkelen van nieuwe systeembiologische hulpmiddelen voor het bestuderen van de moleculaire interacties gevormd door eiwitten. We hebben eerder bewezen dat AP-MS een zeer reproduceerbare methode is, die ook geschikt is voor grootschalige en interlaboratoriumstudies", Dr. Varjosalo zegt. "Onze nieuw ontwikkelde geïntegreerde workflow en de referentie-moleculaire context-proteoomkaart, maakt een gemakkelijke manier mogelijk om de moleculaire lokalisatie van (veel) eiwitten te onderzoeken. De ontwikkelde MAC-tag en de geïntegreerde aanpak moeten versterken, niet alleen de interactie proteomics gemeenschap, maar ook cel, moleculaire en structurele biologen, met een experimenteel bewezen geïntegreerde workflow voor het gedetailleerd in kaart brengen van de fysieke en functionele interacties en de moleculaire context van eiwitten in menselijke cellen."
 In lijn:het eenvoudige ontwerp en de controle van MOF elektrische stroom
In lijn:het eenvoudige ontwerp en de controle van MOF elektrische stroom Cryo-elektronenmicroscopische structuur van een proton-geactiveerd chloridekanaal genaamd TMEM206
Cryo-elektronenmicroscopische structuur van een proton-geactiveerd chloridekanaal genaamd TMEM206 Zichtbaar licht gebruiken om koolstofdioxide efficiënt af te breken
Zichtbaar licht gebruiken om koolstofdioxide efficiënt af te breken Met molybdeen gecoate katalysator splitst water efficiënter voor waterstofproductie
Met molybdeen gecoate katalysator splitst water efficiënter voor waterstofproductie Hoe beïnvloedt zure regen gebouwen en standbeelden?
Hoe beïnvloedt zure regen gebouwen en standbeelden?
Hoofdlijnen
- Hoe Charles Darwin werkte
- Hoe reageert het excretiesysteem op fysieke activiteit?
- Science Fair Project Ideas: Equine
- Vier grote groepen organische verbindingen die levende organismen samenstellen
- Nieuwe filmtechniek onthult bacteriële signalering in scherpere resolutie
- milities, stropers richten grote schade aan in het wild in Centraal-Afrika:monitor
- Emotionele toestanden ontdekt in vissen
- Waarom lijken haaienbeten dodelijker in Australië dan elders?
- Statistische modellering helpt visserijbeheerders invasieve soorten te verwijderen
- Onderzoekers verkrijgen een belangrijke chemische verbinding

- Galvaniseren levert hoge energie, krachtige batterijen
- Onderzoekers ontdekken dat het toevoegen van zeldzame-aarde-elementen aan piëzo-elektrische kristallen de prestaties drastisch verbetert
- Nieuwe high-throughput screeningmethode ontwikkeld voor ketonen
- Eiwitraket verhoogt kans op doorbraken van ziekten

 Uitstulpingen in vulkanen kunnen worden gebruikt om uitbarstingen te voorspellen
Uitstulpingen in vulkanen kunnen worden gebruikt om uitbarstingen te voorspellen Airbnb koopt HotelTonight-app om reisaanbod te verbreden
Airbnb koopt HotelTonight-app om reisaanbod te verbreden Dit mysterieuze seismische evenement had een gigantische onderwatervulkaan kunnen veroorzaken
Dit mysterieuze seismische evenement had een gigantische onderwatervulkaan kunnen veroorzaken Overstromingstol in India stijgt naar 144 als wegen, snelwegen afgesneden
Overstromingstol in India stijgt naar 144 als wegen, snelwegen afgesneden Klimaatverandering test de veerkracht van inheemse planten tegen vuur, van essenbossen tot gymea-lelies
Klimaatverandering test de veerkracht van inheemse planten tegen vuur, van essenbossen tot gymea-lelies Berekening van driehoeks- en vierzijdige zijlengten
Berekening van driehoeks- en vierzijdige zijlengten Hoe werkt een zuigercompressor?
Hoe werkt een zuigercompressor?  Eerste licht voor SPIRou, exoplaneet jager
Eerste licht voor SPIRou, exoplaneet jager
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Portuguese | Swedish | German | Dutch | Danish | Norway | Italian | Spanish |

-
Wetenschap © https://nl.scienceaq.com

