
Wetenschap
Moleculaire interacties meten
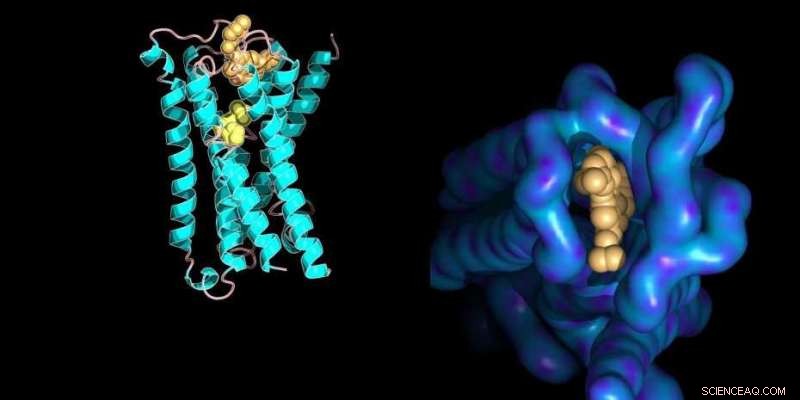
Moleculair model van een eiwitmembraanreceptor gebonden aan zijn kleine molecuulliganden met structurele details van de allosterische bindingsplaats (rechts). Krediet:Ilaria Piazza / ETH Zürich / PDB-databaseinvoer 4MQT
Onderzoekers van ETH Zürich hebben een nieuwe benadering gebruikt om voorheen onbekende interacties tussen eiwitten en kleine metabole moleculen in bacteriële cellen te ontdekken. De techniek kan ook worden gebruikt om het effect van medicijnen te testen.
De wetenschap heeft nu een nieuwe "omics" - een interactomics, die zich bezighoudt met de interacties tussen eiwitten en kleine moleculen. Na eerder te hebben gefocust op genomics of proteomics, systeembiologen kunnen nu eiwit-metaboliet interactomics aan de mix toevoegen. Genomics houdt zich bezig met de systematische analyse van alle genen van een organisme, terwijl proteomics zich bezighoudt met de hele set eiwitten binnen een biologische eenheid.
Met eiwit-metaboliet interactomics, de groep onder leiding van Paola Picotti, Professor van Moleculaire systeembiologie bij ETH Zürich, heeft nu een nieuw niveau toegevoegd aan de bestaande "omics". De onderzoekers publiceerden onlangs een studie in het tijdschrift Cell, waarin ze voor het eerst systematisch de interacties van alle eiwitten met metabolieten (kleine metabole moleculen) op het niveau van het hele proteoom analyseerden en kwantificeerden, het leggen van de onderlinge relaties.
Verschillende snijplaatsen
De onderzoekers hebben aangetoond hoeveel van de eiwitten en enzymen die aanwezig zijn in een E. coli-bacteriecel een interactie aangaan met metabolieten. Om dit te doen, ze gebruikten een benadering die bekend staat als beperkte proteolyse (LiP), gecombineerd met massaspectrometrische analyse.
De onderzoekers extraheren celvloeistof, die eiwitten bevat, uit de bacteriecellen. Vervolgens voegden ze aan elk monster een metaboliet toe en lieten het interageren met de eiwitten. Eindelijk, ze knippen de eiwitten in kleinere stukjes (peptiden) met behulp van "moleculaire schaar". In totaal, de onderzoekers testten op deze manier 20 verschillende metabolieten en hun interacties met eiwitten.
Wanneer een eiwit interageert met een metaboliet, of het zich nestelt op de actieve plaats van het eiwit of zich hecht aan een andere plaats, de eiwitstructuur is veranderd. De "moleculaire schaar" sneed het vervolgens op verschillende plaatsen af van de oorspronkelijke structuur, wat resulteert in een andere set peptiden.
Met behulp van de massaspectrometer, de onderzoekers hebben alle stukjes in het monster gemeten en de verkregen gegevens in een computer ingevoerd om de structurele verschillen en veranderingen te reconstrueren, en waar in het eiwit deze zijn gelokaliseerd.
Honderden nieuwe interacties ontdekt
Kennis over het eiwit-metaboliet interactoom (de interacties tussen eiwitten en metabolieten en de daarbij behorende moleculaire (signaal)netwerken), was voorheen zeer beperkt in vergelijking met wat bekend was over interacties tussen verschillende eiwitten of tussen eiwitten en DNA of RNA. Het onderzoek heeft deze kennis nu sterk vergroot.
Met behulp van deze aanpak met E. coli, Picotti en haar team ontdekten er ongeveer 1, 650 verschillende eiwit-metaboliet interacties, waarvan meer dan 1, 400 waren voorheen onbekend. Ook kwamen duizenden bindingsplaatsen op eiwitten aan het licht waaraan metabolieten zich kunnen hechten. "Hoewel het metabolisme van E. coli en geassocieerde moleculen al heel goed bekend is, we zijn erin geslaagd veel nieuwe interacties en de bijbehorende bindingsplaatsen te ontdekken, ", zegt Picotti. Dit bewijst het enorme potentieel van de methode:"De gegevens die we met deze techniek produceren, zullen helpen bij het identificeren van nieuwe regulerende mechanismen, onbekende enzymen en nieuwe metabolische reacties in de cel."
Structurele verandering regelt activiteit
In hun studie hebben de onderzoekers laten ook zien dat kleine metabole moleculen zich bij voorkeur binden aan (en dus reguleren) eiwitten waarvan de concentratie min of meer constant is in de tijd. Dit suggereert dat de binding van metabolieten aan eiwitten en veranderingen in eiwitconcentraties twee complementaire routes zijn waarmee cellen eiwitactiviteit reguleren.
Eiwitten kunnen relatief snel worden geactiveerd of geïnactiveerd door een structurele verandering die wordt gemedieerd door metabolieten. "Dit soort structurele verandering kan sneller worden teruggedraaid, " legt Picotti uit. Vanuit het perspectief van de cel, dit is vaak logisch, omdat het gebruik van de route die een verandering in concentratie met zich meebrengt, betekent dat de cel eiwitten moet ontmantelen of opnieuw moet opbouwen, wat meer tijd kost, energie en middelen.
Picotti en haar collega's hebben ook kunnen aantonen dat veel enzymen minder kieskeurig zijn dan eerder werd gedacht:ze kunnen blijkbaar binden aan verschillende metabolieten en deze chemisch veranderen. Tot nu, er werd aangenomen dat enzymen meestal specifiek waren voor een paar zeer vergelijkbare moleculen.
De farmaceutische industrie is erg geïnteresseerd in de nieuwe aanpak, omdat het kan worden gebruikt om de interactie van geneesmiddelen met cellulaire eiwitten te testen en de doelen van een geneesmiddel te identificeren. Onderzoekers zouden kunnen onderzoeken aan welke eiwitten en aan welke plaatsen dit medicijn bindt, hoe het hun structuur verandert en dus hun activiteit beïnvloedt. Dit zou het testen en de ontwikkeling van nieuwe medicijnen vergemakkelijken en versnellen.
 Video:Wat is leven? En zullen we het op andere planeten vinden?
Video:Wat is leven? En zullen we het op andere planeten vinden? Oorzaak van kathodedegradatie geïdentificeerd voor nikkelrijke materialen
Oorzaak van kathodedegradatie geïdentificeerd voor nikkelrijke materialen Meer zien met PET-scans:verrassend nieuw mechanisme voor bevestiging van chemische tracers ontdekt
Meer zien met PET-scans:verrassend nieuw mechanisme voor bevestiging van chemische tracers ontdekt Nieuwe aanpak voor het oplossen van eiwitstructuren uit minuscule kristallen
Nieuwe aanpak voor het oplossen van eiwitstructuren uit minuscule kristallen Waterstofproducerend enzym beschermt zichzelf tegen zuurstof
Waterstofproducerend enzym beschermt zichzelf tegen zuurstof
Hoofdlijnen
- Invasieve superschurkenkrab kan door zijn kieuwen heen eten
- Kluizenaar of niet? Wetenschappers gebruiken Twitter om spidervragen aan te pakken
- Wat is kudde-immuniteit?
- Onderzoek toont aan dat plantengroeiregulatoren gunstig kunnen zijn voor de vestiging van uien, productie
- Grootschalige aanpak onthult imperfecte actor in plantenbiotechnologie
- Een geprioriteerde lijst van invasieve uitheemse soorten die het milieu van het VK bedreigen
- Cat Chromosome Information
- Wat is de Chromatins-functie?
- Plankton zwemt tegen de stroom in
- Uit welke elementen bestaat Zuiveringszout?

- Universeel regulatiemechanisme in plantencellen ontdekt
- De houdbaarheid van voedsel verlengen met nanomaterialen

- Nieuw onderzoek biedt uitgebreide inzichten in de reactie van de hersenen op opioïden

- Efficiënte depolymerisatie in vaste toestand van PET-afval

 Verbetering van de weersvoorspelling met een nieuwe IASI-kanaalselectiemethode
Verbetering van de weersvoorspelling met een nieuwe IASI-kanaalselectiemethode Dubbel röntgenzicht helpt bij onderzoek naar tuberculose en osteoporose
Dubbel röntgenzicht helpt bij onderzoek naar tuberculose en osteoporose Membraan geïnspireerd op bot en kraakbeen produceert efficiënt elektriciteit uit zout water
Membraan geïnspireerd op bot en kraakbeen produceert efficiënt elektriciteit uit zout water AI dringt verder door in het dagelijks leven op IFA in Berlijn:analisten
AI dringt verder door in het dagelijks leven op IFA in Berlijn:analisten Retailers kijken naar nieuwe technologie, gegevens om fortuinen te doen herleven
Retailers kijken naar nieuwe technologie, gegevens om fortuinen te doen herleven Wat is de definitie van hydraulische lift?
Wat is de definitie van hydraulische lift?  BepiColombo is klaar voor zijn lange cruise
BepiColombo is klaar voor zijn lange cruise Nieuwe lithografietechniek maakt de productie van nanoschaalpatronen van titania mogelijk voor hightech-toepassingen
Nieuwe lithografietechniek maakt de productie van nanoschaalpatronen van titania mogelijk voor hightech-toepassingen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

