
Wetenschap
Nieuwe studie onderzoekt de passage van geknoopt DNA door nanoporiën
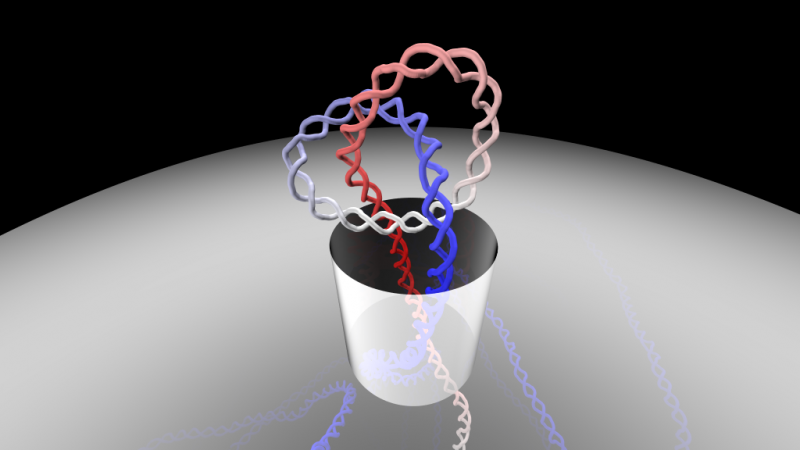
Hoe kunnen lange DNA-filamenten, die een ingewikkelde en zeer geknoopte structuur hebben, erin slagen om door de kleine poriën van verschillende biologische systemen te gaan? Dit is de fascinerende vraag die Antonio Suma en Cristian Micheletti aan de orde stellen, onderzoekers van de International School for Advanced Studies (SISSA) in Triëst die computersimulaties gebruikten om de mogelijkheden van het genetisch materiaal in dergelijke situaties te onderzoeken. De studie is zojuist gepubliceerd in PNAS , het tijdschrift van de National Academy of Sciences van de Verenigde Staten. Krediet:Antonio Suma, SISSA
Iedereen die op een zeilboot heeft gezeten, weet dat een knoop leggen de beste manier is om een touw aan een haak te bevestigen en wegglijden te voorkomen. evenzo, knopen in naaigaren voorkomen dat ze door twee stukken stof glippen. Hoe, dan, kan lange DNA-filamenten, die een ingewikkelde en zeer geknoopte structuur hebben, erin slagen om door de kleine poriën van verschillende biologische systemen te gaan? Dit is de fascinerende vraag die Antonio Suma en Cristian Micheletti aan de orde stellen, onderzoekers van de International School for Advanced Studies (SISSA) in Triëst, die computersimulaties gebruikten om de dynamiek van het molecuul in dergelijke situaties te onderzoeken. De studie is zojuist gepubliceerd in PNAS , het tijdschrift van de National Academy of Sciences van de Verenigde Staten.
"Onze computationele studie werpt licht op de nieuwste experimentele doorbraken op het gebied van geknoopte DNA-manipulatie, en voegt interessante en onverwachte elementen toe, " legt Micheletti uit. "We hebben voor het eerst waargenomen hoe geknoopte DNA-filamenten door minuscule poriën met een diameter van ongeveer 10 nanometer (10 miljardste van een meter) gaan. Het gedrag dat in onze simulaties werd waargenomen, kwam goed overeen met de experimentele metingen verkregen door een internationaal onderzoeksteam onder leiding van Cees Dekker, die slechts een paar maanden geleden werden gepubliceerd in Natuur Biotechnologie . Deze geavanceerde en geavanceerde experimenten markeerden een keerpunt voor het begrijpen van DNA-knopen. Echter, huidige experimenten kunnen niet detecteren hoe DNA-knopen daadwerkelijk door de nauwe porie gaan.
"In feite, het fenomeen doet zich voor op een kleine ruimtelijke schaal die ontoegankelijk is voor microscopen. Dit is de reden waarom onze groep zijn toevlucht nam tot wat de grote Duitse biofysicus Klaus Schulten 'de computationele microscoop, ' dat is, computersimulaties."
Suma en Micheletti leggen uit:"De simulaties onthulden dat het passeren van de knoop op twee verschillende manieren kan plaatsvinden:een waarbij de knoop strak zit, en de andere waar de knoop meer gedelokaliseerd is. In beide gevallen, de knoop gaat niet alleen door de porie, maar het doet dat in een zeer korte tijd."
Bovendien, de knoop gaat meestal over in de laatste stadia van de translocatie, wanneer het grootste deel van de DNA-streng al is gepasseerd. "Maar er is meer dat contra-intuïtief is, zeggen de auteurs. "De grootte van de knoop, klein of groot, lijkt de porie-obstructietijd niet veel te beïnvloeden. Dit laatste hangt in plaats daarvan af van de translocatiesnelheid, die, beurtelings, hangt af van de beginpositie van de knoop langs de gloeidraad." Deze resultaten, zeggen de onderzoekers, zou moeten helpen bij het ontwerpen van toekomstige experimenten die de spontane knoop van DNA onderzoeken, een nog grotendeels onontgonnen locatie, vooral met betrekking tot de grootte van DNA-knopen.
Het bevorderen van ons huidige begrip van knopen in biologische moleculen is belangrijk om hun implicaties in zowel biologische als toepassingscontexten te verduidelijken, zoals DNA-sequencing met behulp van nanoporiën. Suma en Micheletti hopen dat de veelbelovende richtingen die door hun studie worden gesuggereerd, kunnen leiden tot een meer gedetailleerde en nauwkeurige profilering van verstrengeling in DNA, RNA en eiwitten.
 UTA breidt inspanningen uit om technologieën voor waterrecycling te ontwikkelen
UTA breidt inspanningen uit om technologieën voor waterrecycling te ontwikkelen Levenscyclus van luiaards
Levenscyclus van luiaards  Wetenschappers verifiëren de theorie van de rol van de Stille Zuidzee in natuurlijke atmosferische CO2-fluctuaties
Wetenschappers verifiëren de theorie van de rol van de Stille Zuidzee in natuurlijke atmosferische CO2-fluctuaties Everest-redders halen lichamen van twee Indiase klimmers op
Everest-redders halen lichamen van twee Indiase klimmers op Studie:Noordwest hittegolf onmogelijk zonder klimaatverandering
Studie:Noordwest hittegolf onmogelijk zonder klimaatverandering
Hoofdlijnen
- Luipaarden doden minstens 3 kinderen op het Indiase platteland
- Nieuwe functie in genregulerend eiwit ontdekt
- Hoe een iconische foto van een appel de inspiratie vormde voor een verbeterde celanalyse
- Maatregelen voor welzijn en duurzaamheid heroverwegen van lokale naar mondiale schaal
- Hoe een 3D-model van een plantencel te maken
- Wat zijn de vier organische moleculen die in levende wezens worden gevonden?
- Waarom is chemie belangrijk voor de studie van anatomie en fysiologie?
- Onderzoek naar maïsgenetica onthult het mechanisme achter het stil worden van eigenschappen
- Onderbroken herprogrammering zet volwassen cellen om in hoge opbrengsten van voorloperachtige cellen
- Opheldering van nanostructuren in praktische heterogene katalysatoren
- Net als Wolverine, mensen hebben metaal nodig om sterke botten te behouden

- Van kantoorramen tot Mars:wetenschappers debuteren superisolerende gel

- Wetenschappers ontwikkelen zeer gevoelige moleculaire optische druksensor

- Modulerende foto- en elektroluminescentie in een op stimuli reagerende moleculaire kleurstof

 Hydrogeneringsregeling van nitrobenzeen in elektrokatalytische processen gerealiseerd
Hydrogeneringsregeling van nitrobenzeen in elektrokatalytische processen gerealiseerd Spiraalachtige patronen van stervorming ontdekt in oude sterrenstelsels
Spiraalachtige patronen van stervorming ontdekt in oude sterrenstelsels Afbeelding:Marsrover-revival
Afbeelding:Marsrover-revival Natuurkundigen houden toezicht op de vorming van hogere mangaansilicidefilms
Natuurkundigen houden toezicht op de vorming van hogere mangaansilicidefilms NASA-satelliet vindt sterke stormen rond het centrum van Lekimas
NASA-satelliet vindt sterke stormen rond het centrum van Lekimas Ontleding van H2O
Ontleding van H2O  Hoe gewogen waarschijnlijkheden te berekenen
Hoe gewogen waarschijnlijkheden te berekenen Een mogelijke verklaring voor de mysterieuze ijscirkels in het Baikalmeer
Een mogelijke verklaring voor de mysterieuze ijscirkels in het Baikalmeer
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

