
Wetenschap
Vooruitgang in het rijstgenoomonderzoek biedt inzichten en veelbelovende toepassingen voor de landbouw
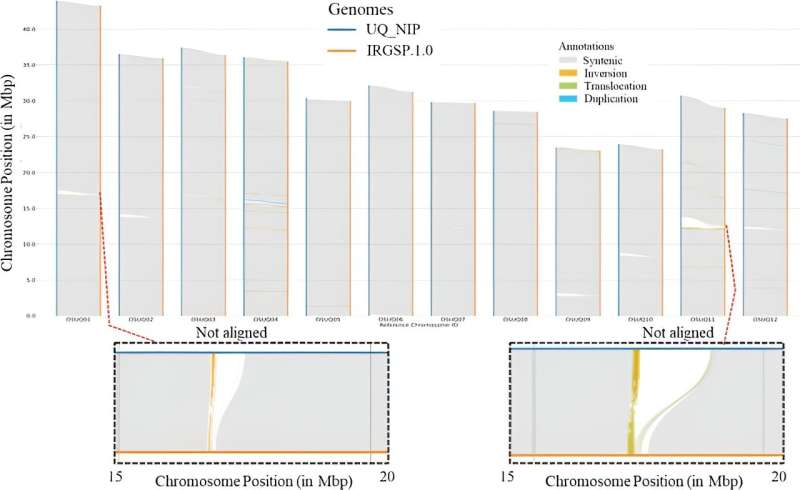
De japonica-rijstcultivar Nipponbare is van cruciaal belang geweest als referentie in de rijstgenomica sinds de eerste sequencing meer dan twintig jaar geleden, wat een belangrijke doorbraak betekende in de plantengenomica. Ondanks voortdurende verbeteringen in de sequentietechnologie bevat de Nipponbare-genoomassemblage nog steeds onopgeloste hiaten, voornamelijk als gevolg van repetitieve DNA-sequenties.
Aanhoudende inspanningen en technologische vooruitgang hebben de genoomassemblage bij andere rijstsoorten verbeterd en uitgebreid tot telomeersequencing. Het bereiken van een volledig haplotype-opgeloste assemblage blijft echter een onopgelost probleem in het rijstgenomisch onderzoek, wat een cruciaal gebied vormt voor toekomstig onderzoek.
Een onderzoek gepubliceerd in Tropical Plants genereert op 3 april 2024 een verbeterd haplotype-opgelost rijstgenoom voor een uitgebreide telomeer-tot-telomeer (T2T) verbetering.
In deze studie werden PacBio HiFi-lezingen en Hi-C-lezingen gebruikt om een contig-assemblage met Hifiasm te genereren, resulterend in een haplotype gefaseerde assemblage. Dit assemblageproces leverde verschillende contigs op voor negen van de chromosomen. Daarentegen omvatte de assemblage van de resterende drie chromosomen voor elk twee afzonderlijke contigs.
De geassembleerde contigs werden vervolgens hiërarchisch georganiseerd in 12 pseudo-chromosomen met behulp van het YaHS-steigertool, culminerend in een T2T-assemblage die groter en completer was dan de vorige IRGSP-1.0-referentie. Deze verfijnde samenstelling onthulde de aanwezigheid van 3.050 nieuwe genen, waarvan meer dan 95% ondersteund werd door transcriptiebewijs, wat wijst op een significante verbetering in de annotatie en het structurele begrip van het genoom.
De bevindingen benadrukken het enorme potentieel van nieuwe sequencingtechnologieën om genomische gegevens uit te breiden en te verfijnen, waardoor de genetische informatie van gevestigde genomen aanzienlijk wordt verbeterd. Het uitgebreide en gedetailleerdere genoom, dat 99,3% van de universele genen met één kopie en een N50 van 30,7 Mb omvat, biedt een robuust raamwerk voor verdere genetische studies en fokprogramma's voor rijst.
De vergelijkende analyse bracht ook structurele varianten en aanvullende niet-uitgelijnde regio's aan het licht, waardoor het begrip van genomische architectuur en functionaliteit werd verrijkt.
Volgens de hoofdonderzoeker van het onderzoek, Robert J. Henry, "zal dit gefaseerde genoom een nuttige hulpbron zijn voor rijstonderzoek." Vooruitkijkend richt dit team zich op het toepassen van hun geavanceerde sequencing- en assemblagetechnieken op andere rijstvariëteiten en nauw verwante soorten.
Samenvattend onderstreept dit werk niet alleen de snelle evolutie van de rijstgenomicatechnologie, maar benadrukt het ook de cruciale behoefte aan voortdurende vooruitgang om complexe genomen nauwkeurig in kaart te brengen, waardoor aanzienlijke vooruitgang op het gebied van de landbouwgenomica mogelijk wordt gemaakt.