
Wetenschap
CMU-software assembleert RNA-transcripten nauwkeuriger
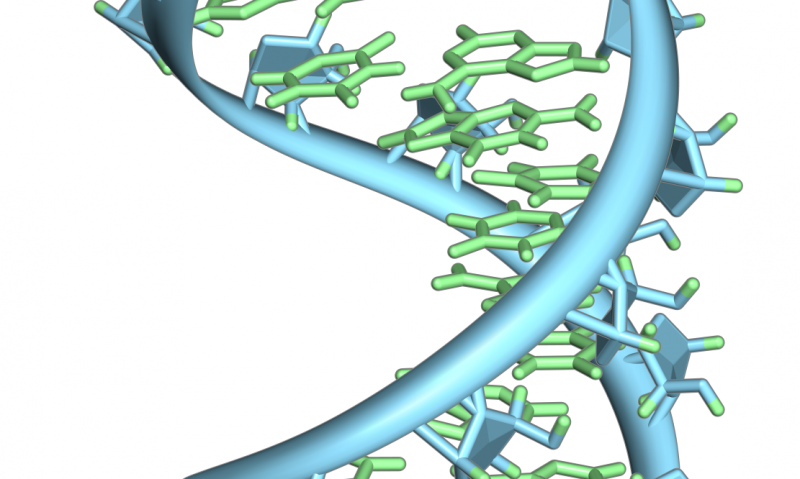
Een haarspeldlus van een pre-mRNA. Gemarkeerd zijn de nucleobasen (groen) en de ribose-fosfaatruggengraat (blauw). Merk op dat dit een enkele streng RNA is die zich op zichzelf terugvouwt. Krediet:Vossman/Wikipedia
Computerbiologen van de Carnegie Mellon University hebben een nauwkeuriger rekenmethode ontwikkeld voor het reconstrueren van de volledige nucleotidesequenties van de RNA-producten in cellen, transcripties genoemd, die informatie van een gen omzetten in eiwitten of andere genproducten.
Hun software, genaamd Sint-jakobsschelp, zal wetenschappers helpen bij het bouwen van een completere bibliotheek van RNA-transcripten en zo wetenschappers helpen de regulatie van genexpressie beter te begrijpen.
Een verslag over Scallop door Carl Kingsford, universitair hoofddocent computationele biologie, en Mingfu Shao, Lane Fellow in de afdeling Computational Biology van de School of Computer Science, wordt vandaag online gepubliceerd door het tijdschrift Natuur Biotechnologie .
Scallop is een zogenaamde transcript assembler, het nemen van fragmenten van RNA-sequenties, genaamd leest, die worden geproduceerd door high-throughput RNA-sequencing-technologieën (RNA-seq), en ze weer in elkaar te zetten, als stukjes van een puzzel, om volledige RNA-transcripten te reconstrueren.
"Er zijn veel bestaande monteurs, "Sho zei, "maar deze bestaande methoden zijn nog steeds niet nauwkeurig genoeg."
In vergelijking met twee toonaangevende assembleurs, StringTie en TransComb, Scallop is 34,5 procent en 36,3 procent nauwkeuriger voor transcripten die bestaan uit meerdere exons - subeenheden van een gen die coderen voor een deel van het genproduct.
Net als andere op referenties gebaseerde assemblers, Scallop begint met het construeren van een grafiek om uitlezingen te organiseren die zijn toegewezen aan de overeenkomstige locaties op het DNA van het gen. Er zijn veel alternatieve paden om de reads met elkaar te verbinden, echter, dus fouten zijn snel gemaakt. Scallop verbetert zijn kansen door een nieuw algoritme te gebruiken om volledig te profiteren van de informatie van reads die verschillende exons overspannen om het naar de juiste assemblagepaden te leiden.
Scallop blijkt bijzonder bedreven bij het samenstellen van minder overvloedige RNA-transcripten, verbetering van de nauwkeurigheid van StringTie en TransComb met 67,5 procent en 52,3 procent.
De onderzoekers hebben Scallop al als open software op de GitHub-repository uitgebracht.
"We hebben al meer dan 100 downloads gehad en, op basis van de feedback die we hebben ontvangen, mensen gebruiken het echt " zei Shao. "We verwachten meer gebruikers nu onze krant uit is."
 Genen ontsluiten aanwijzingen voor de evolutie en overleving van het Great Barrier Reef
Genen ontsluiten aanwijzingen voor de evolutie en overleving van het Great Barrier Reef Hoe worden adsorbentia gebruikt voor het opruimen van het milieu?
Hoe worden adsorbentia gebruikt voor het opruimen van het milieu?  Welk type bonenzaden te gebruiken voor een wetenschappelijk experiment
Welk type bonenzaden te gebruiken voor een wetenschappelijk experiment NASA vangt de verspreide overblijfselen van de voormalige tropische storm Tara
NASA vangt de verspreide overblijfselen van de voormalige tropische storm Tara Ondanks EU-boetes, Griekenland worstelt om recycling te promoten
Ondanks EU-boetes, Griekenland worstelt om recycling te promoten
Hoofdlijnen
- Wat is de formule voor cellulaire ademhaling?
- Lijst van eencellige organismen
- Ecosysteem: definitie, types, structuur en voorbeelden
- Disruptieve bio-engineering - verandert de manier waarop cellen met elkaar omgaan
- Wat doet de temporale kwab?
- Welke koninkrijken zijn heterotroof en autotroof?
- Celmembraan Feiten
- Wetenschappers vinden antarctische microben die alleen van lucht leven
- Ethics of Genetic Engineering
- Darwins afschuwelijke mysterie oplossen:hoe bloeiende planten de wereld veroverden

- Relaties tussen mitose in eukaryote cellen en binaire fissie in Prokaryoten
- Wetenschappers ontcijferen het genoom van herfstlegerworm, mottenplaag die Afrika binnendringt

- Nieuwe studie voorspelt wereldwijde verandering in ondiepe rifecosystemen als het water warmer wordt

- Biomassa versus energiepiramides

 Afbeelding:Slimme deeltjes
Afbeelding:Slimme deeltjes Gezichtsherkenning verboden:wat is de volgende stap in Oakland, bij Amazon en meer
Gezichtsherkenning verboden:wat is de volgende stap in Oakland, bij Amazon en meer Helder en efficiënt licht zonder zeldzame metalen
Helder en efficiënt licht zonder zeldzame metalen Zelfherstellend membraan voor papieren documenten voor brandstofceltoepassingen
Zelfherstellend membraan voor papieren documenten voor brandstofceltoepassingen Een sprankelende nieuwe manier om de magnetische velden van deeltjes op nanometerschaal te detecteren
Een sprankelende nieuwe manier om de magnetische velden van deeltjes op nanometerschaal te detecteren Dieren inheems in de staat North Carolina
Dieren inheems in de staat North Carolina  Bewijs gevonden voor middelgroot zwart gat nabij het centrum van de Melkweg
Bewijs gevonden voor middelgroot zwart gat nabij het centrum van de Melkweg Een pentagon tekenen op grafiekpapier
Een pentagon tekenen op grafiekpapier
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

