Wetenschap
Behoefte aan snelheid maakt genoombewerking efficiënt, zo niet beter
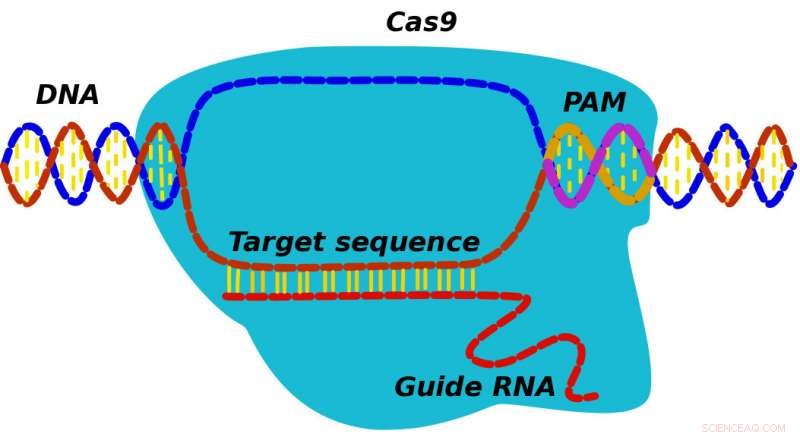
Wanneer een CRISPR-Cas9-eiwit zijn doelwit vindt, het vindt eerst een spacer-sequentie die bekend staat als PAM en zoekt vervolgens aangrenzend DNA om te zien of het overeenkomt met Cas9's gids-RNA. Een nieuw model ontwikkeld door onderzoekers van Rice University zou kunnen helpen bij het blootleggen van details over het mechanisme waarmee CRISPR-Cas9 mutaties kan vervangen door correct DNA. Krediet:Alexey Shvets/Rice University
Rice University-onderzoekers hebben een computermodel ontwikkeld om het mechanisme te kwantificeren waarmee CRISPR-Cas9-eiwitten hun genoombewerkingsdoelen vinden.
Anatoly Kolomeisky, een Rice hoogleraar scheikunde en chemische en biomoleculaire engineering, en alumnus Alexey Shvets paste een systeem aan dat ze eerder ontwikkelden om te laten zien hoe eiwitten hun biologische doelwitten vinden. Ze hopen dat het herziene model de resterende mysteries van CRISPR zal helpen ontrafelen.
In zijn natuurlijke staat, CRISPR, wat staat voor "geclusterde regelmatig interspaced korte palindroomherhalingen, " is het biologische mechanisme waarmee bacteriën zichzelf beschermen tegen virale infecties. De bacteriën nemen een kopie van het vreemde DNA op en bouwen een record op van al degenen die binnenvallen. Ze verwijzen naar dat record wanneer nieuwe indringers worden gedetecteerd en gebruiken het om ze te vernietigen.
In recente jaren, onderzoekers zijn begonnen het mechanisme aan te passen voor gebruik bij genoombewerking, die het potentieel heeft om ziekten te genezen en organismen te verbeteren, inclusief mensen. Maar een struikelblok was het risico dat CRISPR-Cas9-eiwitten, een van de systemen die gebruik maken van de CRISPR-aanpak, zal de verkeerde doelsequenties knippen en vervangen, mutaties introduceren.
Het Rice-model beschreven in de Biofysisch tijdschrift vond het waarschijnlijk dat CRISPR-Cas9 goede doelen efficiënter lokaliseert wanneer deze off-target bewerkingen mogen plaatsvinden, omdat de eiwitten geen tijd verspillen met het loskoppelen van off-targets om door te gaan met zoeken.
Dat kan wel of niet goed zijn, maar het is zeker het bestuderen waard, zei Kolomeisky.
"Het foutenpercentage (off-target snijden) is soms 10-20 procent, " zei hij. "We hebben hier twee ideeën over:de ene is dat virussen zeer snel muteren en dat bacteriën misschien doelen proberen te snijden die slechts licht gemuteerd zijn om flexibeler te zijn. De andere is dat er eiwitten zijn die fouten kunnen corrigeren, dus als er niet veel verkeerde sneden zijn, het systeem kan ze tolereren.
Kolomeisky zei dat zijn model een eenvoudige stap is in de richting van het uitzoeken van de dynamiek van CRISPR-bewerking. "CRISPR-Cas9 is de meest populaire variant omdat het maar één eiwit heeft en gemakkelijker is, biologisch, werken met, " hij zei.
Het Rice-lab ontwikkelde zijn oorspronkelijke model om te leren hoe eiwitten langs DNA glijden om doelen te vinden en processen zoals gentranscriptie te activeren. Kolomeisky merkte op dat CRISPR-pionier Jennifer Doudna ontdekte dat CRISPR-Cas9 niet op dezelfde manier zoekt. "Ze ontdekte dat het nergens op het DNA schuift, " hij zei.
In plaats daarvan, volgens Doudna en haar team, het eiwit herkent aanvankelijk drie-nucleotide PAM (voor protospacer aangrenzende motief) sequenties die de locatie van potentiële doelen markeren. "CRISPR vindt en bindt aan PAM en vervolgens onderzoekt het bijbehorende RNA het aangrenzende DNA om te zien of dit het doelwit is, ' zei Kolomeisky. 'Als dat zo is, het eiwit begint te snijden. Als niet, het distantieert zich en kijkt ergens anders."
In de daaropvolgende experimenten van Doudna met verwijderde PAM-sequenties, CRISPR-Cas9-eiwitten konden hun doelwit helemaal niet vinden. PAM's spelen dus een belangrijke rol en zijn niet alleen een generieke spacer, hij zei. "Zodra ik dit lees, Ik begreep dat we ons model hier ook konden gebruiken."
Het theoretische model kijkt naar first-passage-processen - processen die plaatsvinden wanneer een systeem een fysieke of chemische drempel overschrijdt, zoals het vinden van een relevante PAM - om CRISPR-Cas9-eiwitten te volgen die in een cel zijn ingebracht terwijl ze eerst PAM-sequenties onderzoeken en vervolgens, terwijl gebonden aan PAM's, zoek naar het DNA-doel dat overeenkomt met het RNA van de Cas9.
Ze ontdekten dat CRISPR's die off-target bezuinigingen voorkomen door zich te distantiëren van "verkeerd" DNA langer duren om zich te vestigen dan een die eenvoudig van doelen afsnijdt. "Naar de verkeerde PAM gaan kost tijd, "Zei Kolomeisky. "Onze berekening toont aan dat CRISPR echte doelen sneller kan vinden als het soms op de verkeerde plaatsen snijdt. De fractie die naar de juiste doelen gaat, kan kleiner zijn, maar je zult ze uiteindelijk snijden.
"Het is een eenvoudig model en precies oplosbaar, "Zei Kolomeisky. "Als iemand wil testen, het model kan specifieke voorspellingen doen en in sommige gevallen trends bieden voor wat moet worden waargenomen." Wat nog ontbreekt in het model is de mogelijkheid om te zien of de RNA-sleutel zijn doelwit tegelijkertijd herkent - in één keer aan het DNA bindt - of opeenvolgend, nucleotide voor nucleotide.
"Het meest indrukwekkende aan CRISPR is niet de ontdekking van een immuunsysteem in bacteriën, maar het feit dat dit een revolutie heeft veroorzaakt in de biotechnologie, omdat het betekent dat we in elke cel elk DNA op een specifieke locatie kunnen knippen, heel precies, " zei Kolomeisky. "Ik hoop dat ons werk meer fundamentele studies zal stimuleren, omdat ik de CRISPR-methode erg leuk vind. Maar ik ben niet blij als mensen het toepassen zonder te begrijpen hoe het op moleculair niveau werkt."
Shvets is nu een postdoctoraal onderzoeker aan het Massachusetts Institute of Technology. Kolomeisky is hoogleraar scheikunde en chemische en biomoleculaire engineering.
 Bij veldproeven, apparaat haalt water uit woestijnlucht
Bij veldproeven, apparaat haalt water uit woestijnlucht Nieuw protocol voor het meten van achtergrondniveaus van drugs in misdaadlabs
Nieuw protocol voor het meten van achtergrondniveaus van drugs in misdaadlabs Nieuwe methode voor synthese van moleculaire waterstof stelt maatstaf voor platinavrije elektrokatalysatoren
Nieuwe methode voor synthese van moleculaire waterstof stelt maatstaf voor platinavrije elektrokatalysatoren Onderzoekers krijgen een glimp op nanoschaal van spleet- en putcorrosie terwijl het gebeurt
Onderzoekers krijgen een glimp op nanoschaal van spleet- en putcorrosie terwijl het gebeurt Wat gebeurt er als SO2 reageert met staal?
Wat gebeurt er als SO2 reageert met staal?
 Geconfronteerd met hoge smogniveaus, Milaan gaat auto's zondag verbieden
Geconfronteerd met hoge smogniveaus, Milaan gaat auto's zondag verbieden Beyond EPA's Clean Power-beslissing:klimaatactievenster zou al in 2023 kunnen sluiten
Beyond EPA's Clean Power-beslissing:klimaatactievenster zou al in 2023 kunnen sluiten Spanje, Portugal zet zich schrap voor natuurbranddreiging terwijl de uitzendkrachten stijgen
Spanje, Portugal zet zich schrap voor natuurbranddreiging terwijl de uitzendkrachten stijgen Nile Delta Facts
Nile Delta Facts Onderzoekers meten wereldwijd stikstofgehalte in graslanden
Onderzoekers meten wereldwijd stikstofgehalte in graslanden
Hoofdlijnen
- Hoe robots de productiecrisis van antibiotica kunnen oplossen
- Wat zijn organellen in een prokaryotische cel?
- Vrouwelijke makaak in Italiaans heiligdom heeft haar gemummificeerde dode baby opgegeten
- Wolven bleken meer coöperatief te zijn met hun eigen soort dan honden met die van hen
- Hoe de droogrot Serpula lacrymans zich aanpasten aan een nieuwe ecologische habitat
- Microbeads zorgen ervoor dat ultrasone golven cellen veiliger kunnen stimuleren
- Inzicht in onze levensduur van meer dan 50 jaar die nog steeds in ontwikkeling is, genetische studie toont aan
- Crowdsourced spel is bedoeld om oplossingen te vinden voor aflatoxine
- Verschillen tussen Protista & Monera
 Aardbeving met een kracht van 6,7 op de schaal van Richter treft Filipijnen:USGS
Aardbeving met een kracht van 6,7 op de schaal van Richter treft Filipijnen:USGS Stedelijke groene ruimte brengt geluk als geld het niet meer kan kopen
Stedelijke groene ruimte brengt geluk als geld het niet meer kan kopen Negatief rentebeleid werkt averechts:nieuw onderzoek
Negatief rentebeleid werkt averechts:nieuw onderzoek Smelt plastic flessen voor Molding
Smelt plastic flessen voor Molding Meerdere 12-volt loodzuurbatterijen opladen
Meerdere 12-volt loodzuurbatterijen opladen Rapport:Facebook haalt 25 procent van de Amerikaanse digitale videoadvertentie-inkomsten binnen
Rapport:Facebook haalt 25 procent van de Amerikaanse digitale videoadvertentie-inkomsten binnen Nieuwe studie ondersteunt overleving van microben en organische verbindingen in de ruimte
Nieuwe studie ondersteunt overleving van microben en organische verbindingen in de ruimte Klimaatverandering veroorzaakt veranderingen in mariene fanerogame populaties
Klimaatverandering veroorzaakt veranderingen in mariene fanerogame populaties
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com