
Wetenschap
Het opzetten van een genoombrede kaart van bacteriële genen die cruciaal zijn voor de kolonisatie van planten door nuttige microben
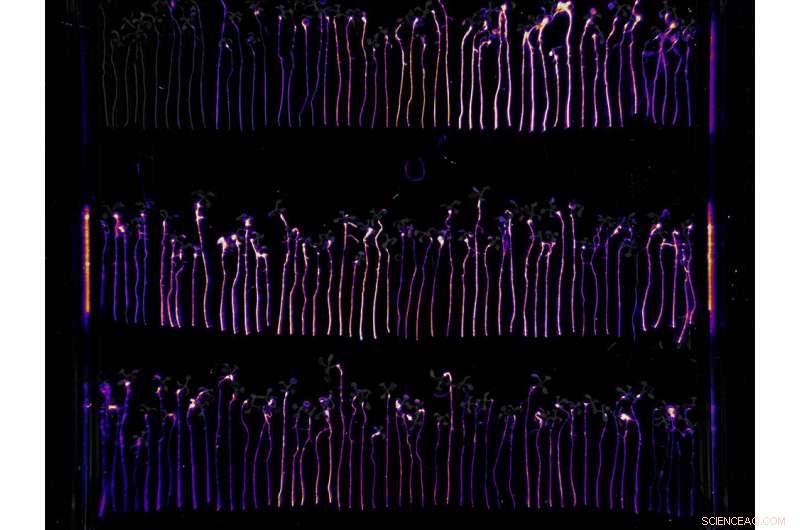
Een bioluminescente test hielp onderzoekers het kolonisatievermogen van P. simiae-mutante stammen visueel te kwantificeren die werden geïdentificeerd door het RB-TnSeq-scherm. Krediet:Benjamin Cole
Werken met de plantengroeibevorderende bacterie Pseudomonas simiae, onderzoekers hebben 115 genen geïdentificeerd die een negatief effect hebben op het vermogen om een plantenwortelsysteem te koloniseren wanneer ze gemuteerd zijn.
De gezondheid en ontwikkeling van een plant wordt beïnvloed door de complexe gemeenschap van microben eromheen. Door de bacteriële genen te identificeren die kunnen veranderen hoe goed microben een plant kunnen koloniseren, onderzoekers kunnen gerichte benaderingen ontwikkelen om de gezondheid en groei van planten te verbeteren voor een aantal toepassingen, inclusief een hogere biomassaopbrengst voor de productie van biobrandstoffen.
De gezondheid en ontwikkeling van een plant wordt beïnvloed door microben die zich in de plant bevinden (endofyten), in de grond, en in het smalle gebied waar de plantenwortels interageren met de bodem (rhizosfeer). Om beter te begrijpen hoe microben het wortelmilieu koloniseren, onderzoekers van het Joint Genome Institute, een DOE Office of Science gebruikersfaciliteit, en hun medewerkers aan het Howard Hughes Medical Institute aan de Universiteit van North Carolina, een genoombrede transposon-mutagenesebenadering toegepast op de modelplantengroeibevorderende bacterie Pseudomonas simiae met behulp van de modelplant Arabidopsis thaliana als gastheer om een genoombrede kaart te genereren van bacteriële genen die de werkzaamheid van microbiële kolonisatie beïnvloeden.
Door middel van willekeurig gebarcodeerde transposon-sequencing (RB-TnSeq), het team identificeerde 115 genen die, wanneer gemuteerd, hebben verminderde wortel kolonisatie mogelijkheden. Deze genen zijn betrokken bij functies zoals suikermetabolisme, celwandsynthese, en beweeglijkheid. Het team identificeerde ook 243 genen die, wanneer gemuteerd, de mogelijkheden voor wortelkolonisatie positief veranderen, velen van hen zijn waarschijnlijk betrokken bij aminozuurtransport en metabolisme. Aanvullend, het team identificeerde 43 genen waaraan zeer weinig of geen functionele informatie kon worden toegewezen. De onderzoekers suggereerden dat deze genen mogelijk nieuwe functies of routes vertegenwoordigen die nog moeten worden gekarakteriseerd. Het werk toont aan dat RB-TnSeq kan worden toegepast om in vivo kolonisatie van bacteriële plantenwortels te beoordelen.
Een van de medewerkers aan dit project was Sabah Ul-Hasan, een stagiaire uit 2015 via de DOE JGI/University of California, Merced Genomics Distinguished Graduate Stageprogramma. Het programma biedt afgestudeerde studenten van UC Merced praktische ervaring in geavanceerd genoomonderzoek als onderdeel van de toewijding van de DOE JGI om de volgende generatie wetenschappelijk talent op te leiden.
Een van de belangrijkste uitdagingen die voortkomen uit snelle sequencing is de toewijzing van functies aan nieuwe genen. De RB-TnSeq-aanpak die hier wordt gebruikt, kan de associatie van nieuwe genen versnellen met kenmerken en gedragingen die van belang zijn voor DOE-missies, zoals inzicht in hoe microben de groei helpen (of belemmeren) van gewassen die kunnen dienen als bio-energiegrondstoffen. Om bij de fundamentele genetische bijdragen te komen, zoals de 115 genen die de interacties tussen microben en planten negatief reguleren, zal helpen om toekomstige inspanningen te concentreren om dit onderzoek vooruit te helpen.
 Mariene ecosystemen zijn bestand tegen klimaatverstoring
Mariene ecosystemen zijn bestand tegen klimaatverstoring 3 eenvoudige wetenschapshacks aan het einde van de zomer die je nu meteen moet proberen
3 eenvoudige wetenschapshacks aan het einde van de zomer die je nu meteen moet proberen De positie en baan van de aarde leidden tot het uitsterven van het oude zeeleven
De positie en baan van de aarde leidden tot het uitsterven van het oude zeeleven Cyanidevrij goud gaat in productie
Cyanidevrij goud gaat in productie 21 doden door hevige regenval in centraal China
21 doden door hevige regenval in centraal China
Hoofdlijnen
- Gestresste stokstaartjes helpen minder snel groep
- Hoe zijn cellen, weefsels en organen aan elkaar gerelateerd?
- Hoe Cytoplasma voor een celproject te maken
- Kwallen hebben superkrachten - en andere redenen waarom ze hun slechte reputatie niet verdienen
- Wetenschappers ontdekken dat de meeste blauwe vinvissen rechtshandig zijn, behalve wanneer ze omhoog zwemmen
- De wetenschap ontdekt waarom sommigen van ASMR-video's houden en anderen ze haten
- De rol van microfilamenten in cytokinese
- Wat onderscheidt mensen van chimpansees en andere apen?
- Dode walvis verrast zwemmers op iconisch strand van Rio
- Gene drives hebben het potentieel om muggenpopulaties te onderdrukken, maar resistente muggen duiken op

- Iteratieve evolutie:is de Aldabra-rail twee keer geëvolueerd?

- Onderzoekers vinden eerste wilde alligator brekende schildpad in Illinois sinds 1984

- Neanderthalers waren eigenlijk prehistorische Picasso's
- Hoe lepra werkt

 GPD converteren naar MGD
GPD converteren naar MGD Schade door overstromingen hoeft niet onvermijdelijk te zijn
Schade door overstromingen hoeft niet onvermijdelijk te zijn Succesvolle waarnemingen van de dynamiek van één molecuul met lage röntgendoses
Succesvolle waarnemingen van de dynamiek van één molecuul met lage röntgendoses Milieuvriendelijke vlamvertrager kan worden afgebroken tot minder veilige verbindingen
Milieuvriendelijke vlamvertrager kan worden afgebroken tot minder veilige verbindingen Hoe de stroomsterkte te berekenen
Hoe de stroomsterkte te berekenen Komeet of asteroïde:wat heeft de dinosaurussen gedood en waar kwam het vandaan?
Komeet of asteroïde:wat heeft de dinosaurussen gedood en waar kwam het vandaan? Vereenvoudiging van de diagnose van huidziekten met actuele nanotechnologie
Vereenvoudiging van de diagnose van huidziekten met actuele nanotechnologie Over de dennenboom Caterpillar
Over de dennenboom Caterpillar
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | German | Dutch | Danish | Norway | Portuguese | Swedish |

-
Wetenschap © https://nl.scienceaq.com

