
Wetenschap
Doolhoven oplossen met DNA-navigators met één molecuul
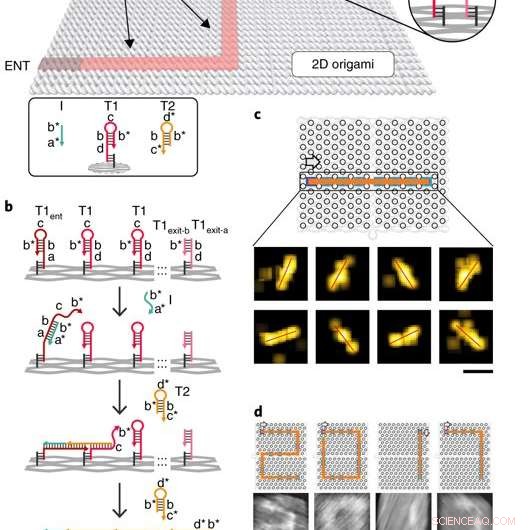
Implementatie van de single-molecule DNA-navigator. a) Schematische weergave van het PSEC-systeem. Een acyclische verbonden grafiek (d.w.z. een boom) wordt geconstrueerd op een rechthoekig DNA-origami-substraat van 100 x 70 nm ^ 2. T1 en T2 zijn twee soorten brandstoffen die de PSEC op de boom drijven. Initiator I werd gebruikt om de initiatie van de PSEC te activeren vanaf het ingangspunt KNO. b) voortplantingsmechanisme van het PSEC-systeem. Initiator I herkent en opent de T1ent-haarspeld bij vertex ENT. De geopende haarspeld vangt en opent vervolgens een T2-haarspeld uit de omgeving om de cascade te starten. c) Een rechte lijn geplaveid door PSEC en gevisualiseerd met DNA-PAINT. d) Vier verharde cijfers van "2017" afgebeeld met behulp van atomaire krachtmicroscopie (AFM). De T1-haarspelden op de hoekpunten zijn aangepast om ongewenste overspanningen te voorkomen. De pijlen geven de voortplantingsrichting aan. Credit: Natuurmaterialen , doi:10.1038/s41563-018-0205-3.
Het gebied van intelligente nanorobotica is gebaseerd op de grote belofte van moleculaire apparaten met informatieverwerkingsmogelijkheden. In een nieuwe studie die de trend van op DNA gebaseerde informatiedragers ondersteunt, wetenschappers hebben een DNA-navigatiesysteem ontwikkeld dat enkelvoudige moleculen kan uitvoeren, parallel, diepte-eerst zoekacties op een tweedimensionaal origami-platform.
Pathfinding-operaties met DNA-navigators maken gebruik van een gelokaliseerd cascadeproces voor strenguitwisseling dat wordt gestart op een unieke triggerlocatie op het origami-platform. Automatische voortgang langs paden wordt mogelijk gemaakt door DNA-haarspelden die een universele traversale reeks bevatten. Met opzet, elke navigator met één molecuul kan autonoom elk van de mogelijke paden verkennen door een boom met 10 hoekpunten die in het onderzoek is geconstrueerd. De doolhoven waren gelijk aan een boom met een ingang aan de wortel en een uitgang door een van de bladeren. De studie uitgevoerd door Jie Chao en collega's resulteerde in het verkennen van alle paden die door de DNA-navigators werden genomen om een specifiek oplossingspad te extraheren dat een bepaald paar begin- en eindpunten in het doolhof met elkaar verbond. Als resultaat, het oplossingspad werd duidelijk op het origami-platform gelegd en geïllustreerd met behulp van beeldvorming met één molecuul. De aanpak is nu gepubliceerd in Natuurmaterialen , detaillering van de realisatie van moleculaire materialen met ingebedde biomoleculaire computerfuncties om te werken op het niveau van het enkele molecuul met het potentieel om intelligente nanorobots te ontwikkelen voor toekomstige toepassingen in de industrie en de geneeskunde.
In het verleden werden geavanceerde moleculaire gereedschappen gebruikt om moleculaire machines te maken die chemische, fotonische of elektrische energie omzetten in roterende of lineaire bewegingen op nanoschaal. Bijvoorbeeld, Brownse beweging op nanoschaal kan gecontroleerd worden omgezet in gerichte bewegingen binnen op DNA gebaseerde nanomachines met behulp van DNA-hybridisatiereacties. Dergelijke op DNA gebaseerde machines werken autonoom door een ingebed 'moleculair programma' te volgen dat vooraf is ontworpen als een cascadereactie die handmatig wordt geactiveerd via een externe stimulus voor elke stap van de operatie.
De focus van het veld is geleidelijk verschoven naar het actualiseren van op DNA gebaseerde logische circuits met behulp van aptameren en DNAzymen om moleculaire logische poorten te ontwerpen. Bijvoorbeeld, in 2006, Stojanovic en collega's integreerden meer dan 100 logische DNA-poorten om een automatisering genaamd MAYA-II te ontwikkelen om een spelletje Tic-Tac-Toe te spelen. Eerdere studies toonden een enzymvrij computersysteem aan op basis van hybridisatiekettingreacties (HCR) om logische poorten en logische circuits te creëren voor robuustere en efficiëntere prestaties dan de originele systemen.
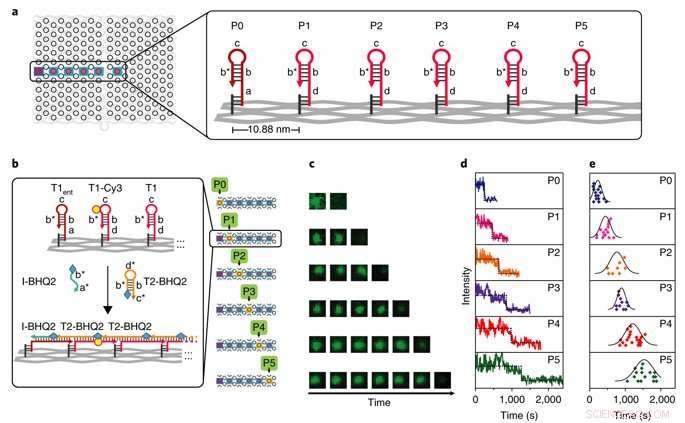
Karakterisering van één molecuul van PSEC-kinetiek. a) Een rechte lijn in het midden van de DNA-origami werd gebruikt als testbed. Vertex P0 is het startpunt en hoekpunten P1-P5 zijn tussenliggende punten. b) Details van het ontwerp van zes parallelle tests om de kinetiek te meten met tijdsopgeloste TIRF. T2 gelabeld met BHQ2 werd gebruikt om T1 gelabeld met Cy3 te doven, het samenstellen van een kinetisch profiel bij elke stap. Het geïllustreerde voorbeeld toont het mechanisme van blussen. c) Voorbeeld TIRF-afbeeldingen laten zien dat de fluorescentie met de tijd verandert over de zes parallelle tests van P0 naar P5. d) Typische fluorescentiesporen van één molecuul die worden gebruikt om uitdovingsgebeurtenissen te volgen die optreden op hoekpunten P0 tot P5. e) Scatterplots met statistische analyse van de lengteverdeling per pad. Credit: Natuurmaterialen , doi:10.1038/s41563-018-0205-3.
In de huidige studie van Chao et al, hetzelfde basisprincipe van het HCR-reactieschema werd in een andere computationele context gebruikt om een DNA-navigatorsysteem met één molecuul te ontwikkelen. Het platform verkende alle mogelijke paden via een boomgrafiek ontworpen op een origami-structuur als een eenvoudig verbonden doolhof zonder cyclische paden. Dergelijke DNA-origamistructuren zijn van nature informatiedragende nanostructuren met een goed gedefinieerde geometrie op nanoschaal. Het doolhof zou kunnen worden onderzocht door proximale strenguitwisselingscascade (PSEC) op basis van hybridisatiekettingreacties. De onderzoekers toonden aan dat een systeem met een groot aantal DNA-navigators met één molecuul gezamenlijk parallelle diepte-eerst zoeken (PDFS) op de boom zou kunnen uitvoeren om het doolhof efficiënt op te lossen in 2D-origami. Aanvankelijk voerden de onderzoekers onderzoeken uit om het PSEC-ontwerp te testen.
Het proximale strenguitwisselingscascadesysteem (PSEC) (werkprincipe van de DNA-navigator) werd gefaciliteerd op een rechthoekig origami-substraat gemaakt van drie componenten, waaronder de fysieke implementatie van een boomgrafiek, volledige strengen en een initiatorstreng. Vacant areas without staple extensions corresponded to walls in the maze, preventing propagation of the strand exchange cascade. The entrance and exit were defined and denoted as ENT and EXIT respectively. In the second component, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
Met opzet, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
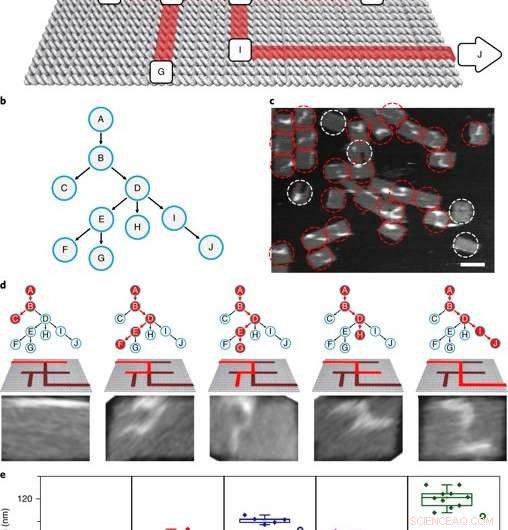
The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Credit: Natuurmaterialen , doi:10.1038/s41563-018-0205-3.
Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, NS, E) an intermediate vertex (I) and four dead ends (C, F, G, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
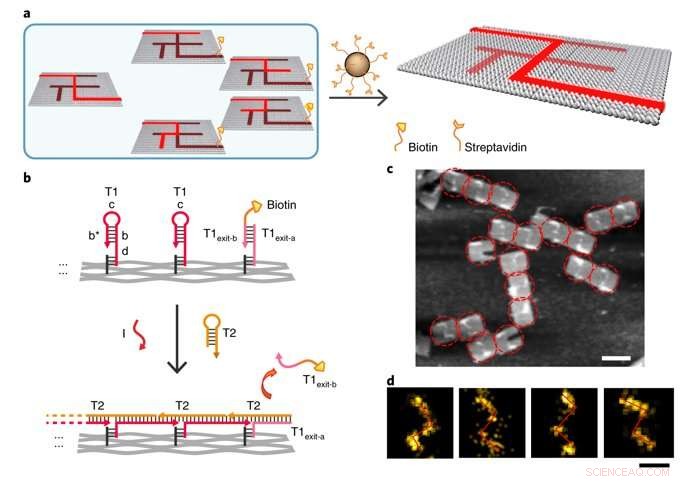
Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Credit: Natuurmaterialen , doi:10.1038/s41563-018-0205-3.
To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P ABDIJ ) was followed in the maze therefore. The computational context used in the study allowed exploration of paths through tree graphs defined on the origami. The autonomous path explored by the DNA navigators proceeded unidirectionally and irreversibly, turning at junctions and corners on the origami platform as they were designed to. The design enabled parallel depth first search (PDFS) allowing each DNA navigator to individually explore any one of the paths through the given graph at a defined speed, greater than that previously achieved.
The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Als resultaat, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades.
© 2018 Wetenschap X Netwerk
 Synthese van een bijna-infrarood lichtabsorberende macrocyclische aromatische verbinding
Synthese van een bijna-infrarood lichtabsorberende macrocyclische aromatische verbinding Neutronen geven inzicht in verbeterde prestaties voor hybride perovskiet-zonnecellen
Neutronen geven inzicht in verbeterde prestaties voor hybride perovskiet-zonnecellen Een wetenschapsproject over wat voor soort sap het reinigt Pennies Best
Een wetenschapsproject over wat voor soort sap het reinigt Pennies Best De grootste maan van Saturnus kan de sleutel zijn tot schonere motoren
De grootste maan van Saturnus kan de sleutel zijn tot schonere motoren De opkomst, val en heropleving van gentherapie
De opkomst, val en heropleving van gentherapie
 Hoe een mannelijke coyote van een vrouw te vertellen
Hoe een mannelijke coyote van een vrouw te vertellen  Een nieuwe kijk op de bronnen en effecten van broeikasgassen in China
Een nieuwe kijk op de bronnen en effecten van broeikasgassen in China insecten, ziekte verwoest bomen in Zuid-Californië. Wie gaat ze stoppen?
insecten, ziekte verwoest bomen in Zuid-Californië. Wie gaat ze stoppen? Langs een rivier in Minnesota, ijs en snoekbaarzen signaleren een veranderend klimaat
Langs een rivier in Minnesota, ijs en snoekbaarzen signaleren een veranderend klimaat Verwachte afname van het zee-ijs in de winter in het Noordpoolgebied gekoppeld aan de Euraziatische circulatie
Verwachte afname van het zee-ijs in de winter in het Noordpoolgebied gekoppeld aan de Euraziatische circulatie
Hoofdlijnen
- Wat is het Genotype voor de Roan-kleur?
- Star Treks Kobayashi Maru Oefening verkent situaties zonder winstoogmerk
- DNA-tests op albatrospoep onthullen geheim dieet van toproofdier
- Ambien
- Studie vertelt over pompoenkleurige zombies
- Bacteriën krijgen resistentie van concurrenten
- Squash-variëteit waarvan ooit werd gedacht dat ze uitgestorven was, gedijt goed op biologische boerderij
- Overeenkomsten van de mitochondria en nucleus
- Vier belangrijke soorten chromosomen
- Onderzoekers gebruiken Raman-spectroscopie en STM om moleculen chemisch in kaart te brengen met een resolutie van 1 nm

- Wetenschappers ontwikkelen kluizen op nanoschaal om nanoschijven in te kapselen voor medicijnafgifte

- Kleurstofgevoelige zonnecel absorbeert een breed scala aan zichtbare en infrarode golflengten

- Koelmagneten met geluid

- Kameleon inspireert een slimme huid die van kleur verandert in de zon

 Optische eigenschappen van polyethyleen
Optische eigenschappen van polyethyleen Welke hulpmiddelen worden in de geografie gebruikt?
Welke hulpmiddelen worden in de geografie gebruikt?  Gezien de impact van de chemiecocktail voor binnenshuis
Gezien de impact van de chemiecocktail voor binnenshuis Spectroscopie:eenvoudige oplossing voor bodemmonster
Spectroscopie:eenvoudige oplossing voor bodemmonster Hoe het coronavirus de handhaving van de Californische luchtvervuilingsregels belemmert
Hoe het coronavirus de handhaving van de Californische luchtvervuilingsregels belemmert Bio-geïnspireerde microvasculatuur gebruiken om materiaaleigenschappen te beheersen
Bio-geïnspireerde microvasculatuur gebruiken om materiaaleigenschappen te beheersen De vorming van steenkool veranderde onze planeet bijna in een sneeuwbal
De vorming van steenkool veranderde onze planeet bijna in een sneeuwbal De geestelijke gezondheidstol van Puerto Ricos langdurige stroomuitval
De geestelijke gezondheidstol van Puerto Ricos langdurige stroomuitval
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

