
Wetenschap
De knoop doorhakken:nieuwe DNA-nanostructuren
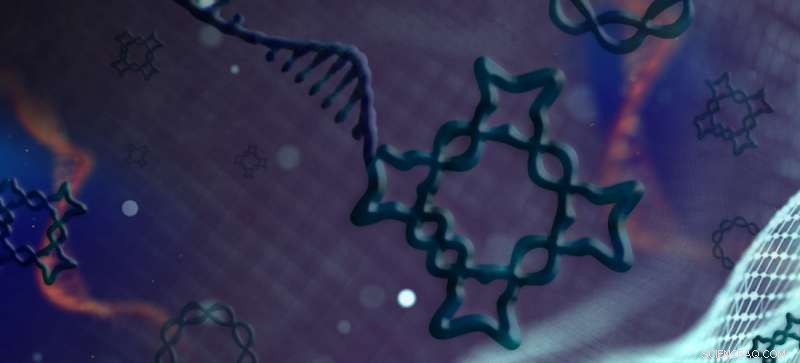
Hao Yan en zijn collega's ontwierpen en bouwden een reeks geknoopte vormen, (elk slechts een miljardste van een meter lang), uit een enkele streng DNA. De zelfassemblerende nanostructuren volgen een nauwkeurig, voorgeprogrammeerde vouwvolgorde. Krediet:Shireen Dooling
Knopen zijn onmisbare hulpmiddelen voor menselijke activiteiten als zeilen, vissen en rotsklimmen, (laat staan, schoenen strikken). Maar een knoop leggen in een kantachtige DNA-streng, met een lengte van slechts een miljardste van een meter, vereist geduld en zeer gespecialiseerde expertise.
Hoi Yan, een onderzoeker bij ASU, is een geoefende hand op dit delicate en exotische gebied, opererend op het kruispunt van nanotechnologie en beeldende kunst.
In nieuw onderzoek dat in het tijdschrift verschijnt Natuurcommunicatie , Yan en zijn collega's Fei Zhang, Xiaodong Qi en anderen beschrijven een methode om segmenten van enkelstrengs DNA over te halen tot complexe 2- en 3-D geknoopte structuren.
De resultaten vertegenwoordigen een belangrijke vooruitgang in het snelle veld van DNA-nanotechnologie, waarin het molecuul van het leven wordt gebruikt als structureel bouwmateriaal voor een groot aantal kleine configuraties. Hiertoe behoren minuscule robotapparaten, fotonische toepassingen, medicijnafgiftesystemen, logische poorten, evenals diagnostische en therapeutische toepassingen.
"De geknoopte DNA-structuren die in dit werk worden aangetoond, vertonen een ongekende topologische complexiteit, veel verder dan wat is bereikt voordat enkelstrengs vouwen werd gebruikt, " zegt Yan. "Inderdaad, het is niet alleen verbazingwekkend, maar ook verrassend dat het enkelstrengs DNA en RNA door zijn eigen ketens kunnen rijgen en een manier vinden om zulke sterk verknoopte structuren te vormen, gezien het feit dat de enkele streng door zoveel klitten moet weven."
Yan leidt het Biodesign Center for Molecular Design and Biomimetics en is de Martin D. Glick Distinguished Professor in de School of Molecular Sciences van ASU.
DNA in de plooi brengen
De nieuwe studie omvat innovaties op het gebied van DNA-origami, die, zoals de naam impliceert, gebruikt nucleïnezuren zoals DNA en RNA om te vouwen en zichzelf te assembleren tot complexe vormen. Dit gebeurt wanneer complementaire nucleotidebasen in het 4-letterige alfabet van DNA met elkaar in contact komen en binden, volgens een strikt regime:C-basen paren altijd met G en A-basen paren altijd met T.
In de natuur, strings van nucleïnezuren leveren de code die nodig is om complexe eiwitten te maken. Deze fundamentele biologie vormt de basis voor al het aardse leven. Door gebruik te maken van de eenvoudige basenparende eigenschappen van DNA, het is mogelijk om structuren te ontwerpen die zichzelf in het lab zullen assembleren. De methode is toegepast op zowel enkelstrengs als dubbelstrengs DNA-vormen, resulterend in nanostructuren van toenemende complexiteit en verfijning.
Hoewel DNA-origami sinds het begin verbazingwekkende vorderingen heeft gemaakt, één technische innovatie was buitengewoon moeilijk te realiseren. Tot nu, het creëren van complexe verknoopte structuren in DNA op een voorspelbare en programmeerbare manier is onderzoekers ontgaan.
Het nieuwe werk overwint deze hindernis, het vaststellen van precieze ontwerpregels die het mogelijk maken dat enkelstrengige DNA-segmenten (of RNA) variërend van 1800-7500 nucleotiden knoopachtige nanostructuren vormen met kruisingsgetallen (waarbij de DNA-streng in en uit zijn eigen lengte weeft) variërend van 9 tot 57 .
De groep toonde verder aan dat deze nucleïnezuur-nanostructuren kunnen worden gerepliceerd en geamplificeerd, zowel onder laboratoriumomstandigheden als binnen levende systemen.

Hao Yan leidt het Biodesign Center for Molecular Design and Biomimetics en is de Martin D. Glick Distinguished Professor in de School of Molecular Sciences van ASU. Krediet:Biodesign Instituut
De knopen van de natuur
Geknoopte structuren, zoals die Yan heeft gefabriceerd, (maar veel eenvoudiger dan de synthetische), correlaties hebben in de natuurlijke wereld. Ze zijn waargenomen in DNA en eiwitten en vormen zich in het algemeen tijdens replicatie en transcriptie, (wanneer een DNA-sequentie wordt gekopieerd naar boodschapper-RNA). Ze kunnen ook voorkomen in het genoom van fagen - virussen die bacteriële cellen infecteren.
Hoe dan ook, de constructie van moleculaire knopen op nanometerschaal, het weergeven van goed gedefinieerde en consistente geometrieën vereist enorme controle en precisie. Zoals het gebeurt, nucleïnezuren zoals DNA zijn ideaal voor het ontwerp en de synthese van dergelijke moleculaire knopen.
Eerder, stukken dubbelstrengs DNA zijn gebruikt voor constructies op nanoschaal, met de toevoeging van korte stukjes of "stapelstrengen" om de resulterende structuren aan elkaar te bevestigen. De nieuwe studie gebruikt in plaats daarvan een enkele lengte DNA die is ontworpen om zich in een nauwkeurige, voorgeprogrammeerde volgorde van stappen.
Zodra de geknoopte DNA-nanostructuren zich met succes assembleren, ze worden afgebeeld met behulp van atoomkrachtmicroscopie. Zorgvuldige berekening stelt de onderzoekers in staat om de vouwroutes te optimaliseren om de hoogste opbrengst voor elke synthetische structuur te produceren. Door het gebruik van enkelstrengs in plaats van dubbelstrengs DNA kunnen de structuren in overvloed worden geproduceerd tegen veel lagere kosten.
Een enkelstrengs benadering opent de deur voor het ontwerpen van nanoarchitecturen met specifieke, goed gedefinieerde functies, die kan worden geproduceerd door opeenvolgende ronden van in vitro evolutie, waar gewenste attributen worden geselecteerd in een zich herhalend proces van verfijning. Verder, de benadering die in de nieuwe studie wordt geschetst, biedt een algemeen platform voor het ontwerpen van moleculaire structuren van grotere omvang en ongekende complexiteit, de weg vrijmaken voor vooruitgang in nanofotonica, medicijnafgifte, cryo-EM-analyse en op DNA gebaseerde geheugenopslag.
Ontwerper-DNA (en RNA)
Voor een van de eerste knoopontwerpen was de strategie die Yan en zijn collega's ontwikkelden om een enkele streng DNA of RNA 9 keer door zichzelf te rijgen volgens een voorgeprogrammeerde volgorde, aantonen dat de nieuwe methode in staat is ingewikkelde geometrische vormen te produceren die programmeerbaar zijn, repliceerbaar en schaalbaar.
De ontwerpstrategie werd vervolgens uitgebreid met enkelstrengs RNA-structuren en 3D-DNA-knopen, waarvan de vormen werden gereconstrueerd met behulp van een techniek die bekend staat als cryogene transmissie-elektronenmicroscopie, bevestiging van hun juiste vouwen in de gewenste vormen.
"Een van de uitdagingen in dit werk is hoe het assemblagerendement van sterk geknoopte constructies kan worden verhoogd." Zei Fei. In tegenstelling tot klassieke DNA-nanostructuren, de enkelstrengs knopen zijn minder vergevingsgezind in termen van precieze vouwvolgorde vanwege de topologische complexiteit. Als een enkele kruising tijdens het proces verkeerd wordt gevouwen, de fout zal nauwelijks zelf worden gecorrigeerd en de meeste misvouwen zullen in de voltooide structuur blijven. "We hebben een hiërarchische vouwstrategie ontwikkeld om de juiste vorming van knopen te begeleiden. We vergeleken de vouwefficiëntie van een knoop met 23 kruisingen door verschillende vouwpaden te gebruiken. De AFM-afbeeldingen toonden een dramatische toename van de vouwopbrengst van goedgevormde structuren van 0,9 % tot 57,9% door een geoptimaliseerd hiërarchisch vouwpad toe te passen." voegde Fei toe.
De ontwerpregels die worden gebruikt om de vouwpaden te optimaliseren, zijn gebaseerd op het aantal kruispunten, de lengte van het DNA en het aantal basenparen in de ontworpen structuur. Er werden drie primaire regels vastgesteld. Eerst, lineaire vouwpaden bleken te verkiezen boven vertakte paden. Tweede, het opengevouwen deel van een DNA-streng mag in de vroege stadia niet door zichzelf heen gaan, wanneer de streng nog lang is. Eindelijk, randen van de gewenste vorm die drie kruisingen hebben, moeten vóór die met twee kruisingen worden gevouwen.
Door de ontwerpstrategie te volgen, het team was in staat om complexere DNA-knopen te maken met toenemende kruisingsgetallen.
Langere ketens van enkelstrengs DNA vormen unieke uitdagingen voor het ontwerpen van geprogrammeerde nanostructuren vanwege de verhoogde kans op onbedoelde zelfcomplementariteit van de basen waaruit de keten bestaat. Een DNA-knoopstructuur met 57 gekruiste knopen die met succes zijn geassembleerd, maar met een lagere opbrengst en minder precisie. Toen het oversteeknummer werd verhoogd tot 67, de opbrengst daalde aanzienlijk en de resulterende structuren, gefotografeerd door AFM, vertoonde meer montagefouten.
De studie rapporteert de grootste DNA-knopen die tot nu toe zijn verzameld, gevormd uit maximaal 7,5k basen, met de meest gecompliceerde topologieën, met maximaal 57 overstekende regio's. De enkelstrengs DNA-sequenties kunnen massaal in levende cellen worden geproduceerd voor een grotere efficiëntie tegen lagere kosten. uiteindelijk, DNA-nanostructuren met verschillende functies kunnen in cellen worden gevormd, innovaties die in de toekomst moeten worden nagestreefd.
 Eigenschappen van het Bose Einstein-condensaat
Eigenschappen van het Bose Einstein-condensaat Unieke interface en onverwacht gedrag helpen verklaren hoe zware metalen werken
Unieke interface en onverwacht gedrag helpen verklaren hoe zware metalen werken Peulvruchten opgewekt als nieuwe studie geheimen onthult van de plant die mensen kalm houdt
Peulvruchten opgewekt als nieuwe studie geheimen onthult van de plant die mensen kalm houdt Elektrochemisch geproduceerde ammoniak kan een revolutie teweegbrengen in de voedselproductie
Elektrochemisch geproduceerde ammoniak kan een revolutie teweegbrengen in de voedselproductie Chemici maken cellulaire krachten zichtbaar op moleculaire schaal
Chemici maken cellulaire krachten zichtbaar op moleculaire schaal
 NASA analyseert de ontwikkeling van System 90L in Straits of Florida
NASA analyseert de ontwikkeling van System 90L in Straits of Florida Wetenschappers gebruiken Piz Daint-simulaties om zware zomerneerslag vanuit de Middellandse Zee te volgen
Wetenschappers gebruiken Piz Daint-simulaties om zware zomerneerslag vanuit de Middellandse Zee te volgen Kunnen landen overbevissing en plasticvervuiling in slechts 10 jaar beëindigen?
Kunnen landen overbevissing en plasticvervuiling in slechts 10 jaar beëindigen? Verhoogd risico in rijst? Onderzoekers ontdekken de toxiciteit van thioarsenaten voor planten
Verhoogd risico in rijst? Onderzoekers ontdekken de toxiciteit van thioarsenaten voor planten Afbeelding:Zware regenval in Lake Success, Californië
Afbeelding:Zware regenval in Lake Success, Californië
Hoofdlijnen
- Wetenschappers ontcijferen mechanismen die ten grondslag liggen aan de biologie van veroudering
- Zijn mannelijke Y-chromosomen korter dan X-chromosomen?
- Wetenschappers visualiseren de structuur van de belangrijkste DNA-reparatiecomponent met een bijna-atomaire resolutie
- Herprogrammering van buiten naar binnen:antilichaamonderzoek suggereert een betere manier om stamcellen te maken
- Vogels leren van elkaars walging, waardoor insecten felle kleuren kunnen ontwikkelen
- Bestaat er zoiets als een werkelijk onzelfzuchtige daad?
- Oesters sluiten hun schelpen als reactie op laagfrequente geluiden
- Laat mijn brein me dingen kopen die ik niet nodig heb?
- Hoe zijn schimmels en planten vergelijkbaar?
- Eerste single-enzym methode om kwantumstippen te produceren onthuld
- Wetenschappers onderzoeken mechanismen op atomaire schaal van het groeiproces van nanodraad

- Verkeer regelen op de elektronensnelweg - onderzoek naar grafeen

- Nanokristallen gemaakt van amalgaam van twee metalen

- Nanodeeltjes die oligonucleotide-geneesmiddelen in cellen afleveren beschreven

 Burundi plant verbod op plastic tassen
Burundi plant verbod op plastic tassen Hoe klimaatverandering cultureel erfgoed beïnvloedt
Hoe klimaatverandering cultureel erfgoed beïnvloedt Nieuwe bio-geïnspireerde hydrogels kunnen werken als superlijm in zeer ionische omgevingen zoals zeewater
Nieuwe bio-geïnspireerde hydrogels kunnen werken als superlijm in zeer ionische omgevingen zoals zeewater Ocean 100:kleine groep bedrijven domineert de oceaaneconomie
Ocean 100:kleine groep bedrijven domineert de oceaaneconomie Hoe wilde paddestoelen te identificeren in Florida
Hoe wilde paddestoelen te identificeren in Florida NASA-veldtest richt zich op wetenschap van lava-terreinen vergelijkbaar met vroege Mars
NASA-veldtest richt zich op wetenschap van lava-terreinen vergelijkbaar met vroege Mars Mac-softwarebedreigingen zijn in 2019 met 400% gestegen, meer dan Microsoft Windows, rapport zegt
Mac-softwarebedreigingen zijn in 2019 met 400% gestegen, meer dan Microsoft Windows, rapport zegt Nieuw materiaal belooft snellere elektronica
Nieuw materiaal belooft snellere elektronica
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

