
Wetenschap
Vernieuwd ontwerp kan krachtige biologische computers van de reageerbuis naar de cel brengen
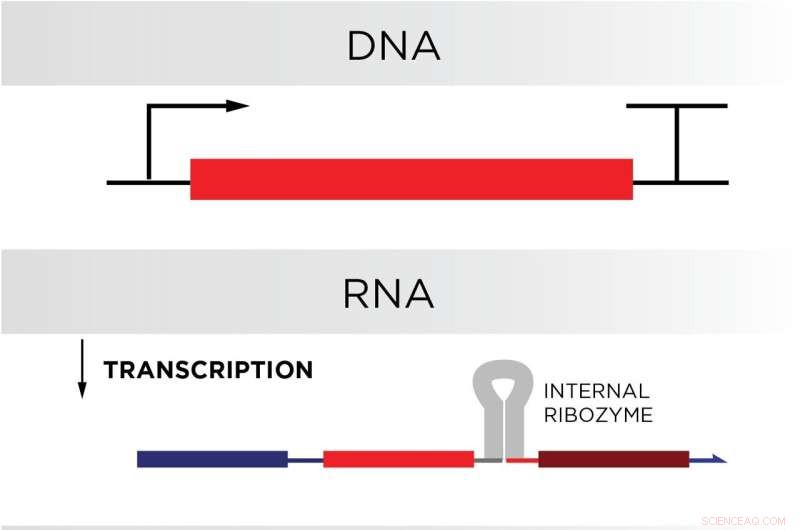
NIST-onderzoekers willen van de cel een biologische computerfabriek maken door DNA te ontwerpen en in het genoom van een cel te plaatsen. Celeiwitten zouden via transcriptie RNA produceren op basis van het DNA. De RNA-streng zou dan vouwen, aan zichzelf binden en in tweeën splitsen, dankzij een speciale zelfsplitsende RNA-sequentie die een ribozym wordt genoemd. De resulterende structuur, een RNA-circuitpoort, zou alleen onder bepaalde omstandigheden ongedaan worden gemaakt en verdere chemische reacties veroorzaken. Krediet:N. Hanacek/NIST
Kleine biologische computers gemaakt van DNA kunnen een revolutie teweegbrengen in de manier waarop we een hele reeks ziekten diagnosticeren en behandelen, zodra de technologie volledig is uitgewerkt. Een groot struikelblok voor deze op DNA gebaseerde apparaten, die zowel in cellen als in vloeibare oplossingen kunnen werken, was echter de korte levensduur ervan. Slechts één keer gebruiken en de computers zijn op.
Nu hebben onderzoekers van het National Institute of Standards and Technology (NIST) mogelijk biologische computers met een lange levensduur ontwikkeld die mogelijk in cellen kunnen blijven bestaan. In een paper gepubliceerd in het tijdschrift Science Advances , zien de auteurs af van de traditionele op DNA gebaseerde benadering en kiezen ze in plaats daarvan voor het gebruik van het nucleïnezuur-RNA om computers te bouwen. De resultaten tonen aan dat de RNA-circuits net zo betrouwbaar en veelzijdig zijn als hun op DNA gebaseerde tegenhangers. Bovendien kunnen levende cellen deze RNA-circuits continu creëren, iets dat niet gemakkelijk mogelijk is met DNA-circuits, waardoor RNA verder wordt gepositioneerd als een veelbelovende kandidaat voor krachtige, duurzame biologische computers.
Net zoals de computer of het slimme apparaat waarop u dit waarschijnlijk leest, kunnen biologische computers worden geprogrammeerd om verschillende soorten taken uit te voeren.
"Het verschil is dat je, in plaats van te coderen met enen en nullen, strings van A, T, C en G schrijft, de vier chemische basen waaruit DNA bestaat", zegt Samuel Schaffter, postdoctoraal onderzoeker van NIST en hoofdauteur van de studie. .
Door een specifieke sequentie van basen in een streng nucleïnezuur te assembleren, kunnen onderzoekers dicteren waaraan het bindt. Een streng kan worden ontworpen om zich te hechten aan specifieke stukjes DNA, RNA of sommige eiwitten die verband houden met een ziekte, en vervolgens chemische reacties teweegbrengen met andere strengen in hetzelfde circuit om chemische informatie te verwerken en uiteindelijk een soort van bruikbare output te produceren.
Die output kan een detecteerbaar signaal zijn dat medische diagnostiek kan helpen, of het kan een therapeutisch medicijn zijn om een ziekte te behandelen.
DNA is echter niet het stevigste materiaal en kan onder bepaalde omstandigheden snel uit elkaar vallen. Cellen kunnen vijandige omgevingen zijn, omdat ze vaak eiwitten bevatten die nucleïnezuren hakken. En zelfs als DNA-sequenties lang genoeg blijven hangen om hun doelwit te detecteren, maken de chemische bindingen die ze vormen ze daarna onbruikbaar.
"Ze kunnen geen dingen doen zoals het continu volgen van patronen in genexpressie. Ze zijn eenmalig, wat betekent dat ze je gewoon een momentopname geven", zei Schaffter.
Omdat het ook een nucleïnezuur is, deelt RNA veel van de problemen van DNA als het gaat om een biologische computerbouwsteen. Het is gevoelig voor snelle afbraak en nadat een streng chemisch bindt aan een doelmolecuul, is die streng af. Maar in tegenstelling tot DNA kan RNA onder de juiste omstandigheden een hernieuwbare hulpbron zijn. Om dat voordeel te benutten, moesten Schaffter en zijn collega's eerst aantonen dat RNA-circuits, die cellen theoretisch zouden kunnen produceren, net zo goed zouden kunnen functioneren als de op DNA gebaseerde soort.
De voorsprong van RNA op DNA komt voort uit een natuurlijk cellulair proces dat transcriptie wordt genoemd, waarbij eiwitten continu RNA produceren met behulp van het DNA van een cel als sjabloon. Als het DNA in het genoom van een cel codeerde voor de circuitcomponenten in een biologische computer, dan zou de cel de computercomponenten continu produceren.
In het biologische computerproces kunnen enkele strengen van nucleïnezuren in een circuit gemakkelijk worden gebonden aan andere strengen in hetzelfde circuit, een ongewenst effect dat voorkomt dat circuitcomponenten zich binden aan hun beoogde doelen. Het ontwerp van deze circuits betekent vaak dat verschillende componenten op natuurlijke wijze bij elkaar passen.
Om ongewenste binding te voorkomen, worden DNA-sequenties die deel uitmaken van computers die bekend staan als strengverdringingscircuits, gewoonlijk afzonderlijk en in dubbelstrengige vorm gesynthetiseerd (in machines in plaats van cellen). Met elke chemische base op elke streng gebonden aan een base op de andere, fungeert deze dubbele streng als een gesloten poort die alleen zou worden ontgrendeld als de doelsequentie langskwam en de plaats innam van een van de strengen.
Schaffter en Elizabeth Strychalski, leider van NIST's Cellular Engineering Group en co-auteur van de studie, probeerden deze "vergrendelde poort" -functie in hun RNA-circuit na te bootsen, in gedachten houdend dat cellen deze gesloten poorten uiteindelijk zelf zouden moeten produceren. Om cellen voor succes te maken, schreven de onderzoekers de sequenties zodat de ene helft van de strengen gelijk kon binden met de andere helft. Op deze manier binden RNA-sequenties zich op zichzelf als een hotdogbroodje, zodat ze in een vergrendelde staat zijn.
Maar om goed te werken, zouden de poorten twee chemisch gebonden maar verschillende strengen moeten zijn, meer als een hamburgerbroodje of sandwich dan een hotdogbroodje. Het team verkreeg het dubbelstrengs ontwerp in hun poorten door te coderen in een stuk RNA dat een ribozym wordt genoemd nabij het vouwpunt van de poorten. Dit specifieke ribozym - afkomstig uit het genoom van een hepatitisvirus - zou zichzelf scheiden nadat de RNA-streng waarin het was ingebed gevouwen, waardoor twee afzonderlijke strengen zouden ontstaan.
De auteurs testten of hun circuits logische basisbewerkingen konden uitvoeren, zoals alleen het ontgrendelen van hun poorten in specifieke scenario's, bijvoorbeeld als een van de twee specifieke RNA-sequenties aanwezig was of alleen als beide tegelijkertijd waren. Ze bouwden en onderzochten ook circuits gemaakt van verschillende poorten die verschillende logische bewerkingen in serie uitvoerden. Alleen wanneer deze circuits de juiste combinatie van reeksen tegenkwamen, zouden hun poorten één voor één worden ontgrendeld, zoals dominostenen.
De experimenten omvatten het blootstellen van verschillende circuits aan stukjes RNA - waarvan sommige waren ontworpen om aan te hechten - en het meten van de output van de circuits. In dit geval was de output aan het einde van elk circuit een fluorescerend reportermolecuul dat zou oplichten zodra de laatste poort was ontgrendeld.
De onderzoekers volgden ook de snelheid waarmee de poorten ontgrendelden terwijl de circuits invoer verwerkten en vergeleken hun metingen met de voorspellingen van computermodellen.
"Voor mij moesten deze in een reageerbuis net zo voorspellend werken als DNA-computing. Het leuke aan DNA-circuits is meestal dat je gewoon een reeks op een stuk papier kunt schrijven, en het zal werken zoals je wilt," zei Schaffter. "Het belangrijkste is dat we ontdekten dat de RNA-circuits erg voorspelbaar en programmeerbaar waren, veel meer dan ik dacht dat ze zouden zijn."
De overeenkomsten in prestaties tussen DNA- en RNA-circuits zouden erop kunnen wijzen dat het voordelig kan zijn om naar de laatste over te schakelen, aangezien RNA kan worden getranscribeerd om de componenten van een circuit aan te vullen. En veel bestaande DNA-circuits die onderzoekers al hebben ontwikkeld om verschillende taken uit te voeren, zouden theoretisch kunnen worden verwisseld voor RNA-versies en zich op dezelfde manier gedragen. Maar om zeker te zijn, moeten de auteurs van de studie de technologie verder ontwikkelen.
In deze studie hebben de auteurs aangetoond dat transcribeerbare circuits werken, maar ze hebben ze nog niet geproduceerd met behulp van de echte cellulaire machinerie van transcriptie. In plaats daarvan synthetiseerden machines de nucleïnezuren via een proces dat vergelijkbaar is met het proces dat wordt gebruikt om DNA voor onderzoek te produceren. Om de volgende stap te zetten, zou DNA in het genoom van een organisme moeten worden ingevoegd, waar het zou dienen als een blauwdruk voor componenten van het RNA-circuit.
"We zijn geïnteresseerd om deze vervolgens in bacteriën te stoppen. We willen weten:kunnen we circuitontwerpen in genetisch materiaal verpakken met behulp van onze strategie? Kunnen we dezelfde soort prestaties en hetzelfde gedrag krijgen als de circuits zich in cellen bevinden?" aldus Scheffer. "We hebben het potentieel om." + Verder verkennen
Wetenschappers ontwikkelen nieuwe logische DNA-circuits
Hoofdlijnen
- 7 soorten bindweefsel
- Wat zijn wierook en mirre?
- Typen agarplaten
- De controverse over de menselijke hersenen die nieuwe cellen maken
- Kenia heft effectief verbod op genetisch gemodificeerde gewassen op
- Welke organellen zouden gevonden kunnen worden in een cel die zowel eukaryoot als autotroof was?
- Zelfgemaakt skeletmodel
- Voorspellen wat uitsterven kan betekenen voor lemuren en de bossen die ze thuis noemen
- Gedetailleerd overzicht van oude Britse vogels onthult potentiële kandidaten voor herwildering
- Batterij kan worden opgeladen met kooldioxide
- Hoe maak je een eierdruppel uit alleen tandenstokers & lijm

- Een milieuvriendelijk alternatief voor giftige scheepscoatings

- Fundamentele stap toont aan dat kwantumcomputers beter kunnen zijn dan de som der delen

- Draadschroefmaat van de vierde generatie die zich kan meten met de beste ter wereld

 Opwarming van de aarde intensiveert extreme neerslag in China
Opwarming van de aarde intensiveert extreme neerslag in China Twitter gaat gemanipuleerde inhoud, waaronder deepfakes, aan banden leggen
Twitter gaat gemanipuleerde inhoud, waaronder deepfakes, aan banden leggen Nieuwe immunotherapietechniek kan specifiek gericht zijn op tumorcellen, studieverslagen
Nieuwe immunotherapietechniek kan specifiek gericht zijn op tumorcellen, studieverslagen Grote aantallen oudere Japanse vrouwen zullen binnenkort in armoede leven, voorspelt nieuw model
Grote aantallen oudere Japanse vrouwen zullen binnenkort in armoede leven, voorspelt nieuw model Onderzoek naar vloeibaar metaal roept Terminator-film op, maar veel vriendelijker
Onderzoek naar vloeibaar metaal roept Terminator-film op, maar veel vriendelijker Difference Between Community & Ecosystem
Difference Between Community & Ecosystem De redenen waarom de VS het metrische systeem niet zouden moeten gebruiken
De redenen waarom de VS het metrische systeem niet zouden moeten gebruiken  Hoe het betrouwbaarheidsinterval van de Mean
Hoe het betrouwbaarheidsinterval van de Mean
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com


