
Wetenschap
Supercomputers simuleren nieuwe paden voor mogelijke behandeling van RNA-virussen
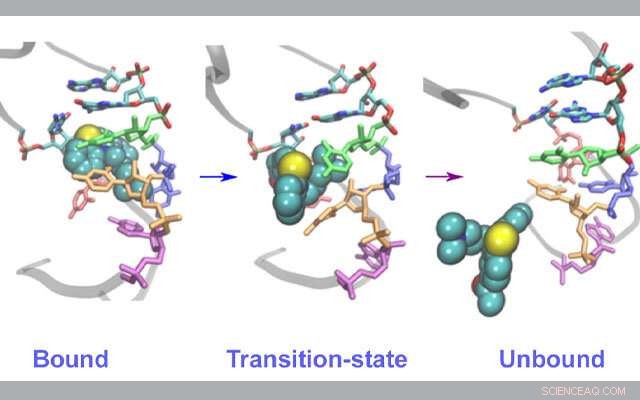
Structurele veranderingen in RNA tijdens het binden / ontbinden van geneesmiddelen. Krediet:Lev Levintov, Universiteit van New Hampshire
Onderzoekers van de Universiteit van New Hampshire (UNH) gebruikten onlangs Comet in het San Diego Supercomputer Center van UC San Diego en Stampede2 in het Texas Advanced Computing Center om nieuwe bindings-/ontbindingsroutes voor remmers in een op RNA gebaseerd virus te identificeren. De bevindingen kunnen nuttig zijn om te begrijpen hoe deze remmers reageren en mogelijk helpen bij het ontwikkelen van een nieuwe generatie medicijnen om virussen met hoge sterftecijfers aan te pakken, zoals hiv-1, Zika, ebola, en SARS-CoV2, het virus dat COVID-19 veroorzaakt.
"Toen we met dit onderzoek begonnen, we hadden nooit verwacht dat we midden in een pandemie zouden zitten die wordt veroorzaakt door een RNA-virus, " zei Harish Vashisth, universitair hoofddocent chemische technologie aan de UNH. "Als dit soort virussen opduiken, onze bevindingen zullen hopelijk een beter begrip bieden van hoe virale RNA's interageren met remmers en worden gebruikt om betere behandelingen te ontwerpen."
Vergelijkbaar met hoe mensen hun genoom coderen met behulp van DNA, veel virussen hebben een genetische samenstelling van RNA-moleculen. Deze op RNA gebaseerde genomen bevatten potentiële plaatsen waar remmers het virus kunnen hechten en deactiveren. Een deel van de uitdaging bij de ontwikkeling van geneesmiddelen is dat variaties of mutaties in het virale genoom kunnen voorkomen dat de remmers zich hechten.
In hun studie hebben onlangs gepubliceerd in de Journal of Physical Chemistry Letters , Vashisth en zijn team creëerden moleculaire dynamica-simulaties met behulp van de Comet- en Stampede2-supercomputers om specifiek te kijken naar een RNA-fragment van het HIV-1-virus en de interactie ervan met acetylpromazine. een klein molecuul waarvan bekend is dat het interfereert met het virusreplicatieproces.
De wetenschappers concentreerden zich op de structurele elementen van het HIV-1 RNA-genoom omdat ze worden beschouwd als een goed model voor het bestuderen van dezelfde processen in een breed scala aan RNA-virussen. Deze simulaties stelden hen in staat om de routes te ontdekken van de remmer die zich losmaakt van het virale RNA in verschillende zeldzame gebeurtenissen - die vaak moeilijk experimenteel waar te nemen zijn - die onverwacht een gecoördineerde beweging vertoonden in veel delen van de bindingsholte die de bouwstenen van RNA zijn.
Dankzij de Extreme Science and Engineering Environment (XSEDE)-toewijzingen van de National Science Foundation (NSF) aan Comet en Stampede2, de onderzoekers waren in staat om honderden simulaties tegelijkertijd uit te voeren om te observeren wat zeldzame base-flipping-gebeurtenissen worden genoemd die betrokken zijn bij het bindings-/ontbindingsproces van de remmer die de nieuwe details van het onderliggende mechanisme van dit proces opleverden.
"Onze hoop is dat dit nieuwe mogelijkheden toevoegt aan een veld dat traditioneel gericht is op statische biomoleculaire structuren en leidt tot nieuwe medicijnen, ' zei Vasist.
 Wetenschappers hebben eiwitten gemaakt die worden gecontroleerd door licht
Wetenschappers hebben eiwitten gemaakt die worden gecontroleerd door licht Wat als we krachtige medicijnen zouden kunnen ontwerpen zonder ongewenste bijwerkingen?
Wat als we krachtige medicijnen zouden kunnen ontwerpen zonder ongewenste bijwerkingen? Wat is een substraat in de chemie?
Wat is een substraat in de chemie?  Slimme materialen die in ultrageluid worden gebruikt, gedragen zich vergelijkbaar met water, chemici melden
Slimme materialen die in ultrageluid worden gebruikt, gedragen zich vergelijkbaar met water, chemici melden Een nieuwe draai om visco-elastische vloeistofbruggen te doorbreken
Een nieuwe draai om visco-elastische vloeistofbruggen te doorbreken
 Herbivore, Omnivoor en Carnivoor Dieren
Herbivore, Omnivoor en Carnivoor Dieren  EPA trekt beleid uit het Trump-tijdperk in dat de regels voor schone lucht versoepelde
EPA trekt beleid uit het Trump-tijdperk in dat de regels voor schone lucht versoepelde Feiten over Honey Pot Ants
Feiten over Honey Pot Ants Onderzoekers leveren het meest gedetailleerde en complete verslag tot nu toe van de laatste magnetische omkering van de aarde
Onderzoekers leveren het meest gedetailleerde en complete verslag tot nu toe van de laatste magnetische omkering van de aarde Waterplanten met speciale adaptieve functies
Waterplanten met speciale adaptieve functies
Hoofdlijnen
- Dominante fysische genen in Humans
- Studie werpt nieuw licht op de overleving van krilllarven
- Hebben hersencellen een lipide-dubbellaag?
- Genetische modificatie: definitie, soorten, proces, voorbeelden
- Schapen kunnen menselijke gezichten van foto's herkennen
- Welke soorten organische moleculen vormen een celmembraan?
- Onderzoekers identificeren een paar receptoren die essentieel zijn voor de communicatie tussen mannen en vrouwen bij planten
- Is Rust Dust schadelijk?
- Anthrax heeft mogelijk 100 nijlpaarden gedood in Namibië
- Hoe bacteriën een spaarvarken creëren voor de magere tijden
- De waterstofproductie versnellen door de magische topologische oppervlaktetoestanden
- Hoge reactiesnelheden, zelfs zonder edele metalen

- Opwekking van waterstof op zonne-energie efficiënter maken in microzwaartekracht
- Palladium, ontmoet koper:onderzoekers gebruiken machine learning om katalysatoren te verbeteren

 Nu het Amerikaanse lek is gerepareerd, Keystone-pijplijn wordt heropend
Nu het Amerikaanse lek is gerepareerd, Keystone-pijplijn wordt heropend Onderzoekers creëren een spelerskenmerkmodel dat gepersonaliseerde games mogelijk maakt
Onderzoekers creëren een spelerskenmerkmodel dat gepersonaliseerde games mogelijk maakt Onderzoekers zeggen dat kortetermijnbeheersreacties op door klimaatverandering gemedieerde rampen op de lange termijn onaangepast kunnen zijn
Onderzoekers zeggen dat kortetermijnbeheersreacties op door klimaatverandering gemedieerde rampen op de lange termijn onaangepast kunnen zijn Slotdans van ongelijke zwarte gatpartners
Slotdans van ongelijke zwarte gatpartners Antropogene forcering vergroot droogterisico's in Zuidoost-Azië
Antropogene forcering vergroot droogterisico's in Zuidoost-Azië Hoogspanning voor deeltjesversneller van morgen
Hoogspanning voor deeltjesversneller van morgen Nieuw algoritme voor misdaadbestrijding kan terugkerende illegale activiteiten voorspellen
Nieuw algoritme voor misdaadbestrijding kan terugkerende illegale activiteiten voorspellen Kan NASA natuurrampen voorspellen?
Kan NASA natuurrampen voorspellen?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | German | Dutch | Danish | Norway | Swedish |

-
Wetenschap © https://nl.scienceaq.com

