
Wetenschap
AI ontcijfert nieuwe genregulerende code in planten en maakt nauwkeurige voorspellingen voor nieuw gesequenced genomen
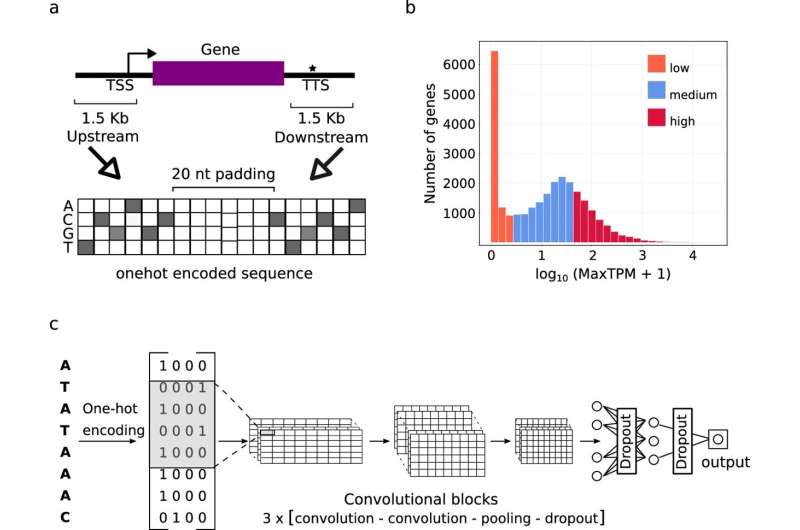
Het inzicht in hoe genetische variatie de genactiviteit op moleculair niveau beïnvloedt, is echter vrij beperkt. Deze kennislacune belemmert het kweken van ‘slimme gewassen’ met verbeterde kwaliteit en verminderde negatieve impact op het milieu, bereikt door de combinatie van specifieke genvarianten met een bekende functie.
Onderzoekers van het IPK Leibniz Instituut en Forschungszentrum Jülich (FZ) hebben een belangrijke doorbraak bereikt om deze uitdaging aan te pakken. Onder leiding van Dr. Jedrzej Jakub Szymanski trainde het internationale onderzoeksteam interpreteerbare deep learning-modellen, een subset van AI-algoritmen, op basis van een enorme dataset met genomische informatie van verschillende plantensoorten.
"Deze modellen waren niet alleen in staat om de genactiviteit op basis van sequenties nauwkeurig te voorspellen, maar konden ook vaststellen welke sequentiedelen bijdragen aan deze voorspellingen", legt het hoofd van IPK's onderzoeksgroep "Network Analysis and Modeling" uit. De AI-technologie die de onderzoekers hebben toegepast, lijkt op de technologie die wordt gebruikt in computer vision, waarbij gezichtskenmerken in afbeeldingen worden herkend en emoties worden afgeleid.
In tegenstelling tot eerdere benaderingen gebaseerd op statistische verrijking, combineerden de onderzoekers hier de identificatie van sequentiekenmerken met de bepaling van het aantal mRNA-kopieën in het kader van een wiskundig model dat getraind is om rekening te houden met biologische informatie over de structuur van het genmodel en de homologie van de sequentie. evolutie.
"We waren echt verbaasd over de effectiviteit. Binnen een paar dagen na de training herontdekten we veel bekende regulerende sequenties en ontdekten dat ongeveer 50% van de geïdentificeerde kenmerken geheel nieuw waren. Deze modellen waren uitstekend te generaliseren over plantensoorten waarvoor ze niet waren getraind, waardoor ze zijn waardevol voor het analyseren van nieuw gesequenced genomen", zegt Dr. Szymanski.
"En we hebben specifiek hun toepassing gedemonstreerd in diverse tomatencultivars met lang gelezen sequentiegegevens. We hebben specifieke variaties in de regulatiesequentie geïdentificeerd die de waargenomen verschillen in genactiviteit verklaarden en, bijgevolg, variaties in vorm, kleur en robuustheid. Dit is een opmerkelijke verbetering ten opzichte van klassiek gebruikte statistische associaties van polymorfismen met één nucleotide."
Het team heeft hun modellen openlijk gedeeld en een webinterface voor hun gebruik ter beschikking gesteld. "Interessant is dat er veel moeite is gestoken in het verslechteren van de prestaties van ons model. Om al te optimistische resultaten als gevolg van het vinden van AI-snelkoppelingen te voorkomen, was van mij een diepe duik in de genregulatiebiologie nodig om eventuele vooroordelen te elimineren, datalekken en overfitting te verminderen", zegt Fritz Forbang Peleke, de hoofdonderzoeker op het gebied van machine learning en eerste auteur van het onderzoek, dat werd gepubliceerd in het tijdschrift Nature Communications .
Dr. Simon Zumkeller, co-auteur en evolutiebioloog van FZ Jülich, zegt:"Met de gepresenteerde analyses kunnen we genregulatie in planten onderzoeken en vergelijken en de evolutie ervan afleiden. Voor praktische toepassingen biedt de methode ook een nieuwe basis. We naderen de routinematige identificatie van genregulerende elementen in bekende en nieuw gesequenced plantengenomen, in verschillende weefsels en onder verschillende omgevingsomstandigheden."