
Wetenschap
Wetenschappers simuleren eerst een grootschalig virus, M13
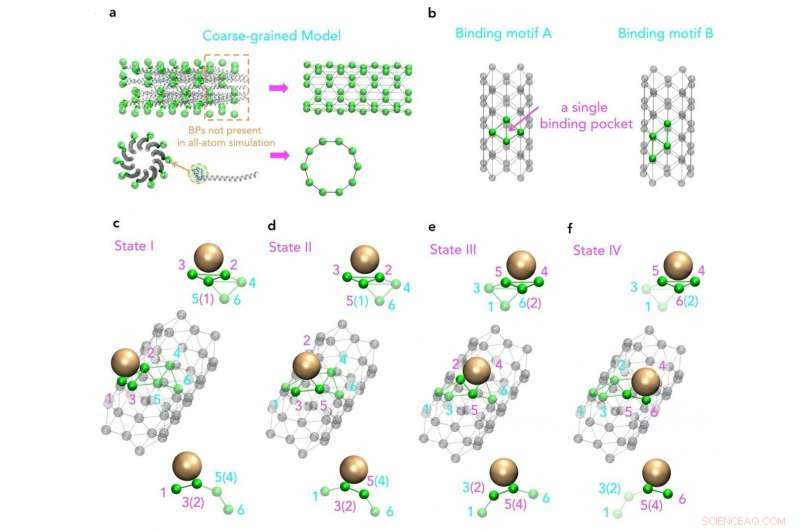
Moleculair beeld van een grofkorrelig model gebaseerd op de originele structuur van M13 belangrijkste manteleiwitten Credit:SUTD
Atomistische simulaties zijn een krachtig hulpmiddel om de beweging en interacties van atomen en moleculen te bestuderen. Bij veel biologische processen grootschalige effecten, bijvoorbeeld, assemblage van grote virussen tot nanodeeltjes zijn belangrijk. De assemblageprocessen van deze grote virussen zijn van fundamenteel belang voor het ontwerp van veel apparaten en op virale eiwit gerichte therapieën. Echter, de tijd- en lengteschaal van deze assemblageprocessen zijn meestal te groot voor simulaties met moleculaire resolutie.
Bovendien, hoewel een toename van de rekenkracht complexere en langere simulaties mogelijk maakt, virusstructuren, zoals M13, zijn nog steeds buiten bereik. Daarom heeft een onderzoeksgroep van de Singapore University of Technology and Design (SUTD) en het Massachusetts Institute of Technology (MIT) een procedure ontwikkeld die grootschalige assemblageprocessen koppelt aan moleculaire simulaties. Assistent Prof Desmond Loke van SUTD's Science, Wiskunde en Technologie cluster zei:"Voor de simulatie van M13, we begonnen met verschillende sets van krachtvelden. Er werden geschikte krachtvelden gekozen en deze werden gebruikt als input voor een molecuuldynamica-simulatie met het grofkorrelige model dat is ontworpen om het sleutelpatroon van het assemblageproces vast te leggen."
"Hoewel we weten dat op M13 gebaseerde productie fundamenteel kan worden aangedreven door nanodeeltjes-peptide-interacties, wat ook een sleutelprincipe kan zijn achter bio-engineering van het type M13, we hebben weinig kennis van hoe herhaalde patronen van korte-end-peptiden op een M13-oppervlak daadwerkelijk betrokken zijn bij deze interacties. Om dit te bestuderen, we moeten idealiter een volledige structuur van het virale manteleiwit opnemen, wat een moeilijke taak is voor de huidige state-of-the-art moleculaire dynamica-simulaties, " voegt dr. Luna Li toe, eerste auteur van het artikel.
De procedure stelt gebruikers in staat om verschillende soorten nanodeeltjes toe te voegen aan een oplossing, op een realistisch niveau. Geïnspireerd door deze procedure, Universitair docent Loke en zijn collega's konden een grootschalig virus met nanodeeltjes en in een oplossing gedurende vijftig nanoseconden simuleren.
Dr. Li zei, "De virusstructuur en -oplossing bevatten ongeveer 700, 000 atomen in totaal." Gezien de vorm en grootte van de kenmerken, de complexiteit van deze simulatie kan groter zijn dan elke eerder uitgevoerde simulatie.
"Een simulatie uitgevoerd in microseconden zou mogelijk zijn geweest als een kleiner M13-model was gebruikt, maar het kan de moeite waard zijn om de tijd te verminderen om daadwerkelijk te observeren hoe de volledige structuur de assemblage tussen de M13 en nanodeeltjes kan beïnvloeden, " verklaarde assistent-prof Loke.
 Materiaalwetenschappers stellen een nieuwe benadering voor om films voor zonnecellen te verkrijgen
Materiaalwetenschappers stellen een nieuwe benadering voor om films voor zonnecellen te verkrijgen De opwarming van de aarde aanpakken met nieuwe nanodeeltjes en zonneschijn
De opwarming van de aarde aanpakken met nieuwe nanodeeltjes en zonneschijn Een manier om de absolute stereochemie van kleine, organische moleculen
Een manier om de absolute stereochemie van kleine, organische moleculen Waarom smelt zout sneller dan suiker?
Waarom smelt zout sneller dan suiker?  Basale radioactiviteitsexperimenten
Basale radioactiviteitsexperimenten
Hoofdlijnen
- Video:Op weg naar nul honger wereldwijd
- Staan we allemaal af van een gemeenschappelijke vrouwelijke voorouder?
- Het simuleren van seks met walvisachtigen met kadaverdelen biedt inzicht in mariene copulatie
- Welk percentage botten in het lichaam bestaat uit het axiale skelet?
- Nieuwe slimme sensor om boeren te helpen kreupelheid bij schapen te herkennen
- De hoogste partiële zuurstofdruk in de bloedsomloop
- Wat is het diploïde nummer?
- Introns versus Exons: Wat zijn de overeenkomsten & verschillen?
- Hoe zijn bacteriën en plantencellen gelijk?
- Slimme stof verzamelt ruimtestof op het internationale ruimtestation

- Voedselverpakkingen kunnen de opname van voedingsstoffen in uw lichaam negatief beïnvloeden

- Nieuwe nanopincetten openen de deur naar innovaties in de geneeskunde, mobiele technologie

- Kleurpixels gemaakt van nanodraden bieden een nieuw paradigma voor digitale camera's

- Biochip-innovatie combineert AI en printen van nanodeeltjes voor analyse van kankercellen
 Moleculen afgekoeld tot onder Doppler-limiet
Moleculen afgekoeld tot onder Doppler-limiet Waarom is warm water minder dicht dan koud water?
Waarom is warm water minder dicht dan koud water?  Detectorarray demonstreert nieuwe microgolfuitlezing
Detectorarray demonstreert nieuwe microgolfuitlezing Trucs voor het oplossen van percentage woordproblemen
Trucs voor het oplossen van percentage woordproblemen  Een vleugje goud en zilver
Een vleugje goud en zilver Rechter keurt AT&T-Time Warner-deal goed regering Trump berispen
Rechter keurt AT&T-Time Warner-deal goed regering Trump berispen Onderzoekers bouwen robotwetenschapper die al een nieuwe katalysator heeft ontdekt
Onderzoekers bouwen robotwetenschapper die al een nieuwe katalysator heeft ontdekt Een nauwkeuriger, goedkope 39 GHz beamforming transceiver voor 5G-communicatie
Een nauwkeuriger, goedkope 39 GHz beamforming transceiver voor 5G-communicatie
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | German | Dutch | Danish | Norway | Swedish |

-
Wetenschap © https://nl.scienceaq.com

