
Wetenschap
Volg het ... en ze zullen komen
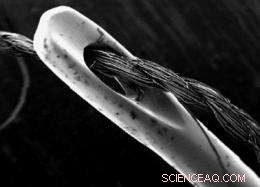
Scanning Electron Micrograph (SEM) van naald en draad. Krediet:afbeelding van het openbare domein
Snelle DNA-sequencing kan binnenkort een routineonderdeel worden van het medisch dossier van elk individu, het verstrekken van enorme informatie die voorheen was opgeslagen in de 3 miljard nucleotidebasen van het menselijk genoom. De NEWSFOCUS-sectie van deze week in het tijdschrift Wetenschap beschrijft recente ontwikkelingen in sequencing-technologie.
Stuart Lindsay, directeur van het Center for Single Molecule Biophysics van het Biodesign Institute, loopt voorop in dit onderzoek, met succes een centraal struikelblok in nanopore-sequencing hebben aangepakt - het lezen van enkele nucleotidebasen in een DNA-keten. Lindsay's laatste experimentele resultaten, die kritische verbeteringen in DNA-lezingen aantonen, zijn net in het journaal verschenen Nanotechnologie .
Zodra de nauwkeurige sequencing onder de drempel van $ 1 komt, 000 per genoom, de technologie moet alomtegenwoordig worden, volgens velen. Zoals het huidige wetenschappelijke overzicht suggereert, die dag kan naderen, aangezien zowel de snelheid als de kosten van de sequencing van het hele genoom vooruitgaan in een tempo dat de beroemde wet van Moore overtreft, (wat een verdubbeling van de rekenkracht dicteert – en een halvering van de kosten – elke 18 maanden).
Bij de nieuwste technologische wedstrijd gaat het om het idee om een enkele DNA-streng door een kleine, oogje op moleculaire schaal dat bekend staat als een nanoporie. Met deze strategie kan binnenkort de hele DNA-sequentie in één keer worden uitgelezen, in plaats van uit elkaar te knippen, ontcijferd in korte fragmenten en nauwgezet opnieuw in elkaar gezet.
Terwijl de eerste sequencing van het menselijk genoom onderzoekers 13 jaar en $ 3 miljard kostte om te bereiken, onder auspiciën van het Human Genome Project, de prestatie kan binnenkort worden bereikt met een verblindende snelheid van 6 miljard nucleotidebasen om de 6 uur tegen een kostprijs van $ 900. Dat is tenminste de extravagante bewering van Oxford Nanopore Technologies, een van de baanbrekende bedrijven die nieuwe sequencing-ontwikkelingen aansturen.
Sinds het schijnbaar quixotische idee van nanopore-sequencing voor het eerst werd bedacht in het midden van de jaren negentig, enorme vorderingen zijn gemaakt. Het basisidee is dat wanneer een nanoporie wordt ondergedompeld in een geleidende vloeistof en er een spanning over wordt aangelegd, geleiding van ionen door de nanoporie zal een meetbare elektrische stroom produceren. Deze stroom is zeer gevoelig voor de grootte en vorm van de nanoporie en in theorie, elke nucleotidebase in de DNA-draad zal de nanoporie belemmeren terwijl deze migreert, het veranderen van de ionenstroom op een herkenbare en reproduceerbare manier.
De DNA-draad is echter lastig te manipuleren, zo fijn dat er ongeveer 5000 DNA-strengen naast elkaar nodig zijn om de breedte van een mensenhaar te evenaren. Alleen al het vinden van een geschikt oogje op deze schaal bleek een uitdaging. Aanvankelijk, poreus, transmembraaneiwitten werden onderzocht. Alfa hemolysine (αHL), een bacterie die lysis van rode bloedcellen veroorzaakt, leek een bijzonder veelbelovende kandidaat, gezien de nanoporiëndiameter die nodig is voor het sequencen van DNA.
Vanaf dat moment, er is gesleuteld aan andere op eiwitten gebaseerde portalen voor DNA en meer recentelijk verschillende "vaste toestand" nanoporiën van silicium of grafeen zijn onderzocht. Deze kunnen gemakkelijker worden vervaardigd en hun eigenschappen, nauwkeuriger gecontroleerd.
Volgens Science's overzicht van de huidige stand van de techniek, nanopore-sequencing "lijkt klaar om het laboratorium te verlaten, ” en de droom van een genoom van $ 1000 kan dichtbij zijn, al blijven er uitdagingen. Een hardnekkig probleem bij het sequencen van individuele basen is dat ze de neiging hebben om te snel door de nanoporiën te stromen om elke base afzonderlijk te kunnen lokaliseren. In plaats daarvan, de gemeten stroom in vroege experimenten weerspiegelde het gemiddelde geproduceerd door een groep bases die zich een weg baant door de tunnel.
Lindsay's techniek is gebaseerd op het lezen van elektrische stroom in een klein circuit dat bestaat uit een DNA-nucleotide opgesloten tussen een paar gouden elektroden, die een nanoporie overspannen. De elektroden worden gemaakt door de punt van een scanning tunneling microscoop (STM) te functionaliseren, met moleculen die individuele DNA-basen kunnen binden terwijl ze hun hoofd door de nanoporie steken.
Erkenning Tunnelen, de naam Lindsay is van toepassing op zijn sequencing-methode, vertrouwt op het uitrusten van een van de twee elektroden met detectiechemicaliën, de andere met het te detecteren nucleotide-doel. Er wordt een signaal geproduceerd wanneer de verbinding tussen het waarnemen van chemische stof en het doel zichzelf assembleert, het circuit sluiten.
Op dit soort kruispunten waar de lengtes tussen elektroden tot op moleculaire schaal zijn, elektronen kunnen vreemd gedrag vertonen dat verband houdt met de subatomaire kwantumwereld, "tunnelen" door barrières onder omstandigheden die door de klassieke fysica zijn verboden. In een dergelijk scenario, elk van de 4 nucleotiden zou een kenmerkende tunnelstroom moeten produceren, die kan worden gebruikt om DNA base-by-base te sequensen terwijl het zich een weg baant door de nanoporie. Door elke basis tijdelijk te vangen, is er tijd voor een nauwkeurige identificatie, voordat het wordt vrijgegeven en de DNA-draad zijn transmigratie door de nanoporie voortzet.
Het vervangen van ionische stroom door tunnelstroom kan de sequentieresolutie aanzienlijk verbeteren en in hun nieuwste werk, Lindsay's groep toont aan dat multiparameteranalyse van de stroompieken die door tunneling worden geproduceerd, inderdaad elke DNA-base kan identificeren, aangezien deze tijdelijk wordt vastgezet door waterstofbinding tussen de gefunctionaliseerde elektroden.
Er is meer.
Naast het lokaliseren van de nucleotide-identiteit met een nauwkeurigheid van meer dan 90 procent, de techniek maakt het ook mogelijk om genmodificaties in het milieu te identificeren, bijvoorbeeld, methylering. Dit betekent een grote vooruitgang voor sequencing, als dergelijke epigenetische veranderingen in het genoom hebben ingrijpende gevolgen voor de studie van de menselijke gezondheid en ziekte, inclusief embryonale en postnatale ontwikkeling, en kanker.
De Nanotechnology-paper beschrijft een nieuwe benadering voor het analyseren van de tunnelingsignalen. De Lindsay-groep gebruikte machine learning (het proces dat IBM's Watson gebruikt om te winnen bij Jeopardy) om een computer te trainen om de DNA-basen te herkennen. De machine riep alle vier de basen (A, T, C en G) evenals de "vijfde base" - methyl - die de epigenetische code draagt, met een nauwkeurigheid van 96 procent op een enkel molecuul gelezen.
“Oxford Nanopore heeft een enorme doorbraak gemaakt in nanopore-sequencing met behulp van ionenstroom, zoals benadrukt in het NEWSFOCUS-verhaal, ' zegt Lindsay. "Maar we denken dat we nog meer op tafel kunnen leggen met de overgevoeligheid en chemische resolutie van Recognition Tunneling."
Roche Pharmaceuticals heeft onlangs de technologie in licentie gegeven.
De race met hoge inzet voor snelle sequencing lijkt de thuisbaan te betreden, hoewel er waarschijnlijk nieuwe verrassingen zijn voor de finish. Als het eenmaal is overgestoken, het tijdperk van gepersonaliseerde geneeskunde zal zijn aangebroken. Veel nieuwe inzichten in de genomische basis van menselijke gezondheid en ziekte zullen vrijwel zeker volgen.
 Spin dragline zijde als torsie-actuator aangedreven door vochtigheid voor toepassingen als kunstmatige spier
Spin dragline zijde als torsie-actuator aangedreven door vochtigheid voor toepassingen als kunstmatige spier Wat is warmtecapaciteit?
Wat is warmtecapaciteit?  Alternatieve cementrecepten - Een recept voor eco-beton
Alternatieve cementrecepten - Een recept voor eco-beton Ontdekking verhoogt de mogelijkheid om neurologische aandoeningen te behandelen
Ontdekking verhoogt de mogelijkheid om neurologische aandoeningen te behandelen Moleculaire twist maakt één katalysator bruikbaar voor drie waterstoftoepassingen
Moleculaire twist maakt één katalysator bruikbaar voor drie waterstoftoepassingen
 Vijf miljoen jaar klimaatverandering bewaard op één plek
Vijf miljoen jaar klimaatverandering bewaard op één plek De betekenissen van de kleuren van lieveheersbeestjes
De betekenissen van de kleuren van lieveheersbeestjes Sydney registreert heetste novembernacht terwijl hittegolf de stad overspoelt
Sydney registreert heetste novembernacht terwijl hittegolf de stad overspoelt Wereldwijde toeleveringsketens als een manier om de CO2-uitstoot te beteugelen
Wereldwijde toeleveringsketens als een manier om de CO2-uitstoot te beteugelen Uw favoriete visstroom loopt mogelijk een hoog risico op klimaatverandering - hier leest u hoe u dit kunt zien
Uw favoriete visstroom loopt mogelijk een hoog risico op klimaatverandering - hier leest u hoe u dit kunt zien
Hoofdlijnen
- Een beetje stress is goed voor de gezondheid van de cellen en een lang leven
- Bacteriën kunnen antibioticaresistentie via de bodem verspreiden
- Celmodelproject voor High School Biology
- Studie beantwoordt een al lang bestaand mysterie over slangenpredatie
- Darwins afschuwelijke mysterie oplossen:hoe bloeiende planten de wereld veroverden
- Hoe noteer ik een Karyotype
- Wat zijn de functies van mRNA & tRNA?
- Video van vier bobcats in een buitenwijk toont gezonde populatie
- Maleisië omarmt het werk van Melanies aan de illegale handel in wilde dieren
- Onderzoekers maken nanomaterialen tegen kanker door vulkanische omstandigheden onder water te simuleren

- Onderzoekers ontwikkelen slimme gouden nanokooien om chemoresistentie te doorbreken
- 3D-geprinte kubus maakt gebruik van energieoogst

- Grafeen:kan de nieuwste vorm van koolstof worden gemaakt om te buigen, Draai en rol
- Nanostraws bemonsteren de inhoud van een cel zonder schade

 Kunnen crunch-gesprekken het klimaatverdrag van Parijs tot leven brengen?
Kunnen crunch-gesprekken het klimaatverdrag van Parijs tot leven brengen? Energiekwantisatie verbetert de prestaties van warmtemachines met één atoom
Energiekwantisatie verbetert de prestaties van warmtemachines met één atoom Hoe de straal en diameter van een ovaal te berekenen
Hoe de straal en diameter van een ovaal te berekenen  Natuurkundige onderzoekt de mogelijkheid van overblijfselen van een universum voorafgaand aan de oerknal
Natuurkundige onderzoekt de mogelijkheid van overblijfselen van een universum voorafgaand aan de oerknal Thermisch geverfde meta-oppervlakken leveren perfecte lichtabsorbers op voor hightech toepassingen
Thermisch geverfde meta-oppervlakken leveren perfecte lichtabsorbers op voor hightech toepassingen Onze darmmicroben hebben circadiane ritmes,
Onze darmmicroben hebben circadiane ritmes,  Je bent op vakantie en je hebt geen telefoonopslag meer - wat te doen?
Je bent op vakantie en je hebt geen telefoonopslag meer - wat te doen? Ariane 5s tweede lancering van 2019
Ariane 5s tweede lancering van 2019
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | German | Dutch | Danish | Norway | Swedish |

-
Wetenschap © https://nl.scienceaq.com

