
Wetenschap
Superresolutiemicroscopie maakt gebruik van digitale weergavetechnologie
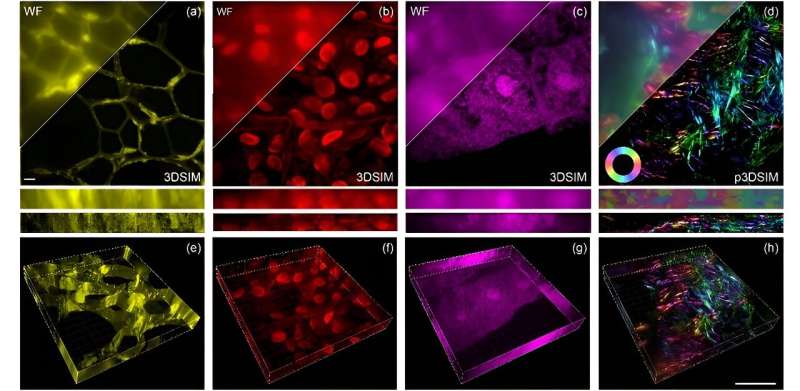
In het steeds evoluerende domein van de microscopie zijn de afgelopen jaren opmerkelijke vooruitgang geboekt op het gebied van zowel hardware als algoritmen, waardoor ons vermogen om de oneindig kleine wonderen van het leven te onderzoeken, is toegenomen. De reis naar driedimensionale gestructureerde verlichtingsmicroscopie (3DSIM) wordt echter belemmerd door uitdagingen die voortkomen uit de snelheid en complexiteit van polarisatiemodulatie.
Maak kennis met het snelle modulatie-3DSIM-systeem "DMD-3DSIM", dat digitale weergave combineert met beeldvorming met superresolutie, waardoor wetenschappers cellulaire structuren in ongekend detail kunnen zien.
Zoals gerapporteerd in Advanced Photonics Nexus , ontwikkelde het team van professor Peng Xi aan de Universiteit van Peking deze innovatieve opstelling rond een digitaal microspiegelapparaat (DMD) en een elektro-optische modulator (EOM). Het pakt resolutie-uitdagingen aan door zowel de laterale (zij-naar-zij) als axiale (boven-naar-onder) resolutie aanzienlijk te verbeteren, voor een ruimtelijke 3D-resolutie die naar verluidt twee keer zo hoog is als die wordt bereikt door traditionele breedveldbeeldvormingstechnieken.
In praktische termen betekent dit dat DMD-3DSIM ingewikkelde details van subcellulaire structuren kan vastleggen, zoals het nucleaire poriëncomplex, microtubuli, actinefilamenten en mitochondriën in dierlijke cellen. De toepassing van het systeem werd uitgebreid om zeer verstrooiende ultrastructuren van plantencellen te bestuderen, zoals celwanden in oleanderbladeren en holle structuren in zwarte algenbladeren. Zelfs in een stukje nier van een muis onthulde het systeem een uitgesproken polarisatie-effect in actinefilamenten.
Een open toegangspoort tot ontdekking
Wat DMD-3DSIM nog spannender maakt, is de toewijding aan open wetenschap. Het team van Xi heeft alle hardwarecomponenten en controlemechanismen openlijk beschikbaar gemaakt op GitHub, waardoor de samenwerking wordt bevorderd en de wetenschappelijke gemeenschap wordt aangemoedigd om op deze technologie voort te bouwen.
De DMD-3DSIM-techniek maakt niet alleen belangrijke biologische ontdekkingen mogelijk, maar legt ook de basis voor de volgende generatie 3DSIM. In toepassingen waarbij live cell imaging betrokken is, beloven ontwikkelingen op het gebied van helderdere en meer fotostabiele kleurstoffen, ruisonderdrukkingsalgoritmen en deep learning-modellen op basis van neurale netwerken de beeldduur, het ophalen van informatie en het real-time herstel van 3DSIM-beelden uit luidruchtige gegevens te verbeteren. Door hardware- en software-openheid te combineren hopen de onderzoekers de weg vrij te maken voor de toekomst van multidimensionale beeldvorming.
Meer informatie: Yaning Li et al., Snelle autopolarisatiesynchronisatiemodulatie, driedimensionale gestructureerde verlichtingsmicroscopie, Advanced Photonics Nexus (2023). DOI:10.1117/1.APN.3.1.016001
Geleverd door SPIE
 Natuurkundigen meten moleculaire elektronische eigenschappen van vitamines
Natuurkundigen meten moleculaire elektronische eigenschappen van vitamines De scheikundige laat zien dat intermoleculaire interacties voorheen onbekende dimensies kunnen bereiken
De scheikundige laat zien dat intermoleculaire interacties voorheen onbekende dimensies kunnen bereiken Wetenschappers ontwikkelen sterke maar herbruikbare lijm uit slimme materialen
Wetenschappers ontwikkelen sterke maar herbruikbare lijm uit slimme materialen  Wat is de chemische formule van bleekmiddel?
Wat is de chemische formule van bleekmiddel?
Bleach is de algemene term voor stoffen die vlekken oxideren of "bleken". Er zijn een aantal in de handel verkrijgbare bleekverbindingen. Ze worden allemaal gebruikt om het wasgoed te ontsmetten en op te
 Een veiligere bouwsteen ontwerpen voor het ontdekken van medicijnen door gebruik te maken van zichtbaar licht
Een veiligere bouwsteen ontwerpen voor het ontdekken van medicijnen door gebruik te maken van zichtbaar licht
Hoofdlijnen
- Overeenkomsten en verschillen tussen osmose en diffusie
- Onderzoek toont aan dat bodemradar fijne wortels in gewassen kan detecteren
- Verplaatste haviken gedijen goed in Hispaniola
- Hoe worden bacteriën resistent tegen antibiotica?
- Studie kijkt naar de invloed van woonwerven op voedselwebben
- De adder-nabootsende slang uit Azië is een unieke tak in de evolutionaire boom van reptielen
- Vier nieuwe soorten slakken beschreven
- Waarom bezorgen bepaalde ervaringen ons kippenvel?
- Kunnen ziektedetecterende honden de dikhoornschapen in South Dakota helpen redden?
- Eerste intrinsieke magnetische topologische isolator ontdekt

- Nieuw niveau van precisie bereikt in gecombineerde metingen van Higgs-bosonkoppelingen

- Uncle Hawking krijgt bijna een half miljard klikken van rouwende Chinezen (Update)

- Kosmologische beperkingen stellen aan de fenomenologie van kwantumzwaartekracht

- Chiraliteit geheugeneffect van ferromagnetische domeinwanden
 Wat is het verschil in graad tussen Celsius versus Fahrenheit?
Wat is het verschil in graad tussen Celsius versus Fahrenheit?  Wat is een gematigd bos?
Wat is een gematigd bos?  Onderzoekers ontwikkelen 's werelds dunste elektrische generator
Onderzoekers ontwikkelen 's werelds dunste elektrische generator Journalisten dringen aan op actie tegen Google wegens EU-auteursrechtgeschil
Journalisten dringen aan op actie tegen Google wegens EU-auteursrechtgeschil Testers onderzoeken beveiliging van auto-alarmen
Testers onderzoeken beveiliging van auto-alarmen China landt op Mars met grote vooruitgang voor zijn ruimtevaartambities
China landt op Mars met grote vooruitgang voor zijn ruimtevaartambities Burst mijndam in Zuid-Afrika:wat moet er gedaan worden om een nieuwe ramp te voorkomen
Burst mijndam in Zuid-Afrika:wat moet er gedaan worden om een nieuwe ramp te voorkomen kleine zat, grote ambities
kleine zat, grote ambities
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

