
Wetenschap
Computationele studie laat zien hoe Ebola nucleocapside stabiliseert
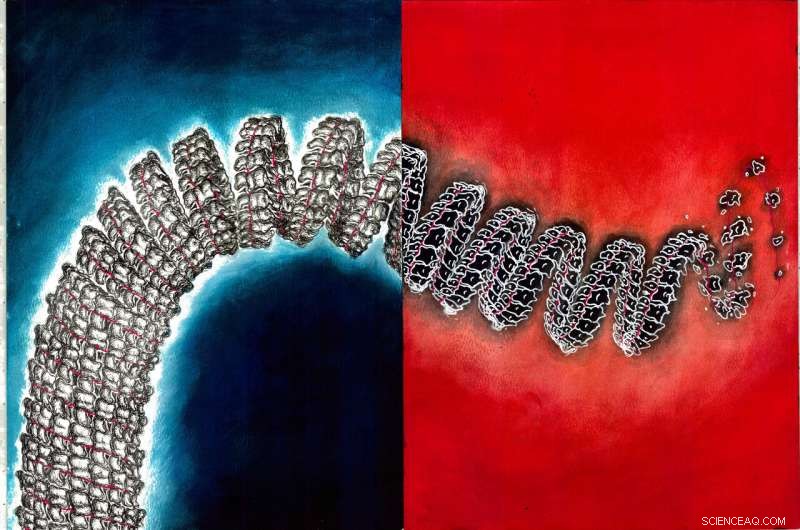
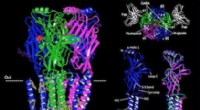
Kennis van de intermoleculaire interacties die het nucleocapside stabiliseren, dat is de spiraalvormige rangschikking van eiwitten en ssRNA hierboven getoond, is cruciaal voor het toekomstige medicijnontwerp. Door een analyse van moleculaire dynamica-simulaties, de krachten die stabiliteit geven aan het nucleocapside werden bestudeerd en gekwantificeerd in dit artikel. Krediet:Tanya Nesterova
Het ebolavirus veroorzaakt een ernstige infectie met een sterftecijfer tussen de 50% en 90%. Nucleoproteïnen in het virus assembleren in een spiraalvormige rangschikking en kapselen een enkelstrengs RNA-genoom in, ssRNA, om een staafachtig complex te vormen dat bekend staat als een nucleocapside, wat cruciaal is voor de functie van het virus. Rodachtige nucleocapsiden worden ook gevonden in andere virussen, zoals SARS-CoV-2, die COVID-19 veroorzaakt.
In de Tijdschrift voor Chemische Fysica , wetenschappers van de Universiteit van Delaware rapporteren een computationele studie van dit nucleocapside en tonen aan dat de binding van het ssRNA het nucleocapside in staat stelt zijn vorm en structurele integriteit te behouden.
Simulaties van virussen zijn moeilijk omdat de systemen erg groot zijn. Slechts een paar capsiden, inclusief hepatitis B, HPV, hiv-1, en het satelliettabakmozaïekvirus, zijn op atomair niveau onderzocht. Moleculaire dynamische simulaties van de Ebola nucleocapside, daten, zijn alleen uitgevoerd op de geïsoleerde bestanddelen en niet op atomair niveau.
Dit werk vertegenwoordigt het eerste computationele onderzoek op atomair niveau van de Ebola-nucleocapside-assemblage. Het model dat door de onderzoekers werd gebruikt, omvatte alle atomen in de spiraalvormige nucleoproteïne-assemblage, het ssRNA, water moleculen, en zelfs ionen, zoals natrium en chloride, die deze sterk geladen structuur stabiliseren.
Het resulterende model heeft 4,8 miljoen atomen, namelijk de nucleocapsidestructuur met het ssRNA aanwezig en zonder. Het tweede systeem werd opgenomen als een controle om de rol van het ssRNA te onderzoeken.
"We ontdekten dat inkapseling van ssRNA resulteert in stabilisatie van het nucleocapside van het ebolavirus en essentieel is voor het behoud van de structurele integriteit van zijn spiraalvormige assemblage, ", zei auteur Juan Perilla.
De onderzoekers ontdekten dat de nucleoproteïne-interacties en ionen bijdragen aan de stabiliteit van het nucleocapside. In het Ebola-nucleocapside, nucleoproteïnen verbinden zich met elkaar om een spiraalvormige assemblage te vormen. Natrium- en chloride-ionen bleken in de simulatie te clusteren in de buurt van het nucleocapside om de ladingsafstoting tegen te gaan.
De staafvormige nucleocapsidestructuur is essentieel voor het vermogen van het ebolavirus om cellulaire afweermechanismen te infecteren en te ontwijken, evenals voor het vermogen om zich binnen gastheercellen te repliceren. Het nucleocapside fungeert als een scaffold voor virusassemblage en als een sjabloon voor transcriptie van de genen van het virus en replicatie. Zijn cruciale rol tijdens infectie maakt het een ideale kandidaat voor antivirale interventie.
Kennis op moleculair niveau van virusdynamiek is nodig om structuur en functie te begrijpen en kwetsbaarheden op te sporen, maar het is meestal ontoegankelijk vanuit experiment. Deze inzichten zijn direct beschikbaar uit computersimulaties, echter.
Deze studie moet wetenschappers helpen bij het ontwikkelen van medicamenteuze behandelingen die gericht zijn op virale nucleocapsiden. De onderzoekers verwachten dat de methodologische benadering die ze hebben ontwikkeld voor Ebola kan worden gebruikt om andere spiraalvormige structuren te bestuderen, zoals het nucleocapside van SARS-CoV-2.
 Borax poeder maken
Borax poeder maken  Hoe houtalcohol te maken door destillatie
Hoe houtalcohol te maken door destillatie Medewerkers van de röntgenfaciliteit van Berkeley Labs mobiliseren zich om COVID-19-gerelateerd onderzoek te ondersteunen
Medewerkers van de röntgenfaciliteit van Berkeley Labs mobiliseren zich om COVID-19-gerelateerd onderzoek te ondersteunen Chemicus synthetiseert nieuwe verbindingen met sterke antidiabetische eigenschappen
Chemicus synthetiseert nieuwe verbindingen met sterke antidiabetische eigenschappen Video:Waarom stinkende kazen stinken
Video:Waarom stinkende kazen stinken
Hoofdlijnen
- Hoe het Alien Hand Syndroom werkt
- Celstructuur van Nostoc
- Wat is Lambda Max?
- Babyneushoorn galoppeert naar publiek in Singapore Zoo
- Bij-nabootsende kaalvleugelmot zoemt na 130 jaar weer tot leven
- Vier hoofdgroepen organische verbindingen waaruit levende organismen bestaan
- Tsunami onthult menselijke geluidsoverlast in Hawaiiaanse wateren
- CRISPR-dragende nanodeeltjes bewerken het genoom
- Vissers in Istanbul houden traditie in stand ondanks voorraadalarm
- Chemici ontdekken sneller werkende vormen van insecticide imidacloprid

- Onderzoekers ontwikkelen een nieuw apparaat om gluten efficiënter te detecteren

- Structuur van de belangrijkste hersenreceptor die het behandeldoel is voor epilepsie, angst opgelost
- Onderzoek van Stanford kan leiden tot injecteerbare gels die na verloop van tijd medicijnen afgeven

- Metaalionkatalysatoren en waterstofperoxide kunnen de plasticproductie groener maken

 Gadgets:slimme tags helpen je georganiseerd te blijven
Gadgets:slimme tags helpen je georganiseerd te blijven Reusachtig, gekartelde ijspieken bedekken Jupiters maan Europa, nieuwe studie suggereert:
Reusachtig, gekartelde ijspieken bedekken Jupiters maan Europa, nieuwe studie suggereert: Onderzoekers verkrijgen decaden, de grootste aceen ooit gesynthetiseerd
Onderzoekers verkrijgen decaden, de grootste aceen ooit gesynthetiseerd Natuurkundigen creëren nieuwe, eenvoudiger dan ooit quantum harde schijf voor licht
Natuurkundigen creëren nieuwe, eenvoudiger dan ooit quantum harde schijf voor licht Een nieuwe lezing over DNA-sequencing
Een nieuwe lezing over DNA-sequencing Klimaatstrijd zal slagen of mislukken in Azië:UN
Klimaatstrijd zal slagen of mislukken in Azië:UN Zwarte en Indiaanse studenten onevenredig gedisciplineerd, studie vondsten
Zwarte en Indiaanse studenten onevenredig gedisciplineerd, studie vondsten Onderzoekers ontdekken dat ruimte tussen polymeerketens de energieconversie beïnvloedt
Onderzoekers ontdekken dat ruimte tussen polymeerketens de energieconversie beïnvloedt
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

