
Wetenschap
Machine learning gebruiken om peptiden te ontwerpen
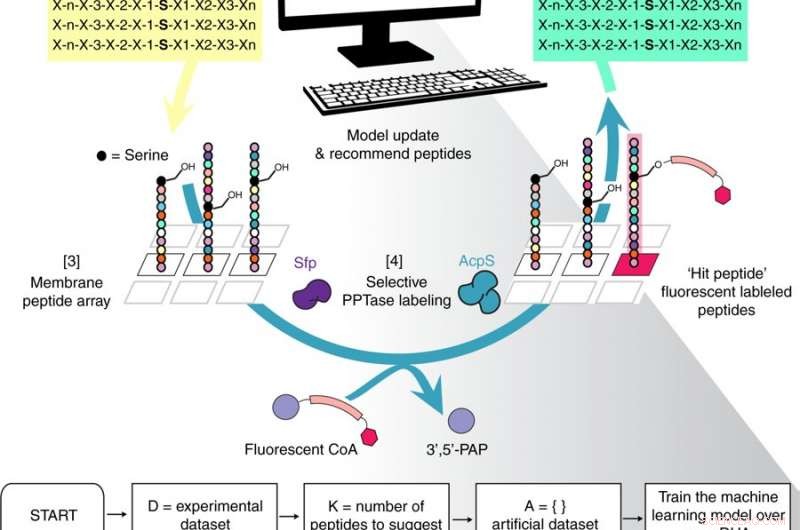
Overzicht van de iteratieve Peptide Optimization with Optimal Learning (POOL) methode workflow. Credit: Natuurcommunicatie (2018). DOI:10.1038/s41467-018-07717-6
Wetenschappers en ingenieurs zijn al lang geïnteresseerd in het synthetiseren van peptiden - ketens van aminozuren die verantwoordelijk zijn voor het uitvoeren van vele functies in cellen - om zowel de natuur na te bootsen als om nieuwe activiteiten uit te voeren. Een ontworpen peptide, bijvoorbeeld, kan een functioneel medicijn zijn dat in bepaalde delen van het lichaam werkt zonder te verslechteren, een moeilijke taak voor veel peptiden.
Maar methoden voor het ontdekken en synthetiseren van peptiden zijn duur en tijdrovend, vaak met maanden of jaren van giswerk en mislukking.
Onderzoekers van de Northwestern University, samenwerken met medewerkers van de Cornell University en de University of California, San Diego, hebben een nieuwe manier ontwikkeld om optimale peptidesequenties te vinden:met behulp van een machine-learning-algoritme als medewerker.
Het algoritme analyseert experimentele gegevens en biedt suggesties voor de volgende beste reeks om te proberen, het creëren van een heen-en-weer selectieproces dat de tijd die nodig is om het optimale peptide te vinden drastisch vermindert.
De resultaten, die een nieuw kader zou kunnen bieden voor experimenten op het gebied van materiaalwetenschap en scheikunde, werden gepubliceerd in Natuurcommunicatie op 7 dec.
"We zien dit als de volgende golf in hoe we moleculen en materialen ontwerpen, " zei de Noordwest-professor Nathan Gianneschi, een corresponderende auteur op het papier. "We kunnen wat we uit intuïtie weten combineren met de kracht van een algoritme en de oplossing vinden met minder experimenten."
Gianneschi is de Jacob en Rosaline Cohn-hoogleraar in de afdeling scheikunde van het Weinberg College of Arts and Sciences in Northwestern en in de afdelingen materiaalkunde en engineering en biomedische technologie bij Northwestern Engineering.
Om de methode te maken, Gianneschi, die ook de associate director is van het Northwestern's International Institute for Nanotechnology, samen met Peter Frazier, een universitair hoofddocent bij Cornell die werkt in operationeel onderzoek en machine learning, en Michael Burkart, een chemisch bioloog en expert in enzymologie aan UC San Diego, om een betere manier te vinden om peptiden te maken die biomaterialen zouden kunnen genereren, met name nanostructuren en microstructuren die eiwitten op bepaalde manieren zouden kunnen wijzigen. De eerste stap was het vinden van de juiste peptiden die als enzymatische substraten voor deze structuren zouden dienen.
Peptiden zijn opgebouwd uit ketens van aminozuren die wel 20 aminozuren lang kunnen zijn, met 20 verschillende mogelijkheden voor elk zuur. Omdat de sequentie de peptidefunctie bepaalt, het uitzoeken van optimale sequenties vereist dure experimenten die vaak met giswerk worden uitgevoerd.
De experimentatoren, Gianneschi en Burkart, werkte meerdere jaren met Frazier aan de ontwikkeling van een systeem dat experimentele gegevens combineerde met een algoritme voor machinaal leren om de beste strategieën te vinden voor het maken van nieuwe materialen.
Nadat Frazier het algoritme had ontworpen en de twee samenwerkten om het te trainen, de experimentatoren ontwikkelden een reeks van 100 peptiden, experimenten uitgevoerd om erachter te komen welke werkten zoals ze bedoeld waren, voerde die informatie vervolgens in het algoritme in. Het algoritme adviseerde vervolgens wat te veranderen voor de volgende ronde van peptideontwikkeling, en adviseerde ook strategieën waarvan het dacht dat het zou mislukken.
"Nu begonnen we selectiviteit te krijgen, " zei Gianneschi. Door dit proces meerdere keren te voltooien, ze waren in staat om optimale peptiden te vinden.
"In plaats van te gissen en naar miljoenen peptiden te kijken, we waren in staat om naar honderden peptiden te kijken en heel snel samen te komen tot sequenties die zich op volledig nieuwe manieren gedroegen, " zei hij. In vergelijking met willekeurige mutaties of giswerk, de algoritmemethode was statistisch gezien veel succesvoller.
Hoewel dit werk gericht was op substraten, dit proces kan worden gebruikt om peptiden voor elk soort doel te ontdekken, zoals medicijnafgifte, en misschien zelfs worden gebruikt om DNA-sequenties te ontdekken, ook. Omdat elke vorm van optimale volgorde kan worden ontdekt, onderzoekers zijn ook niet beperkt tot welke aminozuursequenties in de genetische code worden gevonden.
De volgende stap is het automatiseren van het hele proces. Gianneschi is ook geïnteresseerd in het gebruik van de methode om optimale oppervlakken voor polymeren te vinden, met name polymeren die worden gebruikt in medische implantaten. Het vinden van de juiste oppervlakken die binden aan weefsel of spieren kan helpen om littekenweefsel of implantaatafstoting te voorkomen.
"Je zou in wezen sequenties kunnen ontdekken die specifieke dingen doen, wat echt de kern is van wat peptiden en nucleïnezuren in de natuur doen, " zei hij. "Dit kan een revolutie teweegbrengen in de manier waarop we peptiden maken."
 Voorbeelden van zure buffers
Voorbeelden van zure buffers Het benutten van omkeerbare oplosbaarheid zorgt voor directe, optische patronen van ongekend kleine features.
Het benutten van omkeerbare oplosbaarheid zorgt voor directe, optische patronen van ongekend kleine features. Wetenschappers ontdekken materiaal dat ideaal is voor slimme fotovoltaïsche ramen
Wetenschappers ontdekken materiaal dat ideaal is voor slimme fotovoltaïsche ramen Studie werpt nieuw licht op productie van hydroxylradicalen, die helpen bij het afbreken van luchtverontreinigende stoffen
Studie werpt nieuw licht op productie van hydroxylradicalen, die helpen bij het afbreken van luchtverontreinigende stoffen Waar staat HSS voor in het staal?
Waar staat HSS voor in het staal?
 Japan zet zich schrap voor meer regen na overstromingen aardverschuivingen
Japan zet zich schrap voor meer regen na overstromingen aardverschuivingen Nabijheid van frackingsites beïnvloedt de publieke steun voor hen, studie vondsten
Nabijheid van frackingsites beïnvloedt de publieke steun voor hen, studie vondsten Fotosyntheselaboratoriumexperimenten
Fotosyntheselaboratoriumexperimenten Vroegste bosbranden bewijs van uitbreiding van oude bomen
Vroegste bosbranden bewijs van uitbreiding van oude bomen Vroegste bewijs ooit van loodvervuiling gevonden in de Balkan - van 3600 voor Christus
Vroegste bewijs ooit van loodvervuiling gevonden in de Balkan - van 3600 voor Christus
Hoofdlijnen
- Onderzoek heroverweegt het evolutionaire belang van variabiliteit in een populatie
- Toch niet zo verschillend:menselijke cellen, winterharde microben delen een gemeenschappelijke voorouder
- Voorbeelden van hittebestendige bacteriën
- Uitsterven dreigt voor twee zeldzame vogelsoorten na verwoestende orkanen
- Eenvoudig epitheelweefsel: definitie, structuur en voorbeelden
- Anatomy & Physiology Project Ideeën
- De evolutionaire oorsprong van de darm
- Hoe antibioticagebruik bij dieren bijdraagt aan antibioticaresistentie
- Twee eiwitten behouden de pluripotentie van embryonale stamcellen op verschillende manieren
- Cellulaire klepstructuur opent potentiële nieuwe therapieën
- Wetenschappers testen met succes nieuw watersimulatieprotocol

- Chemische vergelijkingen combineren

- Hoe robotwiskunde en smartphones onderzoekers naar een doorbraak in de ontdekking van medicijnen hebben geleid

- Squishy hydra's eenvoudige circuits klaar voor hun close-up

 Groene oplosmiddelen vinden voor gedrukte elektronica
Groene oplosmiddelen vinden voor gedrukte elektronica Gemotoriseerde sensoren zijn bedoeld om de diagnose van ziekten in een vroeg stadium te verbeteren en te versnellen
Gemotoriseerde sensoren zijn bedoeld om de diagnose van ziekten in een vroeg stadium te verbeteren en te versnellen Onderzoek onthult dat klimaatverandering kwetsbare berghabitats drastisch kan veranderen
Onderzoek onthult dat klimaatverandering kwetsbare berghabitats drastisch kan veranderen Een deep learning neuraal netwerk gebruiken om een auto in slechts 20 minuten te laten leren zelf te rijden
Een deep learning neuraal netwerk gebruiken om een auto in slechts 20 minuten te laten leren zelf te rijden Air Canada mist winstprognose door 737 MAX aan de grond
Air Canada mist winstprognose door 737 MAX aan de grond Wat zijn de basiseenheden lengte, volume, massa en temperatuur in het metrische systeem?
Wat zijn de basiseenheden lengte, volume, massa en temperatuur in het metrische systeem?  VW wil automarkt bestormen met goedkoper elektrisch model
VW wil automarkt bestormen met goedkoper elektrisch model Hoe vangen cellen energie die vrijkomt door cellulaire ademhaling?
Hoe vangen cellen energie die vrijkomt door cellulaire ademhaling?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

