
Wetenschap
Hoe levende machines te zien
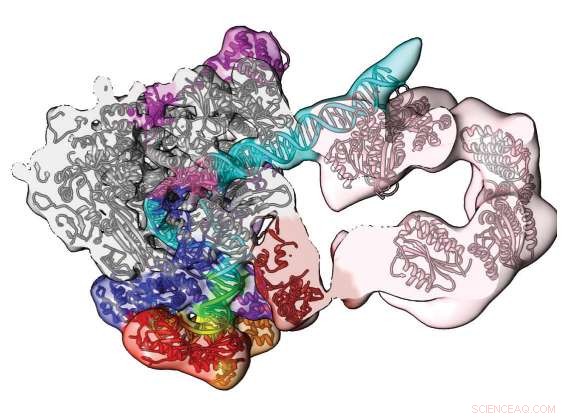
Supercomputing en cryo-elektronenmicroscopie onthullen dit deel van het menselijke pre-initiatiecomplex. De open conformatiedichtheidskaart en het model tonen het pad van het DNA (blauw/groen) en de betrokkenheid ervan door de transcriptiefactorcomponent TFIIH (roze). Herdrukt met toestemming van Macmillan Publishers Ltd:Hij, Y. et al. Bijna-atomaire resolutievisualisatie van de opening van de menselijke transcriptiepromotor. Natuur 533, 359-365 (2016).
Het klinkt als iets uit de Borg in Star Trek. Robots van nanoformaat assembleren zichzelf om biologische machines te vormen die het werk doen dat iemand in leven houdt. En toch gaat zoiets echt door.
Elke cel in ons lichaam - of het nu vlees en bloed is, hersenen en alles daartussenin - heeft identiek DNA, de kronkelende trap van nucleïnezuren die uniek zijn gecodeerd voor elk organisme. Complexe assemblages die lijken op moleculaire machines, nemen stukjes DNA die genen worden genoemd en maken zo nodig een hersencel. in plaats van, zeggen, een botcel. Deze moleculaire machines zijn zo complex, toch zo klein, dat wetenschappers tegenwoordig net hun structuur en functie beginnen te begrijpen met behulp van de nieuwste microscopen en supercomputers. Biologische moleculaire machines zouden de basis kunnen leggen voor het ontwikkelen van behandelingen voor ziekten zoals kanker. Hoe klein kun je zien, en wat zal men vinden?
Cryo-elektronenmicroscopie gecombineerd met supercomputersimulaties hebben tot nu toe het beste model gecreëerd, met bijna atomaire details, van een vitale moleculaire machine, het menselijke pre-initiatiecomplex (PIC). Een wetenschappelijk team van de Northwestern University, Berkeley Nationaal Laboratorium, Staatsuniversiteit van Georgië, en UC Berkeley publiceerden hun resultaten op de PIC mei 2016 in het tijdschrift Natuur .
"Voor de eerste keer, structuren zijn gedetailleerd van de complexe groepen moleculen die menselijk DNA openen, " zei co-auteur van de studie Ivaylo Ivanov, universitair hoofddocent scheikunde aan de Georgia State University. Ivanov leidde het computationele werk dat de atomen van de verschillende eiwitten modelleerde die werken als tandwielen van de PIC-moleculaire machine.
De PIC vindt genen die geassocieerd zijn met het maken van een specifiek eiwit, zoals een antilichaam of een enzym. Daar trekt de PIC de twee DNA-strengen uit elkaar en voedt de coderende streng naar het werkpaard-enzym RNA-polymerase II. Hiermee wordt de transcriptie gestart, waarbij DNA-bits worden gekopieerd door RNA-polymerase II in een enkele streng boodschapper-RNA. Het RNA baant zich een weg naar 'eiwitfabrieken' in de cel, ribosomen genaamd, die ze als opdrachten voor het maken van eiwitten nemen. Als DNA is als de blauwdruk van een nieuw huis, RNA's zijn instructies aan de 'contractors' op het ribosoomwerkstation. De vervaardigde eiwitten zijn als de nagels, hout, gips, en zo ongeveer alles in huis.
Het experiment begon met zorgvuldig genomen foto's van PIC. Ze zijn gemaakt door een groep onder leiding van co-auteur Eva Nogales, een professor in de afdeling Moleculaire en Cellulaire Biologie aan de UC Berkeley en ook Senior Faculty Scientist bij het Lawrence Berkeley National Laboratory en Howard Hughes Medical Investigator.
De groep van Nogales gebruikte cryo-elektronenmicroscopie (cryo-EM), een rijzende ster in laboratoriumtechnieken. Ze bevroor cryogeen menselijke PIC gebonden aan DNA. Door het invriezen bleef het in een chemisch actieve, bijna natuurlijke omgeving. Vervolgens zapten ze het met elektronenstralen. Dankzij recente ontwikkelingen op het gebied van directe elektronendetectortechnologie, cryo-EM kan nu grote en gecompliceerde biologische structuren in beeld brengen met bijna atomaire resolutie die te moeilijk te kristalliseren zijn gebleken. De go-to-techniek, X-ray kristallografie, vereist gekristalliseerde monsters, en cryo-EM vermijdt deze moeilijke stap.
Meer dan 1,4 miljoen cryo-EM 'freeze frames' van PIC werden verwerkt met behulp van supercomputers bij het National Energy Research for Scientific Computing Center om achtergrondruis te filteren en driedimensionale dichtheidskaarten te reconstrueren die details in de vorm van het molecuul laten zien die nooit eerder waren al gezien.
"Cryo-EM maakt een grote expansie door, net als alle computersoftware die wordt gebruikt om zowel de dichtheidskaarten te genereren als om ze te interpreteren, zoals we in deze studie hebben gedaan, "Zei Nogales. "Het stelt ons in staat om een hogere resolutie te krijgen van meer structuren in verschillende staten, zodat we niet slechts één foto kunnen beschrijven van hoe ze eruit zien, maar maar verschillende foto's laten zien hoe ze bewegen. We zien geen continuüm, maar we zien snapshots door het proces van actie."
Studiewetenschappers bouwden vervolgens een nauwkeurig model dat fysieke betekenis gaf aan de dichtheidskaarten van PIC met behulp van XSEDE, de eXtream Science and Engineering Discovery-omgeving, gefinancierd door de National Science Foundation. XSEDE stelt wetenschappers in staat om computerbronnen interactief te delen, data en expertise via één virtueel systeem. Het team van Ivaylo Ivanov heeft meer dan vier miljoen kernuren aan simulaties uitgevoerd op de Stampede-supercomputer in het Texas Advanced Computing Center om complexe moleculaire machines te modelleren, inclusief die voor dit onderzoek. Ivanovs bredere moleculaire machinewerk omvat ook een XSEDE-toewijzing van 1,7 miljoen kernuren op de Comet-supercomputer in het San Diego Supercomputing Center.
"Ik gebruik XSEDE-middelen nu al meer dan 12 jaar, " zei Ivanov. "Zonder de beschikbaarheid van XSEDE-bronnen, al ons onderzoek zou veel beperkter zijn geweest in termen van de systemen die we kunnen aanpakken. Voor ons, XSEDE is absoluut essentieel geweest."
Het doel van al deze rekeninspanningen is om atomaire modellen te produceren die het volledige verhaal vertellen van de structuur en functie van het eiwitcomplex van moleculen. Om daar te komen nam het team van Ivanov de twaalf componenten van de PIC-assemblage en creëerde homologiemodellen voor elke component die verantwoordelijk waren voor hun aminozuursequenties en hun relatie met vergelijkbare bekende eiwit 3-D-structuren.
Vervolgens benaderden ze de experimentele dichtheden die Nogales' team op een raster vond. "We kunnen een methode gebruiken die moleculaire dynamica flexibele aanpassing wordt genoemd, " legde Ivanov uit, "waar je in wezen een moleculaire dynamica-simulatie uitvoert. En je gebruikt de experimentele dichtheid om de atomen in de moleculaire dynamica-simulatie te beïnvloeden om naar de dichtere gebieden van de EM-kaart te gaan. Dat is het proces van flexibel aanpassen aan de EM-kaart."
Ze verfijnden het model met het Phoenix-kristallografische verfijningspakket. "Dat is een aanvullende techniek waarmee we zijketens kunnen positioneren en het model kunnen verbeteren, zodat we alle details kunnen vastleggen die aanwezig zijn in de dichtheidskaart, ' zei Ivanov.
XSEDE was "absoluut noodzakelijk" voor deze modellering, zei Ivanov. "Als we naast het PIC-complex water en tegenionen opnemen in een simulatiebox voor moleculaire dynamica, we krijgen de grootte van het simulatiesysteem van meer dan een miljoen atomen. Dat kun je niet draaien op een werkplek of zelfs op een bescheiden cluster. Daarvoor moeten we echt naar duizend cores. In dit geval, we gingen naar tweeduizend achtenveertig kernen. En daarvoor hadden we toegang nodig tot Stampede, ' zei Ivanov.
Een van de inzichten die in de studie zijn opgedaan, is een werkmodel van hoe PIC de anders stabiele dubbele DNA-helix opent voor transcriptie. Nogales legde uit dat je je een koord kunt voorstellen dat bestaat uit twee draden die om elkaar heen zijn gedraaid. Houd het ene uiteinde heel stevig vast. Pak de andere vast en draai deze in de tegenovergestelde richting van de draad om het koord te ontrafelen. Dat is eigenlijk hoe de levende machines die ons in leven houden het doen.
"Het DNA moet worden geopend en verplaatst naar de actieve plaats van het polymerase om te coderen voor het eerste RNA-nucleotide, ", legt Nogales uit. "Het pre-initiatiecomplex houdt de twee strengen van het DNA aan één uiteinde heel stevig bij elkaar, zodat ze niet kunnen bewegen en ze niet kunnen openen. Aan de andere kant van de PIC staat een machine die energie gebruikt om het DNA te duwen, draai het in de tegenovergestelde richting waarin de twee strengen zijn geregen. En wanneer dit gebeurt, tussen de twee kanten, de strengen gaan open, ' zei Nogales.
Deze studie loste de structuur op van die moleculaire machine die werkt als de draaiende vingers, de transcriptiefactorcomponent TFIIH. "TFIIH heeft een translocase-subeenheid, wiens rol het is om tegelijkertijd het DNA naar de actieve plaats van het polymerase te duwen en het DNA af te wikkelen. Door het gecombineerd duwen en afwikkelen, effectief scheid je de twee strengen van het DNA, ' zei Ivanov.
Beide wetenschappers zeiden dat ze net een atomair begrip van transcriptie beginnen te krijgen. cruciaal voor genexpressie en uiteindelijk voor ziekte. "Veel ziektetoestanden ontstaan omdat er fouten zijn in hoeveel een bepaald gen wordt gelezen en hoeveel een bepaald eiwit met een bepaalde activiteit in de cel aanwezig is, Nogales zei. "Die ziektetoestanden kunnen te wijten zijn aan overmatige productie van het eiwit, of omgekeerd niet genoeg. Het is erg belangrijk om het moleculaire proces te begrijpen dat deze productie reguleert, zodat we de ziektetoestand kunnen begrijpen."
"Dit werk illustreert goed twee algemene principes die de wetenschap in de komende jaren zullen aandrijven, " merkte Peter Preusch op, programmamedewerker bij de National Institutes of Health (NIH). "Een daarvan is de toepassing van hybride methoden - combinaties van biofysische methoden, waaronder röntgenkristallografie en cryoEM, samen met grootschalige computermethoden om informatie over grotere moleculaire complexen te integreren. Twee, er is de vereiste voor teamwetenschap die de expertise van meerdere onderzoekers trekt om problemen op te lossen die niet door een enkel laboratorium alleen kunnen worden aangepakt." Peter Preusch is de Biophysics Branch Chief, Afdeling Celbiologie en Biofysica, Nationaal Instituut voor Algemene Medische Wetenschappen, NIH.
Hoewel dit fundamentele werk niet direct genezingen oplevert, het legt wel de basis om ze in de toekomst te helpen ontwikkelen, zei Ivanov. "Om ziekte te begrijpen, we moeten in de eerste plaats begrijpen hoe deze complexen functioneren... Een samenwerking tussen computationele modelbouwers en experimentele structurele biologen zou in de toekomst zeer vruchtbaar kunnen zijn. "
Het Nature Articles-onderzoek van mei 2016 (DOI:10.1038/nature17970), "Bijna-atomaire resolutievisualisatie van de opening van de menselijke transcriptiepromotor, " is geschreven door Yuan He, Lawrence Berkeley National Laboratory en nu aan de Northwestern University; Chunli Yan en Ivaylo Ivanov, Staatsuniversiteit van Georgië; Jie Fang, Carla Inouye, Robert Tjian, Eva Nogales, UC Berkeley. De financiering kwam van het National Institute of General Medical Sciences (NIH) en de National Science Foundation.
 Onverwachte ontdekking over essentieel enzym
Onverwachte ontdekking over essentieel enzym Mechano-responsieve hydrogel ontwikkeld voor wondgenezing
Mechano-responsieve hydrogel ontwikkeld voor wondgenezing Een methode voor computerondersteunde modellering en simulatie van grote eiwitten en andere biomoleculen
Een methode voor computerondersteunde modellering en simulatie van grote eiwitten en andere biomoleculen Artritis, ontdekking van auto-immuunziekten kan leiden tot nieuwe behandelingen
Artritis, ontdekking van auto-immuunziekten kan leiden tot nieuwe behandelingen Bacteriën groeien in agar
Bacteriën groeien in agar
 Redenen om Pauwen te vrezen
Redenen om Pauwen te vrezen  Gates biedt $ 1,5 miljard aan klimaathulp als de VS wetgevende maatregelen neemt
Gates biedt $ 1,5 miljard aan klimaathulp als de VS wetgevende maatregelen neemt Per saldo, sommige neonicotinoïde pesticiden kunnen bijen ten goede komen:studie
Per saldo, sommige neonicotinoïde pesticiden kunnen bijen ten goede komen:studie Multi-universitair centrum streeft naar minimalisering van mijnafvalrisico
Multi-universitair centrum streeft naar minimalisering van mijnafvalrisico Twee voorbeelden van hoe de Nijl gevormd is Ancient Egypt
Twee voorbeelden van hoe de Nijl gevormd is Ancient Egypt
Hoofdlijnen
- Hoe is een Paramecium Digest Food?
- Temperatuur kan de pollenkleur beïnvloeden
- Factoren die betrokken zijn bij celdifferentiatie
- Wanneer één referentiegenoom niet genoeg is
- Hoe virussen de manier veranderen waarop we naar evolutie kijken
- Welk percentage botten in het lichaam bestaat uit het axiale skelet?
- Het is wiskundig onmogelijk om veroudering te verslaan, wetenschappers zeggen:
- Genoomanalyse van vroege plantenafstamming werpt licht op hoe planten leerden te gedijen op het land
- Stelten vliegen honderden kilometers om eieren te leggen die meer dan 50% van hun lichaamsgewicht uitmaken
- Onderzoek onthult betrouwbaarheidsproblemen voor koolstofnanobuisjes in toekomstige elektronica

- Nieuwe manier om magnetische informatie te schrijven kan de weg vrijmaken voor neurale hardwarenetwerken

- Glas heeft nu slimme potentie

- Degradatie van zonnecellen voor het eerst direct waargenomen

- Nieuwe technologie voor het afleveren van nanocarrier-medicijnen passeert de bloed-hersenbarrière
 NIST-tool maakt uitgebreidere tests op risicovolle software mogelijk
NIST-tool maakt uitgebreidere tests op risicovolle software mogelijk Een ontwerpstrategie om stabiele en energierijke waterige zink-mangaandioxidebatterijen te produceren
Een ontwerpstrategie om stabiele en energierijke waterige zink-mangaandioxidebatterijen te produceren Klimaateffect van irrigatie boven het stroomgebied van de Gele Rivier
Klimaateffect van irrigatie boven het stroomgebied van de Gele Rivier Nieuw-Zeeland laatste land om ruimteovereenkomst met NASA te ondertekenen
Nieuw-Zeeland laatste land om ruimteovereenkomst met NASA te ondertekenen Chemici waarschuwen voor onredelijke verspilling bij aanschaf van wetenschappelijke apparatuur
Chemici waarschuwen voor onredelijke verspilling bij aanschaf van wetenschappelijke apparatuur Langst bekende belichtingsfoto ooit gemaakt met een bierblikje
Langst bekende belichtingsfoto ooit gemaakt met een bierblikje Redoxdivergente strategie voor constructie van (dihydro)thiofenen met dimethylsulfoxide
Redoxdivergente strategie voor constructie van (dihydro)thiofenen met dimethylsulfoxide Parasitisme: definitie, types, feiten en voorbeelden
Parasitisme: definitie, types, feiten en voorbeelden
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

