
Wetenschap
Met kracht vingerafdrukken van eiwitten opent een nieuwe weg voor proteomics met één molecuul
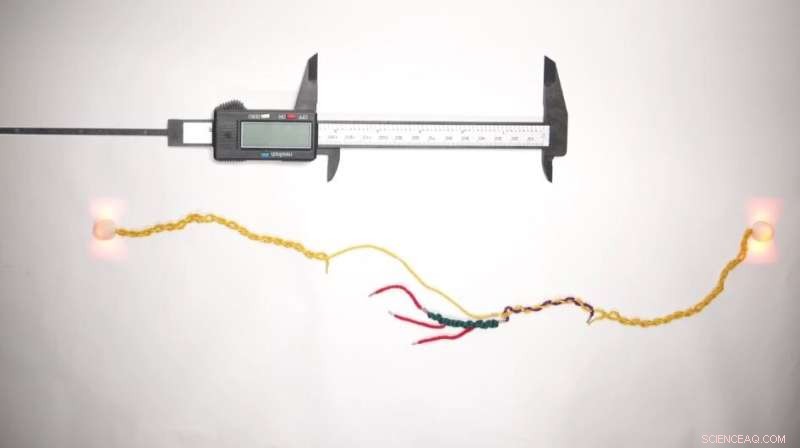
Krediet:Harvard University
Terwijl wetenschappers de mysteries van het leven op steeds kleinere schaal hebben onderzocht, hebben ze hulpmiddelen uitgevonden om hen te helpen begrijpen wat ze waarnemen. Het bepalen van de identiteit van DNA- en RNA-moleculen is nu gemeengoed geworden dankzij de commerciële ontwikkeling van next-generation sequencing-technologieën, maar hetzelfde geldt nog niet voor eiwitten, die van cruciaal belang zijn in bijna alle biologische processen. Eiwitten zijn veel complexer dan DNA en RNA en zijn vaak chemisch gemodificeerd, waardoor het een uitdaging is om eenvoudig afzonderlijke eiwitten in een monster te identificeren (proteomics met één molecuul).
Nu hebben wetenschappers van het Molecular Robotics Initiative binnen het Wyss Institute van de Harvard University, het Blavatnik Institute van de Harvard Medical School (HMS) en het Boston Children's Hospital (BCH) DNA, de fundamentele stof van het leven zelf, gebruikt om te creëren wat mogelijk is. wees 's werelds kleinste liniaal voor het meten van eiwitten.
Deze technologie, genaamd "DNA Nanoswitch Calipers" (DNC), stelt onderzoekers in staat om afstandsmetingen uit te voeren op afzonderlijke peptiden (de bouwstenen van eiwitten) met hoge precisie door kleine hoeveelheden kracht uit te oefenen. Door snel veel afstandsmetingen uit te voeren op hetzelfde molecuul, creëert DNC een unieke "vingerafdruk" die kan worden gebruikt om het in volgende experimenten te identificeren. De prestatie wordt gerapporteerd in Nature Nanotechnology .
"Als je iets in de biologie probeert te begrijpen, zijn er twee belangrijke onderzoeksmethoden:je kunt je onderwerp in zijn natuurlijke staat observeren, of je kunt het verstoren en zien hoe het reageert. Waarnemingen kunnen veel geweldige biologische informatie opleveren, maar soms is de beste manier om iets te leren door er fysiek mee om te gaan, "zei co-corresponderende auteur Wesley Wong, Ph.D., een geassocieerd lid van de faculteit aan het Wyss Institute en universitair hoofddocent bij HMS, die ook onderzoeker is bij BCH . "Het bepalen van het patroon van aminozuren in een peptidemolecuul door kracht uit te oefenen is een nieuw paradigma in de voortdurende wetenschappelijke zoektocht naar technieken die ons in staat zullen stellen eiwitten net zo gemakkelijk te sequencen als we momenteel DNA sequencen."
Gebruik de kracht
DNC is gebaseerd op de onderliggende technologie van de DNA-nanoschakelaar:een enkele DNA-streng waaraan op meerdere punten langs de lengte moleculaire "handvatten" zijn bevestigd. Wanneer twee van deze handvatten aan elkaar binden, creëren ze een lus in de DNA-streng en wordt de totale lengte van de streng verkort. Wanneer kracht wordt uitgeoefend om de handvatten uit elkaar te trekken, strekt de streng zich terug naar zijn oorspronkelijke lengte. Het verschil tussen de lengte van de streng in zijn lus- en niet-lustoestand weerspiegelt de grootte van de lus, en dus de afstand tussen de handvatten.
Het onderzoeksteam realiseerde zich dat ze DNA-nanoschakelaars nog een stap verder konden brengen:als ze in plaats daarvan de handvatten zo zouden ontwikkelen dat ze aan een biomolecuul binden, zouden de handvatten het molecuul effectief tussen elkaar kunnen "knijpen" zoals de twee uiteinden van een schuifmaat, in plaats van aan elkaar te binden. ander. Door te meten hoe de toevoeging van het doelmolecuul tussen de handvatten de totale lengte van de DNA-nanoschakelaar in zijn lus- versus niet-lustoestand veranderde, veronderstelde het team dat ze de grootte van het molecuul effectief konden meten.
"In sommige opzichten maken DNA-nanoschakelaars gebruik van een van de meest klassieke, mechanische methoden voor het meten van objecten:oefen gewoon kracht uit op iets en kijk hoe het als reactie verandert", zegt co-eerste auteur Darren Yang, een postdoctoraal onderzoeker aan het Wyss Institute en BCH. "Het is een benadering die we nog niet echt hebben gebruikt op het gebied van single-molecule proteomics, omdat het uitoefenen van kracht op zulke kleine objecten een ongelooflijke uitdaging is. Maar we waren klaar voor de uitdaging."
Om hun idee van een nieuwe, op kracht gebaseerde meettechniek om te zetten in realiteit, bevestigden Yang en zijn collega's eerst twee verschillende soorten handvatten aan een doelmolecuul:één "sterk" handvat om het molecuul stevig aan één uiteinde van de DNC te verankeren, en verschillende "zwakke" handvatten die aan het andere uiteinde van de DNC kunnen worden bevestigd. Vervolgens bonden ze beide uiteinden van de DNC vast aan twee "optisch opgesloten" kralen die in laserstralen waren opgehangen. Door de kralen dichter bij elkaar te brengen, zorgden ze ervoor dat een van de zwakke handvatten van het doelmolecuul zich aan de DNC bindt, waardoor een lustoestand ontstond. Toen ze vervolgens de kracht verhoogden door de kralen verder uit elkaar te bewegen, maakte de zwakke hendel uiteindelijk zijn binding los, waardoor de DNC terugkeerde naar zijn langere, niet-geluste toestand.
Het team testte deze techniek eerst op eenvoudige, enkelstrengs DNA-moleculen (ssDNA) en bevestigde dat de verandering in afstandsmetingen tussen de lusvormige en niet-lusvormige toestand van de DNC direct correleerde met de lengte van het doelmolecuul. Deze lengteveranderingen konden worden gemeten met een precisie op angstrom-niveau (dat is tien keer kleiner dan de breedte van een dubbele DNA-helix), waardoor de identificatie van veranderingen in lengte zo klein als die van een enkele nucleotide mogelijk is.
Omdat het doelmolecuul meerdere zwakke handvatten bevat die aan de DNC kunnen binden, creëren herhaalde cycli van binden en breken van die handvatten een reeks afstandsmetingen tussen de sterke handgreep en de zwakke handvatten die uniek zijn voor elk gemeten molecuul. Deze "vingerafdruk" kan worden gebruikt om een bekend molecuul in een monster te identificeren of om structurele informatie over een onbekend molecuul af te leiden.
Eiwitten onderzoeken
Nadat ze hadden bevestigd dat DNC op betrouwbare wijze de grootte van DNA-moleculen kon meten, verlegden de onderzoekers de aandacht naar hun echte doel:eiwitten. Ze ontwierpen een synthetisch peptide (een korte keten van aminozuren) met een bekende lengte en volgorde en herhaalden het experiment door het aan het ene uiteinde van de DNC te bevestigen via de sterke handgreep en herhaaldelijk de banden tussen de zwakke handgrepen en de DNC bevestigen en verbreken door verschillende hoeveelheden kracht uit te oefenen. Ze ontdekten dat alle afstanden die hun gereedschap tussen de sterke en zwakke handvatten meet, overeenkwamen met de verwachte afstanden op basis van de lengte van de DNC en de lengtes van de aminozuren in het peptide. Ze kregen ook vergelijkbare resultaten toen ze de DNC gebruikten om een natuurlijk voorkomend gelineariseerd peptide genaamd NOXA BH3 te meten.
Dit proces genereerde ook unieke meetvingerafdrukken voor elk peptide. Het team creëerde een computermodel om te voorspellen hoeveel menselijke eiwitten uniek kunnen worden geïdentificeerd met behulp van deze methode, en ontdekte dat meer dan 75% van de eiwitten in een veelgebruikte eiwitdatabase kon worden geïdentificeerd via vingerafdrukken met een waarschijnlijkheid van ten minste 90%.
"We waren eigenlijk enigszins verrast door hoe goed deze techniek werkte", zegt co-eerste auteur Prakash Shrestha, Ph.D., een postdoctoraal onderzoeker aan het Wyss Institute en BCH. "Optische pincetten bestaan al tientallen jaren en het fietsen van DNA tussen een geluste en een niet-geluste toestand bestaat al ongeveer 10 jaar, en we wisten niet zeker of we metingen met een voldoende hoge resolutie konden krijgen door die ideeën te combineren. Maar het bleek dat deze vingerafdrukken zijn zeer effectief voor het identificeren van eiwitten."
Het identificeren van enkelvoudige eiwitmoleculen is op zich al een indrukwekkende prestatie, maar om dat voor meerdere eiwitten tegelijk te kunnen doen, is de ware heilige graal voor enkelvoudige moleculen proteomics. Het team toonde verder aan dat ze, door de optische kralen te vervangen door een magnetisch pincetsysteem, in staat waren om parallelle metingen uit te voeren op meerdere verschillende peptiden en om de relatieve concentraties van verschillende moleculen te bepalen.
"Single-molecule proteomics is nog steeds grotendeels een luchtkasteel vanwege uitdagingen in schaalvergroting en resolutie. Ons huidige werk laat zien dat op kracht gebaseerde sequentie-vingerafdrukken het potentieel hebben om deze droom te realiseren", zei de corresponderende auteur William Shih, Ph.D. , een kernfaculteitslid aan het Wyss Institute en professor aan HMS en het Dana-Farber Cancer Institute. "Onze ultieme ambitie is om niet alleen eiwitsequenties, maar ook eiwitstructuren efficiënt te lezen op een manier met een hoge doorvoer."
De volgende stap van de wetenschappers in de richting van dat doel is het valideren van hun schuifmaten voor structurele metingen met lage kracht op gevouwen eiwitten en hun complexen, waarbij ze hun potentiële gebruik voor structurele biologie en proteomics onderzoeken. Ze werken ook aan het verhogen van de doorvoer van de technologie om de analyse van gemengde monsters verder te versnellen.
"Dit onderzoek integreert moleculaire biofysica met geavanceerde DNA-nanotechnologie die hier bij het Wyss Institute is gepionierd om ons in staat te stellen op een werkelijk nieuwe manier te interageren met biologische moleculen en deze te analyseren. Toen William en Wesley dit idee voor het eerst stelden als een kernuitdaging voor de nieuw gevormde Molecular Robotics Initiative, het leek echt sciencefiction, maar dat is precies het soort project dat we bij de Wyss willen aannemen. Ik ben erg trots op het team dat deze technologie heeft gerealiseerd - het heeft het potentieel om totaal te veranderen hoe we wetenschap bedrijven en therapieën ontwikkelen", zegt Wyss Founding Director Don Ingber, M.D., Ph.D., tevens Judah Folkman Professor of Vascular Biology aan de Harvard Medical School en BCH, en Professor of Bioengineering aan de Harvard John A. Paulson School of Engineering en Toegepaste Wetenschappen. + Verder verkennen
Democratisering van high-throughput enkelvoudige molecuulkrachtanalyse
 Grootschalige bereiding van op polymeer gebaseerde fosforescentie bij kamertemperatuur via klikchemie
Grootschalige bereiding van op polymeer gebaseerde fosforescentie bij kamertemperatuur via klikchemie Onderzoekers rapporteren fasestabiel anorganisch halide perovskiet
Onderzoekers rapporteren fasestabiel anorganisch halide perovskiet Termietendarmmicroben kunnen de productie van biobrandstoffen ondersteunen
Termietendarmmicroben kunnen de productie van biobrandstoffen ondersteunen Ecologische energie-accu gemaakt van vanilline
Ecologische energie-accu gemaakt van vanilline Door koper gekatalyseerde enantioselectieve trifluormethylering van benzylische radicalen ontwikkeld
Door koper gekatalyseerde enantioselectieve trifluormethylering van benzylische radicalen ontwikkeld
Hoofdlijnen
- Lichaamsdelen en functies
- Gloeiende tags onthullen een fractie van een seconde activiteit van pathogene circuits
- De adelaars scouten:het bewijs dat het beschermen van nesten de voortplanting bevordert
- Kun je verslaafd zijn aan endorfine?
- Wat volgt glycolyse als zuurstof aanwezig is?
- Een plantencel maken uit gerecycleerde materialen
- Golgi-apparaat: functie, structuur (met analogie en diagram)
- Relatie tussen DNA-basen Genen, eiwitten en eigenschappen
- 5 mensen die niet begraven kunnen blijven
- Nanostructuur verhoogt de stabiliteit van organische dunnefilmtransistoren

- Elektrisch circuit gemaakt van gel kan zichzelf repareren

- Laser-geïnduceerd grafeen wordt taai, met hulp
- Nanorobots kunnen zich richten op kankers en bloedstolsels verwijderen
- Een LiDAR-apparaat ter grootte van een vinger nu beschikbaar
 Plant op proefschaal om zeldzame aardelementen te winnen in het hart van het kolenland
Plant op proefschaal om zeldzame aardelementen te winnen in het hart van het kolenland Het samenvoegen van geheugen en berekening, programmeerbare chipsnelheden AI, verlaagt stroomverbruik
Het samenvoegen van geheugen en berekening, programmeerbare chipsnelheden AI, verlaagt stroomverbruik Onze kusten op natuurlijke wijze beschermen
Onze kusten op natuurlijke wijze beschermen Nieuwe testmethode suggereert dat baby Anzick-1 even oud was als de omliggende Clovis-artefacten
Nieuwe testmethode suggereert dat baby Anzick-1 even oud was als de omliggende Clovis-artefacten Een nieuwe benadering van ultrasnelle lichtpulsen
Een nieuwe benadering van ultrasnelle lichtpulsen SpaceX klaar om vracht te lanceren vanaf historisch NASA-pad
SpaceX klaar om vracht te lanceren vanaf historisch NASA-pad Thaise retailers verbieden plastic tassen voor eenmalig gebruik
Thaise retailers verbieden plastic tassen voor eenmalig gebruik Het is niet hoe je het spel speelt, maar hoe werden de dobbelstenen gemaakt?
Het is niet hoe je het spel speelt, maar hoe werden de dobbelstenen gemaakt?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

