
Wetenschap
3D-AFM-beelden simuleren voor systemen die niet in evenwicht zijn
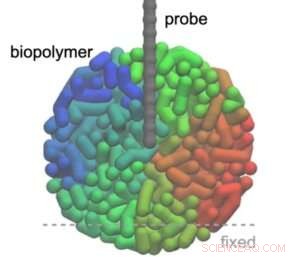
Krediet:Kanazawa University
Onderzoekers van Kanazawa University rapporteren in het Journal of Physical Chemistry Letters hoe 3D atoomkrachtmicroscopiebeelden te simuleren van systemen die niet in evenwicht zijn met biomoleculen. De benadering maakt gebruik van een beroemde vergelijking uit de thermodynamica die van toepassing is op situaties zonder evenwicht.
Driedimensionale atoomkrachtmicroscopie (3D-AFM) is een techniek die wordt gebruikt voor het onderzoeken van de verdeling van oplosmiddelmoleculen op vaste-vloeistofgrensvlakken. Aanvankelijk toegepast voor het bestuderen van situaties waarin het oplosmiddel water is, wordt de methode nu ook gebruikt voor andere moleculen. Een recente ontwikkeling is het gebruik van 3D-AFM voor het oplossen van de organisatie van biopolymeren zoals chromosomen of eiwitten in cellen. Vanwege de complexiteit van dergelijke systemen zijn echter simulaties van het 3D-AFM-beeldvormingsproces nodig om te helpen bij de interpretatie ervan. Simulatiemethoden die tot nu toe zijn ontwikkeld, hebben aangenomen dat het gesondeerde systeem in evenwicht is tijdens de AFM-scancyclus. Dit beperkt hun geldigheid tot situaties waarin de oplosmiddelmoleculen veel sneller bewegen dan de scanningsonde. Nu hebben Takeshi Fukuma van Kanazawa University en collega's een 3D-AFM-simulatiebenadering ontwikkeld die werkt voor systemen die niet in evenwicht zijn, van toepassing op situaties waarin moleculaire beweging plaatsvindt op tijdschalen die vergelijkbaar zijn met of groter zijn dan die van de AFM-sonderingscyclus.
Het basisprincipe van AFM is om een zeer kleine tip te maken, bevestigd aan een cantilever, om het oppervlak van een monster te scannen. De reactie van de tip op hoogteverschillen in het gescande oppervlak geeft structurele informatie over het monster. In 3D-AFM wordt de tip gemaakt om het monster te penetreren, en de kracht die de tip ervaart is het resultaat van interacties met nabijgelegen (delen van) moleculen. Voor een gegeven horizontale (xy) positie van de punt, wordt de afhankelijkheid van de kracht F van de verticale (z) positie van de punt wanneer deze in het monster doordringt, vastgelegd in een kracht-afstand (F versus z) curve. Het combineren van alle kracht-afstandscurven die zijn verkregen tijdens de xy-scan geeft het 3D-AFM-beeld.
Fukuma en collega's beschouwden de situatie waarin een AFM-tip een bolvormig biopolymeer meet, en zowel de tip als het molecuul modelleerden als kralen verbonden door veren (2.000 kralen voor het molecuul, 50 kralen voor de tip). Ze berekenden de kracht-afstandskrommen met behulp van de zogenaamde Jarzynski-gelijkheid, een vergelijking die het vrije energieverschil tussen twee toestanden van een systeem relateert aan de arbeid (evenredig met de kracht) die nodig is om van de ene toestand naar de andere te gaan. Belangrijk is dat de gelijkheid geldt voor situaties die niet in evenwicht zijn.
De onderzoekers konden aantonen dat de simulaties de interne structuur van het biopolymeer reproduceerden, waarbij sommige vezelkenmerken duidelijk waarneembaar waren. Ze keken ook naar hoe de scansnelheid de simulatieresultaten beïnvloedt en ontdekten dat er een optimaal snelheidsbereik is voor de verticale (z) scan. Ten slotte simuleerden Fukuma en collega's 3D-AFM-afbeeldingen van cytoskeletvezels waarvoor experimenteel verkregen 3D-AFM-afbeeldingen bestaan, en ontdekten dat de simulaties goed overeenkomen met het experiment. De wetenschappers concludeerden daarom dat hun methode "van toepassing is op verschillende vezels in cellen zoals DNA enzovoort door parameters zoals stijfheid te veranderen, wat een belangrijke theoretische basis vormt voor dergelijke experimentele metingen." + Verder verkennen
Bewegende kenmerken toewijzen in snelle atoomkrachtmicroscopie
 Magnetische tanden zijn veelbelovend voor materialen en energie
Magnetische tanden zijn veelbelovend voor materialen en energie In het laboratorium gemaakt hormoon kan geheime levens van planten onthullen
In het laboratorium gemaakt hormoon kan geheime levens van planten onthullen Thermische oorkappen beschermen de batterijen van mobiele telefoons tegen extreme temperaturen
Thermische oorkappen beschermen de batterijen van mobiele telefoons tegen extreme temperaturen Wetenschappers ontwikkelen Venus flytrap-biosensoren om verontreinigende stoffen te vangen
Wetenschappers ontwikkelen Venus flytrap-biosensoren om verontreinigende stoffen te vangen Berekeningskracht van aantrekkingskracht tussen Ions
Berekeningskracht van aantrekkingskracht tussen Ions
 Bosbranden, barbecueverboden:hittegolf verschroeit Europa
Bosbranden, barbecueverboden:hittegolf verschroeit Europa Wetenschappers vestigen nieuwe records van de geschiedenis van de zeespiegel in Singapore
Wetenschappers vestigen nieuwe records van de geschiedenis van de zeespiegel in Singapore Hoe vang je een blauwe krab in Florida
Hoe vang je een blauwe krab in Florida Detectie van microplastics eerste stap bij beoordeling van milieuschade
Detectie van microplastics eerste stap bij beoordeling van milieuschade Canadezen sterven sneller in gebieden met meer luchtvervuiling
Canadezen sterven sneller in gebieden met meer luchtvervuiling
Hoofdlijnen
- Wat hebben spam-e-mail en hiv met elkaar gemeen?
- Wat maakt de mens menselijk?
- De koala:wanneer het slim is om langzaam te zijn
- Graslandmussen constant op zoek naar een mooier huis
- Een modelhart maken met materialen uit uw huis
- Studie vindt variatie binnen soorten een cruciaal aspect van biodiversiteit
- De Manchineel,
- Illinois sportvis herstel een resultaat van 1972 Clean Water Act, wetenschappers rapporteren
- Hoe ver reizen invasieve soorten?
- Onderzoekers geven een nieuwe draai aan de oude techniek om beter absorberende materialen te ontwikkelen

- Nagemaakte likeuren opsnuiven

- Oppervlaktemodificatie voor het verbeteren van de in vivo levensduur van bio-implantaten

- Hoe maak je een lab-on-a-chip helder en biocompatibel (met minder bloedspatten)

- Onderzoekers boeken vooruitgang in groene chemie met nieuwe katalysator voor reductie van koolstofdioxide

 Een nieuw mechanisme in celdeling ontdekken
Een nieuw mechanisme in celdeling ontdekken Astronomen brengen massieve structuur in kaart voorbij Laniakea Supercluster
Astronomen brengen massieve structuur in kaart voorbij Laniakea Supercluster Hoe weten wetenschappers de structuur van het binnenste van de aarde?
Hoe weten wetenschappers de structuur van het binnenste van de aarde?  Wetenschappers brengen de puls van grondwaterstroming door Californias Central Valley in kaart
Wetenschappers brengen de puls van grondwaterstroming door Californias Central Valley in kaart Geavanceerde DNA-labs onthuld om de vermisten op te sporen
Geavanceerde DNA-labs onthuld om de vermisten op te sporen Het produceren van foutloze metaalkristallen van ongekende grootte
Het produceren van foutloze metaalkristallen van ongekende grootte Goud extraheren van Scrap
Goud extraheren van Scrap Hoe de barometrische druk in mijn gebied te vinden
Hoe de barometrische druk in mijn gebied te vinden
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

