Wetenschap
Nieuwe structurele eenheid vereenvoudigt het proces van het op maat ontwerpen van eiwitten
Tegoed:Unsplash/CC0 Publiek domein
Een paar onderzoekers van de Universiteit van Californië, San Francisco, heeft een nieuwe eiwitstructuur ontwikkeld waarmee het proces van het op maat ontwerpen van eiwitten kan worden vereenvoudigd. In hun artikel gepubliceerd in het tijdschrift Wetenschap , Nicholas Polizzi en William DeGrado bespreken hun structurele eenheid en hoe ze die hebben gebruikt. Anna Pauw, met de Universiteit van Birmingham, heeft in hetzelfde tijdschriftnummer een Perspective-stuk gepubliceerd waarin het werk van het team in Californië wordt geschetst.
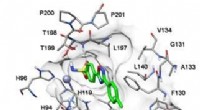
Een van de dingen die scheikundigen moeten doen, zijn op maat gemaakte eiwitten voor gebruik in bepaalde speciale toepassingen. Zoals de onderzoekers opmerken, dit wordt als zeer uitdagend beschouwd. Het gaat meestal gepaard met een aanzienlijke hoeveelheid vallen en opstaan, wat zich over het algemeen vertaalt in hoge ontwikkelingskosten. In deze nieuwe poging de onderzoekers hebben een nieuwe eenheid van eiwitstructuur bedacht om bij dergelijke projecten te helpen. Ze noemen het een van der Mer-structuur en beschrijven hoe het kan worden gebruikt om de functionaliteit van de chemische groep van liganden direct toe te wijzen aan de coördinaten van de peptideresidu-backbone.
Om tot de nieuwe structuur te komen, het onderzoekspaar goot en analyseerde duizenden eiwitstructuren in de Protein Data Bank. Hun aanpak verschilde van de norm doordat ze de positionering van zijketens voor het aminozuurresidu negeerden en zich in plaats daarvan concentreerden op de chemische groepen die in contact kwamen met de residuen. Met behulp van deze methode, ze waren in staat om twee nieuwe eiwitten te ontwerpen die kunnen worden gebruikt om een medicijn genaamd apixaban te identificeren. Opmerkelijk, hun aanpak omvatte het maken van slechts zes sequenties. Voorafgaand aan hun werk, zo'n proces zou doorgaans veel meer hebben gekost.
De naam voor de nieuwe structuur kwam van het combineren van de aantrekkingskrachten van Van der Waals met rotameren - het soort zijketenconformaties die aminozuren kunnen aannemen. De nieuwe structuur werkt door de ruggengraat van aminozuren in kaart te brengen naar locaties van chemicaliën in de Protein Data Bank die betrokken zijn bij interacties ermee. De onderzoekers merken op dat de databank pas sinds kort voldoende informatie bevat om het in een dergelijke toepassing te kunnen gebruiken. En ze merken ook op dat de techniek en structuur ook kunnen worden gebruikt om leveringsvehikels te produceren op basis van eiwitten en ook toepassingen met kleine moleculen.
© 2020 Wetenschap X Netwerk
Hoofdlijnen
- Arctisch, grote visserslanden zijn het erover eens dat er niet gevist wordt in het noordpoolgebied, voor nu
- Wat is histonacetylatie?
- Zeldzame vliegende vossen neergeschoten bij gruwelijke aanval in Australië
- Dark Repair Mechanism Vs. Lichtreparatie in DNA
- Oorzaken van Cytoplasmic Streaming
- GMO-experimenten
- Wat zijn de drie belangrijkste verschillen tussen een plantencel en een dierencel?
- Celstructuurdefinities
- Hevig debat komt tot leven over annulering jacht op grizzlyberen
 Ierlands enige dinosaurussen ontdekt in Antrim
Ierlands enige dinosaurussen ontdekt in Antrim Biobased chemicaliën staan centraal
Biobased chemicaliën staan centraal Jelly Beans tellen in een pot
Jelly Beans tellen in een pot Zes soorten kristallijne vaste stoffen
Zes soorten kristallijne vaste stoffen Nieuw opgewaardeerde laser tuurt verder in het extreme universum bij SLAC's LCLS
Nieuw opgewaardeerde laser tuurt verder in het extreme universum bij SLAC's LCLS Wetenschappers beweren dat controversiële resultaten van komeetwaarnemingen consistent zijn
Wetenschappers beweren dat controversiële resultaten van komeetwaarnemingen consistent zijn DNA-kooi kan nanopore-technologie verbeteren
DNA-kooi kan nanopore-technologie verbeteren De Kaderrichtlijn Water op de proef stellen
De Kaderrichtlijn Water op de proef stellen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- Italian | Spanish | Swedish | German | Dutch | Danish | Norway | French | Portuguese |

-
Wetenschap © https://nl.scienceaq.com