
Wetenschap
Nieuwe stoffenbibliotheek om het zoeken naar actieve stoffen te versnellen
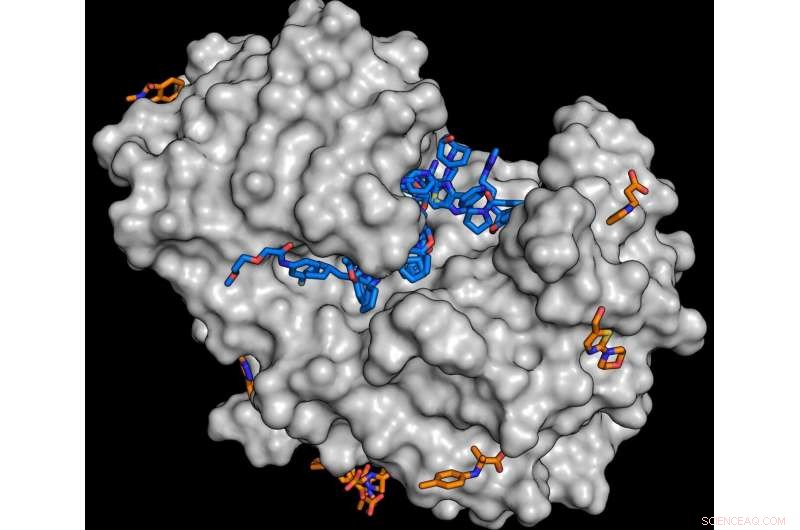
Voor de studie, het enzym endothiapepsine (grijs) werd gecombineerd met moleculen uit de fragmentbibliotheek. Uit de analyse blijkt dat tal van stoffen aan het enzym kunnen koppelen (blauwe en oranje moleculen). Elke gevonden stof is een mogelijk startpunt voor de ontwikkeling van grotere moleculen. Credit:J. Wollenhaupt/HZB
Om de systematische ontwikkeling van medicijnen te versnellen, het MX-team van het Helmholtz-Zentrum Berlin (HZB) en de Drug Design Group van de Universiteit van Marburg hebben een nieuwe stoffenbibliotheek opgezet. Het bestaat uit 1103 organische moleculen die kunnen worden gebruikt als bouwstenen voor nieuwe medicijnen. Het MX-team heeft deze bibliotheek nu gevalideerd in samenwerking met de FragMAX-groep bij MAX IV. De stoffenbibliotheek van de HZB is wereldwijd beschikbaar voor onderzoek en speelt ook een rol bij het zoeken naar stoffen die werkzaam zijn tegen SARS-CoV-19.
Om medicijnen effectief te laten zijn, ze moeten meestal aanmeren aan eiwitten in een organisme. Als een sleutel in een slot, een deel van het geneesmiddelmolecuul moet in uitsparingen of holten van het doeleiwit passen. Sinds een aantal jaren, het team van de afdeling Macromoleculaire Kristallografie (MX) van de HZB onder leiding van Dr. Manfred Weiss samen met de Drug Design Group onder leiding van prof. Gerhard Klebe (Universiteit van Marburg) heeft daarom gewerkt aan het opbouwen van zogenaamde fragmentbibliotheken. Deze bestaan uit kleine organische moleculen (fragmenten) waarmee de functioneel belangrijke holtes op het oppervlak van eiwitten kunnen worden gesondeerd en in kaart gebracht. Eiwitkristallen worden verzadigd met de fragmenten en vervolgens geanalyseerd met krachtig röntgenlicht. Hierdoor kan driedimensionale structurele informatie worden verkregen op niveaus van atomaire resolutie. Onder andere, het is mogelijk om erachter te komen hoe goed een specifiek molecuulfragment aan het doeleiwit aanmeert. De ontwikkeling van deze stoffenbibliotheken vond plaats als onderdeel van het gezamenlijke Frag4Lead-onderzoeksproject en werd gefinancierd door het Duitse federale ministerie van Onderwijs en Onderzoek (BMBF).
Het MX-team (MX staat voor Macromolecular Crystallography) heeft nu het ontwerp gepubliceerd van een chemisch diverse fragmentbibliotheek genaamd de "F2X-Universal" -bibliotheek, die bestaat uit 1, 103 verbindingen. Een representatieve selectie van 96 verbindingen is uit deze bibliotheek gehaald, dat het F2X-invoerscherm wordt genoemd. Tijdens de publicatie van de bibliotheek, deze selectie is nu met succes getest en gevalideerd door het MX-team van de HZB bij de MAX IV-röntgenbron in Lund, Zweden en bij BESSY II.
In de studie, de HZB- en MAX IV-teams hebben de efficiëntie van de F2X Entry-bibliotheek geverifieerd door endothiapepsine en het Aar2/RnaseH-eiwitcomplex als de doelenzymen te screenen. In de volgende stap, het MX-team zal de volledige universele bibliotheek gebruiken.
"Voor het huidige onderzoek de fragmentscreeningsexperts van HZB—BESSY II werkten nauw samen met het FragMAX-projectteam van MAX IV", zei Dr. Uwe Müller van het MX-team van HZB, die hielp bij het opzetten van de drie MX-bundellijnen bij BESSY II en de BioMAX-bundellijn bij MAX IV. "Hierdoor konden beide partners hun eigen technologieplatforms verder ontwikkelen en gebruiken voor het in beeld brengen van de functionele oppervlakken van verschillende eiwitten. Dit zal een uitstekende basis zijn voor toekomstige samenwerking tussen MAX IV en HZB."
 Nieuwe materialen met een hoge zuurstof-ionengeleiding die een duurzame toekomst opent
Nieuwe materialen met een hoge zuurstof-ionengeleiding die een duurzame toekomst opent Onderzoekers doen nieuwe ontdekking van mechanisme voor ladingopslag
Onderzoekers doen nieuwe ontdekking van mechanisme voor ladingopslag Polymer twin:nieuw implantaat imiteert botstructuur
Polymer twin:nieuw implantaat imiteert botstructuur Magnesiumlegering als lichter alternatief voor aluminiumlegering
Magnesiumlegering als lichter alternatief voor aluminiumlegering Actief leren versnelt het ontdekken van redox-flow-batterijen
Actief leren versnelt het ontdekken van redox-flow-batterijen
 Luchtvervuiling kost $ 2,9 biljoen per jaar:NGO
Luchtvervuiling kost $ 2,9 biljoen per jaar:NGO Verband tussen aardbevingen, regenval en voedselonzekerheid in Nepal
Verband tussen aardbevingen, regenval en voedselonzekerheid in Nepal Hoe zal La Niña de winter in de VS beïnvloeden?
Hoe zal La Niña de winter in de VS beïnvloeden? Simulatie suggereert twee pluimen die betrokken zijn bij de productie van Deccan Traps
Simulatie suggereert twee pluimen die betrokken zijn bij de productie van Deccan Traps Met zuiveraars en rechtszaken, Pakistanen vechten terug tegen smog
Met zuiveraars en rechtszaken, Pakistanen vechten terug tegen smog
Hoofdlijnen
- Wat zijn de drie belangrijkste verschillen tussen een plantencel en een dierencel?
- Twilight-truc:er is een nieuw type cel gevonden in het oog van een diepzeevis
- Tornado's Effecten op mensen
- Wetenschappers brengen nieuwe inzichten in de erfelijkheid van de ernst van de hiv-infectie
- Hoe maak je een celmodel van een Amoeba
- Braziliaanse onderzoekers ontdekken zes nieuwe soorten zijdemiereneter
- Antibioticaresistentie:onderzoekers slagen erin resistentiegenen te blokkeren
- Wetenschappers ontcijferen het genoom van herfstlegerworm, mottenplaag die Afrika binnendringt
- Parasitaire ooginfectie vormt een aanzienlijke bedreiging voor Britse honden, waarschuw experts
- Wetenschappers ontwikkelen Z-schema-katalysator voor verontreinigingen in water
- Ultrasnelle lasers onderzoeken ongrijpbare chemie op het vloeistof-vloeistofgrensvlak

- Tethered chem combo's kunnen een revolutie teweegbrengen in kunstmatige fotosynthese

- Bloedvlekken detecteren - met een antimalariamiddel

- Onderzoek creëert waterstofproducerende levende druppeltjes, weg vrijmaken voor alternatieve toekomstige energiebron
 Elektronische huid opgerekt tot nieuwe grenzen
Elektronische huid opgerekt tot nieuwe grenzen Onderzoekers ontwikkelen een eerste-principes kwantum Monte Carlo-pakket genaamd TurboRVB
Onderzoekers ontwikkelen een eerste-principes kwantum Monte Carlo-pakket genaamd TurboRVB wetenschappers, Zo gebruik je minder plastic
wetenschappers, Zo gebruik je minder plastic Nieuwe lijm plakt gemakkelijk, houdt sterk, en is een gas om uit elkaar te trekken
Nieuwe lijm plakt gemakkelijk, houdt sterk, en is een gas om uit elkaar te trekken Een kleine drempel voor de mens:Kosmische mat verwelkomt buitenaardse wezens
Een kleine drempel voor de mens:Kosmische mat verwelkomt buitenaardse wezens Een 20% -suikeroplossing maken
Een 20% -suikeroplossing maken Groeiende Europese nanodraden
Groeiende Europese nanodraden Tiener klimaatactivist tegen Franse critici:luister naar de wetenschap
Tiener klimaatactivist tegen Franse critici:luister naar de wetenschap
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

