Wetenschap
Wetenschappers identificeren schadelijke bacteriën op basis van hun DNA tegen zeer lage kosten

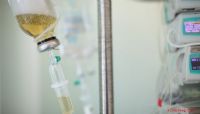
MinION-apparaat (rechts) sequentiëring van bacteriële genomische fragmenten volgens de ON-rep-seq-methode. Hier is het apparaat gekoppeld aan een MinIT voor data-acquisitie (links) en aangestuurd door een smartphone. Krediet:Universiteit van Kopenhagen
Een nieuwe bacteriële identificatiemethode, genaamd ON-rep-seq, onderzoekt selectief, stamspecifieke fragmenten van het bacteriële genoom, het genereren van resultaten die eerder DNA-sequencing van het gehele bacteriële genoom of vervelende benaderingen zoals gepulseerde veldgelelektroforese vereisten, wat voorheen de gouden standaard was voor het typeren van micro-organismen op stamniveau. Vandaar, de methode heeft het potentieel om de benadering te veranderen die wordt gebruikt voor het onderzoeken van op voedsel gebaseerde ziekte-uitbraken door analyse veel minder tijd- en kostenrovend te maken.
Vandaag, bacteriële detectie en identificatie op basis van bacterieel DNA vereist dure instrumentatie en vele uren werk door hoog opgeleide specialisten. Laten we ons voorstellen, bijvoorbeeld, er is een vermoeden Salmonella uitbraak. Meestal om de oorsprong te lokaliseren, niet alleen zullen onderzoekers veel monsters moeten analyseren, maar de analyse moet nauwkeurig zijn om de ene bacteriestam van de andere te kunnen onderscheiden.
"Onze nieuwe methode maakt identificatie en typering van honderden monsters in minder dan twee uur mogelijk, en we verwachten dat dit in korte tijd zelfs zal worden teruggebracht tot "realtime", " zegt een van de onderzoekers achter de studie, Lukasz Krych, Universitair hoofddocent bij de afdeling Voedingswetenschappen van de Universiteit van Kopenhagen, Denemarken.
Methode bouwt voort op een apparaat dat werd gebruikt voor DNA-sequencing in de ruimte
De nieuwe methode is gebaseerd op nanopore sequencing, dat is een nieuwe, real-time DNA-sequencing-aanpak "die zeker een revolutie teweeg zal brengen in de toekomst van DNA-sequencing, " volgens Lukasz Krych.
Het onderzoeksproject werd uitgevoerd in samenwerking met het Poolse bedrijf GenXone S.A., die hielp bij het opzetten van een bioinformatica-pijplijn die nodig is om een snelle en efficiënte analyse van de sequencinggegevens uit te voeren.

Na monstervoorbereiding, het MinION-apparaat is geladen met de genomische fragmenten voor identificatie van bacteriestammen van honderden bacteriële isolaten. Links:Lukasz Krych, Universitair hoofddocent en Josue L. Castro Mejia, postdoc, beide van het Department of Food Science aan de Universiteit van Kopenhagen. Krediet:Afdeling Voedingswetenschappen, Universiteit van Kopenhagen
De kleinste sequencer ooit aangeboden door Oxford Nanopore Technologies, genaamd MinION, is een handheld van $ 999, USB-aangedreven apparaat dat in 2015 in de handel verkrijgbaar werd. Een jaar later werd het naar het internationale ruimtestation ISS gebracht, waar het de eerste DNA-sequencing in de geschiedenis bereikte die werd uitgevoerd in omstandigheden zonder zwaartekracht. Ondanks de onbetwistbare revolutie in DNA-sequencing aangeboden door MinION, het werd al snel duidelijk dat de gegevens die met het apparaat worden gegenereerd nog steeds niet perfect zijn vanwege sequencing-fouten, bijvoorbeeld, terwijl de analyse relatief duur bleef om uit te voeren (ongeveer $ 150 per bacterie).
Klein apparaatje met snelle en goedkope analyse biedt enorme kansen binnen voedselveiligheid
De wetenschappers van het Department of Food Science aan de Universiteit van Kopenhagen hebben een manier gevonden om deze technologie te gebruiken om honderden bacteriën tegelijk te analyseren. de kosten verlagen tot minder dan $ 2 per bacterie, terwijl tegelijkertijd de nauwkeurigheid wordt verhoogd tot meer dan 99%.
"Onze methode is zowel toepasbaar binnen voedselveiligheidsorganisaties, waar je snel ziekteverwekkende of gezondheidsbevorderende bacteriën kunt vinden, en ook in de gezondheidssector, waar u bepaalde analyses kunt uitvoeren die vandaag niet eens worden overwogen vanwege de prijs en het tijdrovende karakter van traditionele analyse, " zegt een andere van de onderzoekers achter de studie, Postdoc Josue Leonardo Castro Mejia.
Momenteel, er zijn verschillende bedrijven die de methode testen om in hun systemen te implementeren voor het opzetten van snelle screeningprogramma's voor duizenden stammen.
 Britse voorspeller geeft eerste onweerswaarschuwing ooit
Britse voorspeller geeft eerste onweerswaarschuwing ooit De wortel van microplastics in planten
De wortel van microplastics in planten Satellietstudie van landbedekking in het Amazone-regenwoud geeft inzicht in branden in 2019
Satellietstudie van landbedekking in het Amazone-regenwoud geeft inzicht in branden in 2019 Welke levende dingen moeten hun voedsel opnemen of absorberen en kunnen geen voedsel intern maken?
Welke levende dingen moeten hun voedsel opnemen of absorberen en kunnen geen voedsel intern maken?  10 manieren om uw boodschappenlijst groener te maken
10 manieren om uw boodschappenlijst groener te maken
Hoofdlijnen
- Kan slapen met een hersenschudding je doden?
- Diepe wortels in planten aangedreven door bodemhydrologie
- Onderzoeker onderzoekt de rol van kleine RNA's in communicatie tussen cellen
- Sonic Kayaks:Milieumonitoring en experimentele muziek door burgers
- Vermindering van individuele plantengroei verhoogt soms de veerkracht van de gemeenschap
- Zwavelademhaling bij zoogdieren
- Hands-on Science-activiteiten over bloed
- De voor- en nadelen van het Bacillus-expressiesysteem
- Onderzoekers schijnen de schijnwerpers op illegale handel in wilde orchideeën
 Midden-infraroodflits gedetecteerd in een nabijgelegen actief sterrenstelsel
Midden-infraroodflits gedetecteerd in een nabijgelegen actief sterrenstelsel voorbij Jupiter, onderzoekers ontdekken een bakermat van kometen
voorbij Jupiter, onderzoekers ontdekken een bakermat van kometen Deutsche Post graaft diep om pakketboom te beheersen
Deutsche Post graaft diep om pakketboom te beheersen Irmas zware regenval gemeten door NASA's IMERG
Irmas zware regenval gemeten door NASA's IMERG Nieuwe screeningsmethode ontwikkeld om te bevestigen of deuteratie de metabole stabiliteit verbetert
Nieuwe screeningsmethode ontwikkeld om te bevestigen of deuteratie de metabole stabiliteit verbetert Dorian kruipt naar de Amerikaanse kust, verzwakking tot categorie 2 storm
Dorian kruipt naar de Amerikaanse kust, verzwakking tot categorie 2 storm Wetenschappers ontdekken diëlektronlading van water nanodruppeltje
Wetenschappers ontdekken diëlektronlading van water nanodruppeltje Leven in een tijd van het gekwantificeerde zelf:hoe apps ons zowel versterken als onderdrukken
Leven in een tijd van het gekwantificeerde zelf:hoe apps ons zowel versterken als onderdrukken
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com