
Wetenschap
Beter begrip van cellulair metabolisme met behulp van AI
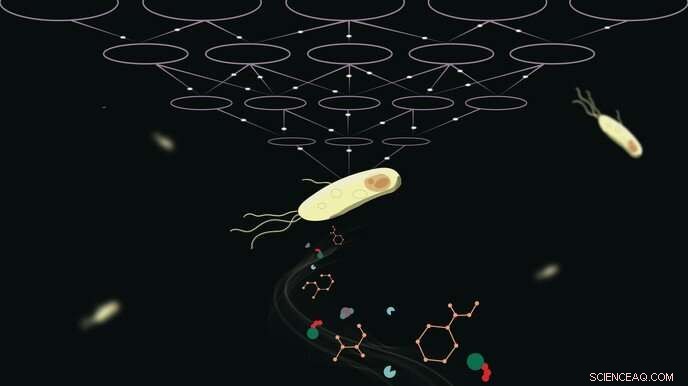
Krediet:Subham Choudhury
Metabolisme is essentieel voor alle levende organismen, en het modelleren van de chemische reacties die het leven in stand houden is geen gemakkelijke taak. Nu hebben EPFL-wetenschappers REKINDLE uitgebracht, een diepgaand leerproces dat de weg vrijmaakt voor efficiëntere en nauwkeurigere modellering van metabolische processen.
De manier waarop een organisme voedingsstoffen metaboliseert, is een complex proces. In de literatuur kan het proces worden gemodelleerd door een reeks wiskundige vergelijkingen met parameters die specifiek zijn voor elk organisme.
De parameters die men voor een mens zou meten, zouden anders zijn dan die van een muis, bacteriën, gist of welk levend organisme dan ook. Als we toegang hadden tot deze parameters voor een bepaald levend organisme, dan zou het bijbehorende model compleet zijn, zou het overeenkomen met real-world observatie, en in silico-studies zouden mogelijk zijn voor een beter ontwerp van in vitro en in vivo studies.
Op praktisch niveau is het bepalen van die parameters echter een gecompliceerde zaak door het ontbreken van experimentele gegevens. Meestal hebben onderzoekers grote hoeveelheden experimentele gegevens en rekenbronnen nodig om deze parameters te bepalen. Maar wat als u de behoefte aan uitgebreide gegevens zou kunnen omzeilen en toch een model kunt produceren dat overeenkomt met experimentele waarneming en meting? EPFL-wetenschappers stellen precies dat voor met REKINDLE, een op diepgaand leren gebaseerd computerraamwerk dat de dynamische metabolische eigenschappen die in cellen worden waargenomen, reproduceert. De resultaten zijn gepubliceerd in Nature Machine Intelligence .
"REKINDLE zal de onderzoeksgemeenschap in staat stellen de computerinspanningen bij het genereren van kinetische modellen met verschillende ordes van grootte te verminderen. Het zal ook helpen bij het postuleren van nieuwe hypothesen door biochemische gegevens in deze modellen te integreren, experimentele observaties op te helderen en nieuwe therapeutische ontdekkingen en biotechnologische ontwerpen te sturen, " zegt Ljubisa Miskovic van EPFL's Laboratory of Computational Systems Biotechnology en co-PI van de studie.
"Het overkoepelende doel van metabole modellering is om het cellulaire metabolische gedrag zodanig te beschrijven dat het begrijpen en voorspellen van de effecten van variaties in cellulaire toestanden en omgevingscondities betrouwbaar kan worden getest voor een breed scala aan onderzoeken in gezondheid, biotechnologie en systemen en synthetische biologie", legt Subham Choudhury, eerste auteur van de studie, uit. "We hopen dat REKINDLE het bouwen van metabole modellen voor de bredere gemeenschap faciliteert."
De methode heeft directe biotechnologische toepassingen, aangezien kinetische modellen belangrijke hulpmiddelen zijn voor verschillende onderzoeken, waaronder bioproductie, targeting van geneesmiddelen, microbioominteracties en bioremediatie.
De EPFL-wetenschappers zijn vooral enthousiast over hoe REKINDLE kan worden gebruikt om het metabolische netwerk van microben te optimaliseren om chemische verbindingen op industriële schaal te produceren, bijvoorbeeld door de traditionele petrochemische industrie te vervangen door celgebaseerde fabrieken.
Een grote uitdaging die het wijdverbreide gebruik van kinetische modellering in de onderzoeksgemeenschap verhindert, zijn de uitgebreide rekenvereisten en het ontbreken van gestandaardiseerde rekensoftware. De EPFL-wetenschappers hopen dat hun op diepgaand leren gebaseerde raamwerk de inspanningen in de wetenschappelijke gemeenschap zal verenigen.
"REKINDLE gebruikt standaard, veelgebruikte Python-bibliotheken die het toegankelijk en gebruiksvriendelijk maken", vervolgt Choudhury. "Ons hoofddoel met deze studie is om de weg vrij te maken om dit soort modelleringsinspanningen open source en toegankelijk te maken, zodat iedereen in de synthetische en systeembiologische gemeenschappen ze kan gebruiken voor hun eigen onderzoeksdoel, wat ze ook mogen zijn." + Verder verkennen
AI als 'wijze raad' voor synthetische biologie
 Nieuwe biochemische route die meer veerkrachtige gewasvariëteiten kan ontwikkelen
Nieuwe biochemische route die meer veerkrachtige gewasvariëteiten kan ontwikkelen Het zwavelgehalte in sommige hoogwaardige stoffen verzwakt ze bij blootstelling aan vocht
Het zwavelgehalte in sommige hoogwaardige stoffen verzwakt ze bij blootstelling aan vocht Polarisatie van broommolecuul in vanadiumoxideclusterholte en nieuwe bromering van alkaan
Polarisatie van broommolecuul in vanadiumoxideclusterholte en nieuwe bromering van alkaan Nanofiltratiemembranen voor de behandeling van industrieel afvalwater van zware metalen
Nanofiltratiemembranen voor de behandeling van industrieel afvalwater van zware metalen Strijd tegen antibioticaresistente bacteriën heeft een gloednieuw wapen
Strijd tegen antibioticaresistente bacteriën heeft een gloednieuw wapen
 Door droogte getroffen Jordanië om ontziltingsinstallatie Rode Zee te bouwen
Door droogte getroffen Jordanië om ontziltingsinstallatie Rode Zee te bouwen Het geheim van het weerstaan van klimaatverandering ligt aan onze voeten
Het geheim van het weerstaan van klimaatverandering ligt aan onze voeten Hoe aerosolen ons klimaat beïnvloeden
Hoe aerosolen ons klimaat beïnvloeden Terugkijkend op de zomer van Amerika vol hitte, overstromingen en klimaatverandering:welkom bij het nieuwe abnormale
Terugkijkend op de zomer van Amerika vol hitte, overstromingen en klimaatverandering:welkom bij het nieuwe abnormale Wetenschappers vinden persistente organische verontreinigende stoffen in dierenbont
Wetenschappers vinden persistente organische verontreinigende stoffen in dierenbont
Hoofdlijnen
- 3 geredde dolfijnen zwemmen vrij uit het Indonesische heiligdom
- Structuur van tarwe-immuun-eiwit opgelost:belangrijk hulpmiddel in de strijd om voedselzekerheid
- 10 verschillende soorten lachen
- Wat is de basis voor uitzonderingen op het Aufbau-principe?
- Roofzuchtige bacteriën die patrijspoorten maken en fresco's schilderen in schadelijke bacteriën
- Wat is het doel van ademhaling?
- Twee muziekbloemlezingen nu voor eeuwig opgeslagen in DNA
- Hoe bacteriën hun motoren turbochargeerden
- Waarom lijden 600 meisjes in Mexico aan collectieve hysterie?
- Stijgende CO2 zorgt ook voor overlast in zoetwater, studie suggereert:

- Een epigenetische sleutel om gedragsverandering te ontgrendelen

- Nieuw machine learning-systeem kan automatisch vormen van rode bloedcellen identificeren
- Artificiële intelligentie gebruiken om tuberculosebehandelingen te verbeteren
- Nieuwe mobiele app diagnosticeert gewasziekten in het veld en waarschuwt boeren op het platteland

 Arctische meren stoten relatief jonge koolstof uit, studie ontdekt
Arctische meren stoten relatief jonge koolstof uit, studie ontdekt Nieuwe studie laat zien wat interstellaire bezoeker Oumuamua ons kan leren
Nieuwe studie laat zien wat interstellaire bezoeker Oumuamua ons kan leren Marskrater herinnert aan Apollo-maanwandeling
Marskrater herinnert aan Apollo-maanwandeling Hoe het gewicht van staal te berekenen
Hoe het gewicht van staal te berekenen Warren zegt dat techreuzen te veel macht hebben, moet uit elkaar gaan
Warren zegt dat techreuzen te veel macht hebben, moet uit elkaar gaan Deezer verkent AI-systeem voor muziek die bij je stemming past
Deezer verkent AI-systeem voor muziek die bij je stemming past Onderzoek toont aan dat bevooroordeelde houdingen – geen economische bezorgdheid – de meeste kiezers naar Trump dreven
Onderzoek toont aan dat bevooroordeelde houdingen – geen economische bezorgdheid – de meeste kiezers naar Trump dreven De verhuizing van Australië om koolstof in de bodem op te slaan is een probleem bij het aanpakken van klimaatverandering
De verhuizing van Australië om koolstof in de bodem op te slaan is een probleem bij het aanpakken van klimaatverandering
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

